Médiathèque

Médiathèque
2455 médias

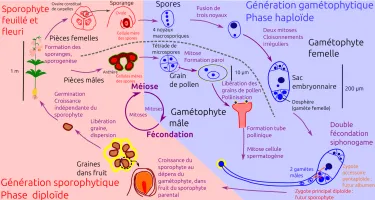
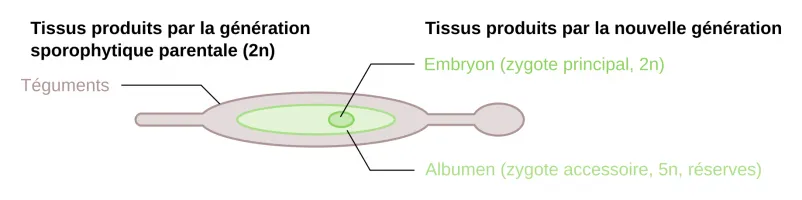
On retrouve globalement les structures observées dans l’ovaire de la fleur, dont le volume a considérablement augmenté. Les carpelles forment une enveloppe dans laquelle se développent six rangées de graines. À maturité, des fentes de déhiscence permettront la libération des graines.
Licence : CC-BY-SA
Fruit de lis martagon
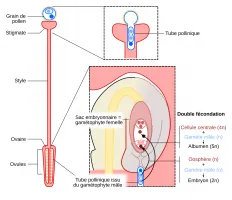
Suite à son dépôt par le vent ou par un pollinisateur sur le stigmate, le grain de pollen forme un tube pollinique dont la croissance permet aux gamètes mâles d’être acheminés depuis le stigmate jusqu’à un ovule en passant par le style. Le tube pollinique arrive finalement au niveau du micropyle d’u...
Licence : CC-BY
Schéma illustrant la double fécondation du gamétophyte femelle par le gamétophyte mâle