Chez l’espèce humaine, la reconnaissance des virus est permise par trois grandes familles de récepteurs de reconnaissance de motifs moléculaires : les TLR, les RLR et le système cGAS-STING. Les recherches menées sur les organismes modèles de laboratoire ainsi que sur des animaux moins couramment étudiés, conjuguées à des approches de génomique comparative, ont permis de montrer que ces trois familles de récepteurs, ainsi que leurs voies de signalisation associées, sont présentes dans la grande majorité du règne animal.
Cet article est une version traduite et adaptée de Majzoub, K., Wrensch, F., and Baumert, T.F. (2019). The Innate Antiviral Response in Animals : An Evolutionary Perspective from Flagellates to Humans. Viruses 11, 758. https://doi.org/10.3390/v11080758
La reconnaissance de motifs moléculaires et l’immunité innée antivirale chez les animaux
Les virus, comme les autres agents pathogènes, expriment des motifs moléculaires associés aux pathogènes, ou PAMP (Pathogen-Associated Molecular Patterns). Ces signatures moléculaires peuvent être reconnues par des récepteurs de reconnaissance de motifs moléculaires, les PRR (Pathogen Recognition-Receptors), qui sont membranaires ou cytosoliques. L’activation des PRR aboutit à la translocation vers le noyau de différents facteurs de transcription. Ceux-ci permettent l’expression de gènes impliqués dans la défense antivirale.
Initialement, une grande partie de notre connaissance de ces voies de signalisation et des récepteurs qui dictent leurs activités est due aux études expérimentales menées sur des organismes modèles tels que la mouche du vinaigre, le nématode C. elegans ou la souris. Plus récemment, grâce aux nouvelles méthodes de séquençage à haut débit et de données génomiques, les scientifiques ont pu découvrir que les gènes constituant ces voies existent également chez plusieurs autres espèces animales. Ainsi, par homologie de séquences, nous savons maintenant que les gènes qui constituent ces voies, tels que ceux codant les récepteurs qui les initient, existent chez des espèces animales dont l’immunité n’a pas encore été assez étudiée expérimentalement comme les Porifères (éponges), les Cnidaires (en particulier les anémones) et les Mollusques. Grâce à ces nouvelles données, nous commençons tout juste à apprécier la conservation de ces voies et leur diversité en fonction des espèces animales. En outre, le développement des expérimentations sur des organismes qui ne sont pas des modèles classiques en recherche promet de nouvelles découvertes sur l’immunité animale et, notamment, humaine.
Notre compréhension des défenses antivirales chez les animaux a été surtout façonnée par des études conduites durant les soixante dernières années chez les Vertébrés et plus particulièrement chez les Mammifères. Il a ainsi été mis en évidence que, chez les Vertébrés, les principales protéines antivirales sécrétées correspondent aux interférons de type I et III (IFN-I et IFN-III) qui agissent de manière autocrine et paracrine en induisant l’expression de centaines de gènes stimulés par les interférons : les ISG (Interferon-Stimulated Genes). Toutefois, durant la dernière décennie, les chercheurs ont pu apprécier que ce « système interféron » est une particularité des Vertébrés. En effet, certaines espèces d’invertébrés comme les Insectes ou les Nématodes sont complètement dépourvues d’interférons et utilisent l’interférence par ARN comme principale voie antivirale. D’autres espèces animales comme les Mollusques, ont conservé toutes les protéines qui conduisent à la production d’interférons sans posséder d’homologues évidents aux interférons de type I. Les interférons semblent donc constituer une innovation évolutive des Vertébrés alors que les voies de signalisation qui dictent leur expression semblent exister tôt durant l’évolution des Métazoaires [1,2].
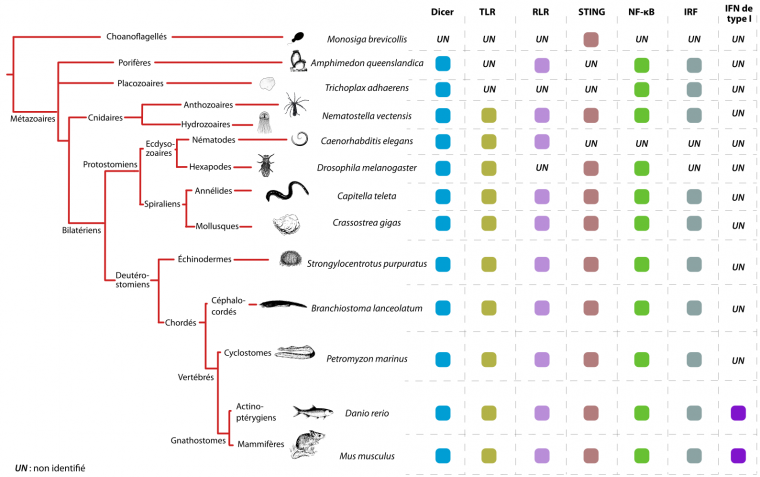
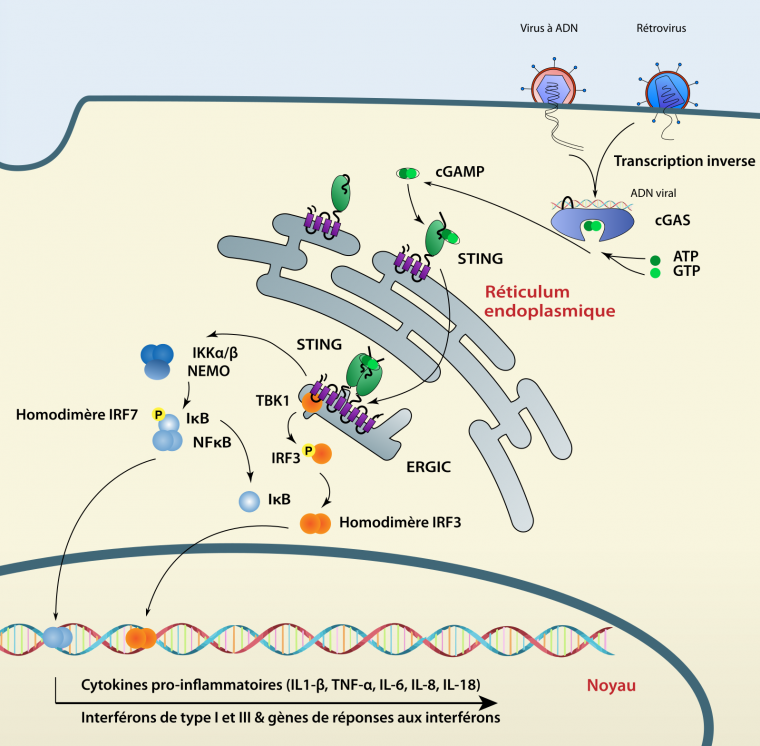
Dans cette mini-revue nous allons nous concentrer sur trois familles de PRR impliquées dans la détection des infections virales chez les animaux. Nous décrirons d’abord le rôle des TLR (Toll-Like Receptors) et des RLR (RIG-I-like Receptors) dans la détection des ARN viraux ainsi que leur conservation à travers les espèces animales. Ensuite, nous nous concentrerons sur un acteur central dans la détection de l’ADN viral, le système cGAS-STING (Stimulator of Interferon Genes), qui semble également être bien conservé chez la plupart des espèces animales (Figure 1).
Les facteurs de l’immunité innée présents chez des espèces représentatives des différents groupes de Métazoaires (animaux) est indiquée par des carrés colorés. UN : non-identifié. Les Choanoflagellés constituent l'extragroupe.
TLR : Toll-like receptors ; RLR : RIG-I-like receptors ; STING : Stimulator of interferon genes ; NF-κB : Nuclear factor κB ; IRF : Interferon regulatory factors ; IFN : interférons. Le rôle de ces différentes molécules ou groupes de molécules est discuté dans la suite de l'article.
Les TLR, une ancienne famille de récepteurs
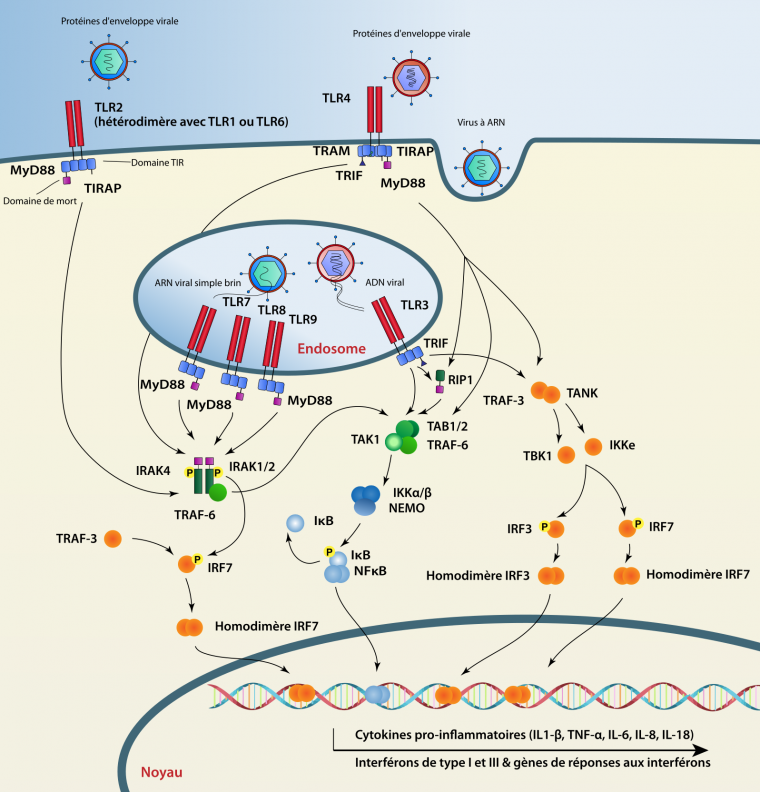
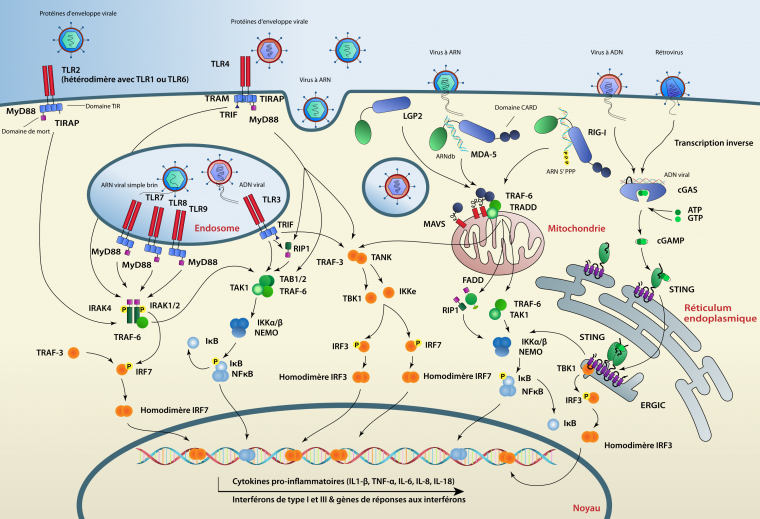
Les TLR sont des protéines transmembranaires composées de trois domaines : un domaine extracellulaire (ou tourné vers la lumière de l’endosome) contenant un nombre variable de répétitions riches en leucine ou LRR (Leucine Rich Repeats), un domaine transmembranaire et un domaine cytoplasmique, le domaine TIR (Toll/interleukin-1 receptor homology domain). Chez l’Homme, dix TLR ont été identifiés mais seuls les TLR3, 7 et 8, présents au niveau des endosomes, seront activés après interaction avec l’ARN viral. Le TLR3 se lie de manière spécifique aux ARN double brin et, après oligomérisation, recrute la protéine adaptatrice TRIF (TIR-domain-containing adapter-Inducing IFN-β). Deux voies de signalisation peuvent ainsi être activées, la voie interféron via le facteur de transcription IRF3 permettant l’expression de l’IFN-β, ou la voie NF-κB via le facteur de transcription NF-κB permettant l’expression de cytokines pro-inflammatoires. Les TLR7 et 8 vont être activés après la reconnaissance d’ARN simple brin et vont recruter la protéine adaptatrice MyD88 (Myeloid Differentiation primary response 88). Ceci va entraîner l’activation des facteurs de transcription IRF5, IRF7 et NF-κB permettant l’expression de l’IFN-α et de cytokines pro-inflammatoires [3].
Le gène Toll a été identifié initialement en 1985, pour son rôle dans le développement embryonnaire de la mouche du vinaigre (Drosophila melanogaster) [4]. Dans les années 90 il a été démontré que ce gène est également impliqué dans l’immunité innée, en jouant un rôle essentiel dans la défense des mouches contre les infections fongiques1 [5]. On sait aujourd’hui que le génome des êtres humains code dix TLR qui peuvent reconnaître de nombreux PAMP bactériens, viraux et eucaryotes. Du point de vue de l’évolution, les TLR appartiennent à une famille de récepteurs très ancienne (voir Figure 1). En effet, deux domaines importants qui constituent les TLR (LRR et TIR) se trouvent dans l’embranchement des Placozoaires (Métazoaires avec une organisation très simple tels que les Trichoplax) et l’embranchement des Porifères (éponges) [6,7]. La structure complète d’un TLR (LRR + TIR) est retrouvée dans l’embranchement des Cnidaires tels que les anémones de mer ou les coraux [8,9]. De plus, chez les Cnidaires, la voie de signalisation induite par les TLR commence par le recrutement de la protéine adaptatrice MyD88 et se termine par l’activation du facteur NF-κB [10].
Dans le sous-règne des Bilatériens, les TLR existent chez un grand nombre d’espèces étudiées mais en nombres variables : d’un seul gène dans l’embranchement des Nématodes, représenté par exemple par Caenorhabditis elegans, à plus de deux cents dans l’embranchement des Échinodermes tel que l’oursin pourpre (Strongylocentrotus purpuratus), montrant de nouveaux mécanismes de détection des pathogènes [11]. La majorité de ces gènes partagent des caractéristiques communes avec ceux du sous-embranchement des Vertébrés et un nombre important semble avoir été dupliqué récemment. Environ 25 à 30 % sont des pseudogènes, des gènes rendus inactifs via des altérations génétiques.
Un autre exemple de la grande diversité des TLR vient des Mollusques, un embranchement des Spiraliens, avec 83 TLR exprimés chez l’huître du Pacifique (Crassotrea gigas). Cela pourrait être entre autres expliqué par une forte pression de sélection due à la diffusion rapide de pathogènes [12]. Par exemple, l’herpesvirus OsHV1 (Ostreid Herpesvirus 1) qui a causé une mortalité de masse chez ces huîtres, est détecté par les TLR induisant l’expression de plus de mille gènes, connus pour être principalement exprimés lors d’infections virales [13].
Contrairement à tous ces embranchements, chez les Chordés et plus particulièrement chez les Vertébrés, un nombre moins important de TLR est exprimé. Cela pourrait être expliqué par l’acquisition de l’immunité adaptative chez ces espèces. En général, chez les Vertébrés, les TLR sont regroupés en six familles majeures dont la famille des TLR3, celle des TLR7 (TRL7, 8 et 9) et celle des TLR11 (TLR11, 12, 13, 19, 20, 21, 22, 23 et 26) qui ont la capacité de reconnaître les PAMP viraux [14]. Tous les Vertébrés ne possèdent pas l’intégralité de ces TLR. Par exemple, TLR22, impliqué dans la reconnaissance des PAMP viraux, ne se retrouve que chez les Ostéichtyens non Tétrapodes (donc en particulier chez les Téléostéens et les Amphibiens, mais pas chez les Oiseaux ni chez les Mammifères), ce qui suggère que ce récepteur est spécifique de virus trouvés dans le milieu aquatique [15]. De manière intéressante chez les chauves-souris (Mammifères), des mutations dans le site de fixation du ligand du TLR ont été retrouvées pour les TLR3, 7, 8 et 9 [16,17]. Certains suggèrent que ces adaptations pourraient expliquer pourquoi ces espèces, réservoirs de nombreux pathogènes, peuvent être infectées d’une manière persistante par plusieurs virus sans pour autant présenter de symptômes particuliers [18].
Évolution des RLR
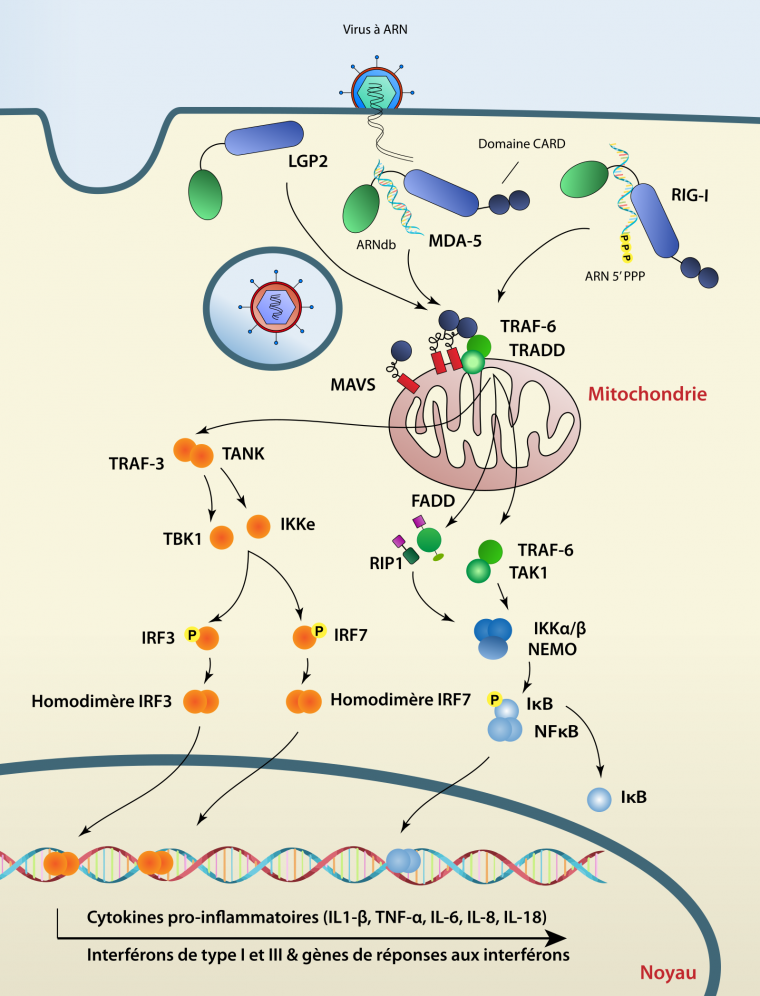
Les RLR sont des protéines cytoplasmiques capables de détecter la présence d’ARN double brin dans le cytoplasme, caractéristique d’une infection par un virus. Dans l’espèce humaine, la famille des RLR est composée de trois membres : RIG-I (retinoic acid-inducible gene I), MDA5 (melanoma differentiation associated gene 5) et LGP2 (laboratory of genetics and physiology 2). Les RLR sont caractérisées par un domaine central de type DEAD-box helicase/ATPase et un domaine de régulation C-terminal (CTD) indispensable pour la reconnaissance des ARN et l’autorépression en absence d’ARN. En plus, RIG-I et MDA5 possèdent un domaine CARD (caspase activation and recruitment domains) en position N-terminale, absent chez LGP2. L’oligomérisation de RIG-I et MDA5 permet le recrutement de la protéine adaptatrice MAVS (mitochondrial antiviral signaling protein) située au niveau de la mitochondrie via une interaction entre les domaines CARD respectifs des deux protéines. Le déclenchement d’une cascade de signalisation entraîne l’activation des facteurs de transcription NF-κB et IRF3 induisant l’expression des gènes antiviraux [3].
RIG-I et MDA5 possèdent deux domaines CARD. Le premier domaine CARD aurait été présent chez le gène ancêtre commun aux gènes RIG-I et MDA5 avant sa duplication et le second domaine CARD aurait été acquis indépendamment après une divergence [19]. Plusieurs études montrent la présence de RLR chez les invertébrés mais ces gènes ont sans doute subi des évolutions spécifiques aux Vertébrés [20] (voir Figure 1). Une étude plus récente suggère que le gène ancestral aux RLR était déjà présent chez l’ancêtre commun des animaux [21]. Les auteurs montrent que les RLR se sont diversifiés fonctionnellement après des duplications et des mutations qui modulent leurs propriétés d’interaction à l’ARN. En effet, les auteurs ont identifié la présence de gènes codant des RLR dans les embranchements des Porifères et des Cnidaires mais pas chez les Eucaryotes non-Métazoaires comme dans la classe des Choanoflagellés ou dans le règne des Champignons [21]. Le gène ancestral des RLR aurait été dupliqué dans le sous-règne des Bilatériens en deux lignées : la lignée RIG-I et la lignée MDA5/LGP2. Au sein des Chordés, la lignée MDA5/LPG2 se serait dupliquée chez Gnathostomes (Vertébrés à mâchoire tel que l’Homme) en deux lignées distinctes, MDA5 et LGP2, car cette duplication est absente dans la super-classe des Cyclostomes. Chez les Mollusques comme C. gigas, on retrouve douze RLR ainsi que MAVS ou des protéines de la famille des IRF (interferon regulatory factors, des facteurs de transcription) [13,22,23]. De plus, il a été montré que cet ensemble de protéines possède une activité antivirale chez cette espèce [23].
L’étude des RLR chez les Bilatériens montre une évolution très dynamique de ces gènes illustrée par la perte de l’une ou l’autre lignée au sein de certains taxons. En effet, des homologues de MDA5 et LGP2 ont été identifiés chez un grand nombre de Téléostéens (« poissons » dont la nageoire caudale présente une apparente symétrie) alors que des homologues de RIG-I n’ont été retrouvés que chez quelques-unes de ces espèces comme le saumon et la carpe [24]. Une étude chez les Oiseaux a révélé l’absence d’homologue de RIG-I chez le poulet mais la présence d’homologues de MDA5 et LGP2, alors que le canard colvert possède les deux lignées (RIG-I et MDA5/LGP2). De manière intéressante, le poulet est très susceptible au virus de la grippe alors que le canard colvert ne l’est pas, suggérant un rôle potentiel de RIG-I contre ce virus dans les cellules épithéliales [25,26]. Chez les Mammifères, la seule espèce connue pour avoir perdu RIG-I est le toupaye de Belanger (Tupaia belangerie). Avec la perte de RIG-I dans cette espèce, MDA5 et LGP2 ont subi une forte pression de sélection, particulièrement dans des sites de MDA5 qui semblent compenser l’absence de RIG-I [27,28].
Un autre exemple éloquent de l’évolution dynamique des RLR est l’absence de la voie RLR/MAVS/IRF chez les Insectes (voir Figure 1). Il est important de noter que ces derniers utilisent l’interférence par ARN (ARNi) comme mécanisme majeur de défense antivirale [29], [30]. Une des protéines clés dans ce système est Dicer-2, une RNAse de type III qui clive l’ARN viral double brin en petits ARN interférents (small interfering RNA – siRNA). Ces petits ARN s’hybrident ensuite avec l’ARN viral simple brin, entraînant son clivage. De manière surprenante, Dicer-2 possède en N-terminal une DEAD/H-box avec une forte homologie à celle retrouvée chez les RLR [31,32]. Une dernière observation qui illustre l’évolution dynamique et rapide des récepteurs RLR vient des Mammifères où RIG-I semble avoir accumulé des mutations adaptatives qui modifient ses propriétés de liaison à l’ARN [33]. En outre, chez l’espèce humaine, un grand nombre de polymorphismes a été identifié dans RIG-I ce qui semble contribuer à une variabilité dans la susceptibilité aux infections virales et dans le risque des maladies auto-immunes [34,35].
Évolution de cGAS-STING
La reconnaissance d’ADN viral dans le cytosol est permise par cGAS. Lorsqu’elle se lie à l’ADN, cette enzyme synthétise, à partir d’ATP et de GTP, le 2’3’-GMP-AMP cyclique (cGAMP). Ce second messager active alors STING, une protéine adaptatrice qui, comme MAVS et TRIF, est fixée au réticulum endoplasmique grâce à des domaines transmembranaires. Le changement de conformation de STING, induit par son interaction avec le 2’3’-cGAMP, libère son domaine C-terminal (CTT, C-terminal tail) dans le cytoplasme. Pour activer IRF3 et la réponse interféron, STING forme un homodimère au niveau périnucléaire. Ceci permet sa phosphorylation par la kinase TBK1, servant ainsi de point d’ancrage à IRF3 [3]. STING est indispensable à la réponse antivirale contre les virus à ADN tels que le virus de l’herpès simplex I (HSV-I) [36]. De plus, STING a la capacité d’interagir, avec une plus faible affinité, avec d’autres dinucléotides cycliques tels que le 2’2’-cGAMP, le 3’2’-cGAMP ou les dinucléotides cycliques bactériens comme ceux produits par Listeria monocytogenes [37–39].
La voie cGAS-STING semble être plus ancienne que celle des RLR car STING est retrouvée dans les génomes d’un grand nombre d’espèces du règne des Métazoaires et dans ceux d’une classe d’Eucaryotes unicellulaires : les Choanoflagellés [40,41] (voir Figure 1). Un homologue de STING a été identifié chez l’anémone étoilée (Nematostella vectensis : nvSTING) faisant partie de l’embranchement des Cnidaires [42]. La structure de nvSTING est similaire à son homologue humain et cette protéine interagit avec le cGAMP avec une forte affinité. Cependant, le domaine CTT phosphorylé par TBK1 n’est retrouvé que chez les Vertébrés, nvSTING n’a donc pas la capacité de déclencher la réponse interféron [42,43]. Cependant, STING peut tout de même avoir un rôle antiviral chez les invertébrés. En effet, dans le super-embranchement des Spiraliens, l’huître du Pacifique (C. gigas) et l’annélide Capitella teleta expriment une version de STING fusionnée à un domaine TIR impliqué dans les voies de signalisation de l’immunité innée [43]. De plus, dans l’embranchement des Arthropodes comme chez la mouche drosophile où le système interféron est absent, STING (sans CTT) est important dans la réponse antibactérienne et antivirale via l’activation de NF-κB [44]. Néanmoins, chez les Vertébrés, la capacité d’induire une réponse interféron semble être très variable. Cette plasticité est illustrée par un recrutement variable des facteurs de transcription et donc d’activation de la réponse interféron [45]. Chez les Mammifères, la signalisation via STING aboutit à une forte activation de la réponse interféron et un faible recrutement de NF-κB. Cependant, dans la classe des Actinoptérygiens, une extension du domaine CTT entraîne une forte activation de NF-κB et une faible activation de la voie interféron via IRF3 [45]. Un autre exemple de la plasticité de STING provient d’une étude sur des chauves-souris où une mutation de la sérine 358 de cette protéine diminue fortement la réponse interféron [46]. Cette évolution éviterait une forte réponse interféron pour un animal qui peut vivre avec des charges virales très importantes [46].
Le rôle de cGAS comme protéine ayant une activité antivirale est plus complexe. Même si cette protéine est apparue tôt dans l'histoire des Métazoaires, sa capacité à détecter l’ADN double brin ne serait apparue que chez les Vertébrés [40]. Cette hypothèse repose sur le fait que le domaine de cGAS permettant l’interaction avec l’ADN n’est présent que chez les Vertébrés [40]. Clairement, cGAS et STING semblent acquérir des nouvelles caractéristiques durant l’évolution. Pour prendre l’exemple des Vertébrés, la protéine cGAS a acquis un domaine de liaison à l’ADN tandis que l’évolution du domaine CTT de STING a conditionné la voie de signalisation majoritairement activée par cette protéine chez différents taxons.
Conclusion
L’étude comparée des récepteurs et voies de signalisation impliqués dans la réponse antivirale chez les Métazoaires montre que la plupart de ces éléments sont largement partagés au sein de ce règne et qu’ils ont donc été mis en place tôt dans l’évolution de ce taxon. Cependant, si les gènes codant les PRR et les protéines des voies de signalisation associées se dupliquent dans certaines lignées, ils sont perdus dans d’autres. Ces évolutions contrastées proviennent probablement en partie de pressions de sélection différentes, reflétant la grande diversité des micro-organismes auxquels sont exposés les différents groupes de Métazoaires, notamment en fonction de leurs milieux de vie. L’évolution de la réponse immunitaire aux virus est également nécessairement en partie contingente. Toujours est-il que les variations observées dans les récepteurs et mécanismes de signalisation des animaux actuels sont probablement en partie responsables de la susceptibilité variable aux pathogènes des différentes espèces animales.
L’étude de l’évolution de la réponse immunitaire contre les virus montre l’étroite relation entre un hôte et ses pathogènes. Le système immunitaire innée des animaux actuels est généralement peu optimisé face aux nouveaux virus, car il a été sélectionné contre des virus anciens grâce à une forte coévolution. Cette évolution permanente nous ramène à l’hypothèse de la reine rouge émise par Leigh Van Valen [47] et qui dit : « l’évolution permanente d’une espèce est nécessaire pour maintenir son aptitude face aux évolutions des espèces avec lesquelles elle coévolue ».
Références
- C. J. Secombes and J. Zou, ‘Evolution of Interferons and Interferon Receptors’, Front Immunol, vol. 8, p. 209, 2017, doi : 10.3389/fimmu.2017.00209.
- J. Nehyba, R. Hrdlickova, and H. R. Bose, ‘Dynamic evolution of immune system regulators : the history of the interferon regulatory factor family’, Mol Biol Evol, vol. 26, no. 11, pp. 2539–50, Nov. 2009, doi : 10.1093/molbev/msp167.
- K. Majzoub, F. Wrensch, and T. F. Baumert, ‘The Innate Antiviral Response in Animals : An Evolutionary Perspective from Flagellates to Humans’, Viruses, vol. 11, no. 8, Aug. 2019, doi : 10.3390/v11080758.
- K. V. Anderson, L. Bokla, and C. Nusslein-Volhard, ‘Establishment of dorsal-ventral polarity in the Drosophila embryo : the induction of polarity by the Toll gene product’, Cell, vol. 42, no. 3, pp. 791–8, Oct. 1985.
- B. Lemaitre, E. Nicolas, L. Michaut, J. M. Reichhart, and J. A. Hoffmann, ‘The dorsoventral regulatory gene cassette spatzle/Toll/cactus controls the potent antifungal response in Drosophila adults’, Cell, vol. 86, no. 6, pp. 973–83, Sep. 1996.
- J. J. Brennan and T. D. Gilmore, ‘Evolutionary Origins of Toll-like Receptor Signaling’, Mol Biol Evol, vol. 35, no. 7, pp. 1576–1587, Jul. 2018, doi : 10.1093/molbev/msy050.
- K. Kamm, B. Schierwater, and R. DeSalle, ‘Innate immunity in the simplest animals – placozoans’, BMC Genomics, vol. 20, no. 1, p. 5, Jan. 2019, doi : 10.1186/s12864-018-5377-3.
- J. J. Brennan, J. L. Messerschmidt, L. M. Williams, B. J. Matthews, M. Reynoso, and T. D. Gilmore, ‘Sea anemone model has a single Toll-like receptor that can function in pathogen detection, NF-kappaB signal transduction, and development’, Proc Natl Acad Sci U A, vol. 114, no. 47, pp. E10122–E10131, Nov. 2017, doi : 10.1073/pnas.1711530114.
- D. J. Miller et al., ‘The innate immune repertoire in cnidaria--ancestral complexity and stochastic gene loss’, Genome Biol, vol. 8, no. 4, p. R59, 2007, doi : 10.1186/gb-2007-8-4-r59.
- L. M. Williams et al., ‘A conserved Toll-like receptor-to-NF-kappaB signaling pathway in the endangered coral Orbicella faveolata’, Dev Comp Immunol, vol. 79, pp. 128–136, Feb. 2018, doi : 10.1016/j.dci.2017.10.016.
- J. P. Rast, L. C. Smith, M. Loza-Coll, T. Hibino, and G. W. Litman, ‘Genomic insights into the immune system of the sea urchin’, Science, vol. 314, no. 5801, pp. 952–6, Nov. 2006, doi : 10.1126/science.1134301.
- E. E. Philipp et al., ‘Massively parallel RNA sequencing identifies a complex immune gene repertoire in the lophotrochozoan Mytilus edulis’, PLoS One, vol. 7, no. 3, p. e33091, 2012, doi : 10.1371/journal.pone.0033091.
- L. Zhang, L. Li, X. Guo, G. W. Litman, L. J. Dishaw, and G. Zhang, ‘Massive expansion and functional divergence of innate immune genes in a protostome’, Sci Rep, vol. 5, p. 8693, Mar. 2015, doi : 10.1038/srep08693.
- J. C. Roach et al., ‘The evolution of vertebrate Toll-like receptors’, Proc Natl Acad Sci U A, vol. 102, no. 27, pp. 9577–82, Jul. 2005, doi : 10.1073/pnas.0502272102.
- A. Matsuo et al., ‘Teleost TLR22 recognizes RNA duplex to induce IFN and protect cells from birnaviruses’, J Immunol, vol. 181, no. 5, pp. 3474–85, Sep. 2008.
- J. Schad and C. C. Voigt, ‘Adaptive evolution of virus-sensing toll-like receptor 8 in bats’, Immunogenetics, vol. 68, no. 10, pp. 783–795, Nov. 2016, doi : 10.1007/s00251-016-0940-z.
- M. Escalera-Zamudio et al., ‘The evolution of bat nucleic acid-sensing Toll-like receptors’, Mol Ecol, vol. 24, no. 23, pp. 5899–909, Dec. 2015, doi : 10.1111/mec.13431.
- S. Subudhi, N. Rapin, and V. Misra, ‘Immune System Modulation and Viral Persistence in Bats : Understanding Viral Spillover’, Viruses, vol. 11, no. 2, Feb. 2019, doi : 10.3390/v11020192.
- D. Sarkar, R. Desalle, and P. B. Fisher, ‘Evolution of MDA-5/RIG-I-dependent innate immunity : independent evolution by domain grafting’, Proc Natl Acad Sci U A, vol. 105, no. 44, pp. 17040–5, Nov. 2008, doi : 10.1073/pnas.0804956105.
- J. Zou, M. Chang, P. Nie, and C. J. Secombes, ‘Origin and evolution of the RIG-I like RNA helicase gene family’, BMC Evol Biol, vol. 9, p. 85, Apr. 2009, doi : 10.1186/1471-2148-9-85.
- K. Mukherjee, B. Korithoski, and B. Kolaczkowski, ‘Ancient origins of vertebrate-specific innate antiviral immunity’, Mol Biol Evol, vol. 31, no. 1, pp. 140–53, Jan. 2014, doi : 10.1093/molbev/mst184.
- T. J. Green and P. Speck, ‘Antiviral Defense and Innate Immune Memory in the Oyster’, Viruses, vol. 10, no. 3, Mar. 2018, doi : 10.3390/v10030133.
- B. Huang, L. Zhang, Y. Du, F. Xu, L. Li, and G. Zhang, ‘Characterization of the Mollusc RIG-I/MAVS Pathway Reveals an Archaic Antiviral Signalling Framework in Invertebrates’, Sci Rep, vol. 7, no. 1, p. 8217, Aug. 2017, doi : 10.1038/s41598-017-08566-x.
- S. N. Chen, P. F. Zou, and P. Nie, ‘Retinoic acid-inducible gene I (RIG-I)-like receptors (RLRs) in fish : current knowledge and future perspectives’, Immunology, vol. 151, no. 1, pp. 16–25, May 2017, doi : 10.1111/imm.12714.
- M. Liniger, A. Summerfield, G. Zimmer, K. C. McCullough, and N. Ruggli, ‘Chicken cells sense influenza A virus infection through MDA5 and CARDIF signaling involving LGP2’, J Virol, vol. 86, no. 2, pp. 705–17, Jan. 2012, doi : 10.1128/JVI.00742-11.
- M. R. Barber, J. R. Aldridge, R. G. Webster, and K. E. Magor, ‘Association of RIG-I with innate immunity of ducks to influenza’, Proc Natl Acad Sci U A, vol. 107, no. 13, pp. 5913–8, Mar. 2010, doi : 10.1073/pnas.1001755107.
- Y. Fan et al., ‘Genome of the Chinese tree shrew’, Nat Commun, vol. 4, p. 1426, 2013, doi : 10.1038/ncomms2416.
- L. Xu, D. Yu, Y. Fan, L. Peng, Y. Wu, and Y. G. Yao, ‘Loss of RIG-I leads to a functional replacement with MDA5 in the Chinese tree shrew’, Proc Natl Acad Sci U A, vol. 113, no. 39, pp. 10950–5, Sep. 2016, doi : 10.1073/pnas.1604939113.
- D. Galiana-Arnoux, C. Dostert, A. Schneemann, J. A. Hoffmann, and J.-L. Imler, ‘Essential function in vivo for Dicer-2 in host defense against RNA viruses in drosophila’, Nat. Immunol., vol. 7, no. 6, pp. 590–597, Jun. 2006, doi : 10.1038/ni1335.
- R. P. van Rij et al., ‘The RNA silencing endonuclease Argonaute 2 mediates specific antiviral immunity in Drosophila melanogaster’, Genes Dev, vol. 20, no. 21, pp. 2985–95, Nov. 2006, doi : 10.1101/gad.1482006.
- S. Deddouche et al., ‘The DExD/H-box helicase Dicer-2 mediates the induction of antiviral activity in drosophila’, Nat Immunol, vol. 9, no. 12, pp. 1425–32, Dec. 2008, doi : 10.1038/ni.1664.
- S. Paro, J. L. Imler, and C. Meignin, ‘Sensing viral RNAs by Dicer/RIG-I like ATPases across species’, Curr Opin Immunol, vol. 32, pp. 106–13, Feb. 2015, doi : 10.1016/j.coi.2015.01.009.
- D. C. Rawling, A. S. Kohlway, D. Luo, S. C. Ding, and A. M. Pyle, ‘The RIG-I ATPase core has evolved a functional requirement for allosteric stabilization by the Pincer domain’, Nucleic Acids Res, vol. 42, no. 18, pp. 11601–11, Oct. 2014, doi : 10.1093/nar/gku817.
- E. Vasseur et al., ‘The selective footprints of viral pressures at the human RIG-I-like receptor family’, Hum Mol Genet, vol. 20, no. 22, pp. 4462–74, Nov. 2011, doi : 10.1093/hmg/ddr377.
- J. A. Tennessen et al., ‘Evolution and functional impact of rare coding variation from deep sequencing of human exomes’, Science, vol. 337, no. 6090, pp. 64–9, Jul. 2012, doi : 10.1126/science.1219240.
- H. Ishikawa and G. N. Barber, ‘STING is an endoplasmic reticulum adaptor that facilitates innate immune signalling’, Nature, vol. 455, no. 7213, pp. 674–8, Oct. 2008, doi : 10.1038/nature07317.
- D. L. Burdette et al., ‘STING is a direct innate immune sensor of cyclic di-GMP’, Nature, vol. 478, no. 7370, pp. 515–8, Sep. 2011, doi : 10.1038/nature10429.
- B. W. Davies, R. W. Bogard, T. S. Young, and J. J. Mekalanos, ‘Coordinated regulation of accessory genetic elements produces cyclic di-nucleotides for V. cholerae virulence’, Cell, vol. 149, no. 2, pp. 358–70, Apr. 2012, doi : 10.1016/j.cell.2012.01.053.
- L. Jin et al., ‘MPYS is required for IFN response factor 3 activation and type I IFN production in the response of cultured phagocytes to bacterial second messengers cyclic-di-AMP and cyclic-di-GMP’, J Immunol, vol. 187, no. 5, pp. 2595–601, Sep. 2011, doi : 10.4049/jimmunol.1100088.
- X. Wu et al., ‘Molecular evolutionary and structural analysis of the cytosolic DNA sensor cGAS and STING’, Nucleic Acids Res, vol. 42, no. 13, pp. 8243–57, Jul. 2014, doi : 10.1093/nar/gku569.
- P. Schaap, ‘Cyclic di-nucleotide signaling enters the eukaryote domain’, IUBMB Life, vol. 65, no. 11, pp. 897–903, Nov. 2013, doi : 10.1002/iub.1212.
- P. J. Kranzusch, S. C. Wilson, A. S. Lee, J. M. Berger, J. A. Doudna, and R. E. Vance, ‘Ancient Origin of cGAS-STING Reveals Mechanism of Universal 2’,3’ cGAMP Signaling’, Mol Cell, vol. 59, no. 6, pp. 891–903, Sep. 2015, doi : 10.1016/j.molcel.2015.07.022.
- S. R. Margolis, S. C. Wilson, and R. E. Vance, ‘Evolutionary Origins of cGAS-STING Signaling’, Trends Immunol, vol. 38, no. 10, pp. 733–743, Oct. 2017, doi : 10.1016/j.it.2017.03.004.
- A. Goto et al., ‘The Kinase IKKbeta Regulates a STING – and NF-kappaB-Dependent Antiviral Response Pathway in Drosophila’, Immunity, vol. 49, no. 2, pp. 225-234 e4, Aug. 2018, doi : 10.1016/j.immuni.2018.07.013.
- C. C. de Oliveira Mann, M. H. Orzalli, D. S. King, J. C. Kagan, A. S. Y. Lee, and P. J. Kranzusch, ‘Modular Architecture of the STING C-Terminal Tail Allows Interferon and NF-kappaB Signaling Adaptation’, Cell Rep, vol. 27, no. 4, pp. 1165-1175 e5, Apr. 2019, doi : 10.1016/j.celrep.2019.03.098.
- J. Xie et al., ‘Dampened STING-Dependent Interferon Activation in Bats’, Cell Host Microbe, vol. 23, no. 3, pp. 297-301 e4, Mar. 2018, doi : 10.1016/j.chom.2018.01.006.
- L. H. Liow, L. Van Valen, and N. Chr. Stenseth, ‘Red Queen : from populations to taxa and communities’, Trends Ecol. Evol., vol. 26, no. 7, pp. 349–358, Jul. 2011, doi : 10.1016/j.tree.2011.03.016.