La famille des protéines IFITM (interferon-induced transmembrane, IFITM dans la présente revue) regroupe plusieurs protéines, parmi lesquelles les IFITM 1, 2 et 3 qui sont des facteurs de restriction antivirale induits par les interférons (IFN). Les IFITM inhibent la fusion membranaire d’un grand nombre de virus enveloppés chez les vertébrés. En nous appuyant sur les données les plus récentes de la littérature scientifique, nous étudions dans cette revue la structure et la localisation subcellulaire des IFITM, les mécanismes par lesquels elles inhibent la fusion virale, la résistance de certains virus vis-à-vis de ces protéines et leur importance in vivo dans un contexte infectieux.
Les virus enveloppés disposent d’une membrane. Lors de la première étape de leur cycle infectieux, ils entrent dans leurs cellules cibles suite à la fusion de leur membrane avec une membrane cellulaire. Il peut s’agir de la membrane plasmique, ou de la membrane d’un endosome ou d’un lysosome si le virus est endocyté par sa cellule cible. La fusion membranaire est réalisée par une protéine virale nommée glycoprotéine de fusion. Une fois entré dans la cellule, le virus réplique son génome et assemble de nouvelles particules virales qui quittent la cellule par bourgeonnement.
Lorsqu’une cellule est infectée par un virus, ou que le système immunitaire détecte une infection virale, l’organisme produit des cytokines telles que les interférons (IFN) qui indiquent aux cellules encore saines le danger d’infection. Les cellules réceptrices de ces signaux commencent alors à exprimer des facteurs de restriction, c’est-à-dire des protéines qui inhibent certaines étapes du cycle infectieux d’un ou plusieurs virus. Les facteurs de restriction figurent parmi les principaux acteurs de l’immunité innée, un ensemble de réponses rapides mais non spécifiques aux infections.
En 2009, une étude visant à identifier les facteurs de restriction impliqués dans la défense contre le virus de l’influenza A (IAV, le principal pathogène responsable de la grippe) a détecté qu’une protéine encore peu connue, IFITM3 (interferon-induced transmembrane 3), restreint une étape précoce du cycle viral dans plusieurs types de cellules (Brass et coll., 2009). Il a ensuite été démontré que l’étape en question est la fusion membranaire virale, et que les IFITM 1, 2 et 3, que l’on retrouve chez tous les vertébrés (Bailey et coll., 2014), interfèrent avec le cycle infectieux d’un grand nombre de virus enveloppés (Li et coll., 2013).
Localisation et structure des protéines IFITM
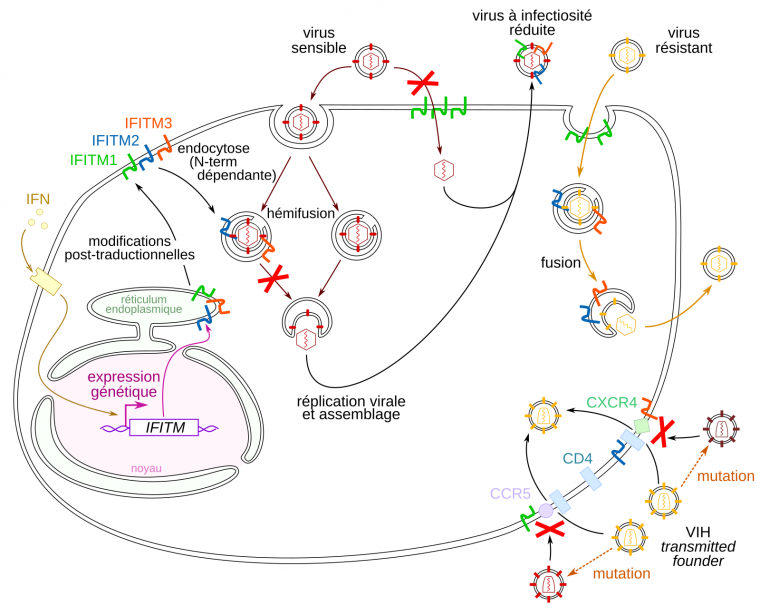
Les IFITM sont des protéines transmembranaires. Les IFITM 2 et 3 se trouvent majoritairement dans les membranes endosomales et lysosomales, tandis qu’IFITM1 se situe principalement à la membrane plasmique. Les IFITM sont synthétisées à la membrane du réticulum endoplasmique, puis leur oligomérisation et des modifications post-traductionnelles induisent leur adressage à la membrane plasmique (John et coll., 2013 ; Yount et coll., 2012 ; Chesarino et coll., 2014). Les IFITM 2 et 3 sont ensuite adressées aux endosomes. En effet, IFITM3 possède un motif 20-YXXF-231 ; la phosphorylation de la tyrosine 20 permet la reconnaissance du motif par AP-2, une protéine adaptatrice impliquée dans l’endocytose dépendante des clathrines (Foster et coll., 2016). Le segment N-terminal d’IFITM1 est plus court que celui des IFITM 2 et 3 d’environ 20 acides aminés et ne dispose pas du motif d’adressage aux endosomes.
Les IFITM peuvent subir de nombreuses maturations post-traductionnelles (Tableau 1), qui peuvent moduler leur oligomérisation et leur activité en participant à l’effet antiviral ou au contraire en facilitant l’entrée de certains virus (Zhao et coll., 2018 ; John et coll., 2013 ; Shan et coll., 2013). Ainsi, l’oligomérisation d’IFITM3 est nécessaire à la restriction de certains virus, mais d’autres, comme NL63-CoV, peuvent être restreints par des IFITM3 monomériques (Zhao et coll., 2018).
| Maturations post-traductionnelles | Résidus impliqués chez IFITM3 |
|---|---|
| S-palmitoylation | Cys71, Cys72, Cys105 |
| Phosphorylation | Tyr20, Tyr27 |
| Ubiquitinylation | Lys24, Lys83, Lys88 et Lys104 |
| Méthylation | Lys88 |
Les IFITM disposent d’un segment N-terminal intracytosolique, d’une hélice amphipathique insérée dans le feuillet cytosolique de leur membrane de résidence, d’un segment transmembranaire et d’un segment C-terminal luminal ou extracellulaire (Bailey et coll., 2013) ; il est cependant envisageable que les IFITM adoptent une topologie dynamique avec plusieurs conformations possibles.
Mécanismes de la restriction virale par les IFITM
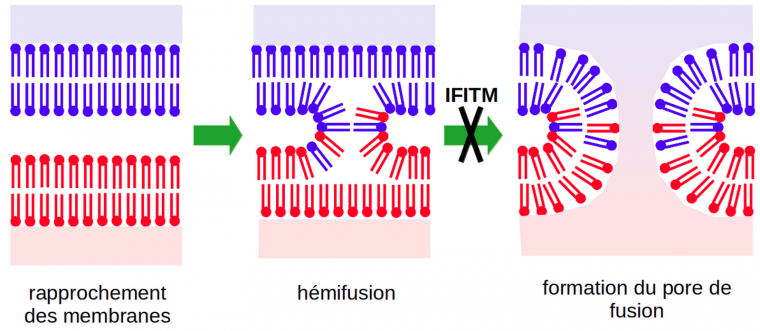
La restriction de l’infection par les IFITM s’explique probablement par une diminution de la fluidité des membranes dans lesquelles les IFITM sont insérées (Lin et coll., 2013) et peut-être par une modification de leur composition lipidique (Amini-Bavil-Olyaee et coll., 2014). Ces mécanismes expliqueraient que la fusion de l’enveloppe virale avec la membrane modifiée par les IFITM devienne moins favorable et réussisse donc plus rarement.
IFITM3 inhibe la fusion virale après l’étape d’hémifusion (Figure 1) lorsqu’elle se trouve dans la membrane de l’endosome qui contient le virion (Suddala et coll., 2019). De plus, il semble qu’IFITM3 accélère l’envoi des virions aux lysosomes (Spence et coll., 2019), ce qui faciliterait leur dégradation.
D’après les résultats de Desai et coll., 2014.
Dans le cas du virus de l’immunodéficience humaine (VIH), les IFITM coalescent avec les protéines GAG de la particule virale et sont incorporées dans son enveloppe lors du bourgeonnement ; il en résulte une production de particules virales d’infectivité moins élevée car celles-ci fusionnent plus difficilement à la membrane plasmique lorsqu’elles entament un nouveau cycle infectieux (Compton et coll., 2013 ; Tartour et coll., 2014). Les IFITM restreignent des virus de plusieurs autres familles virales par le même processus. Ainsi, les IFITM ont une activité bimodale : elles restreignent la fusion virale non seulement au début du cycle infectieux, quand elles se trouvent dans les membranes cellulaires, mais aussi indirectement à la fin du cycle infectieux, quand leur incorporation dans les nouvelles particules virales rend ces particules moins infectieuses.
Impact des IFITM dans l’immunité antivirale à l’échelle de l’organisme
Les effets de la restriction des infections virales par les IFITM ne s’observent pas uniquement à l’échelle cellulaire, mais sont visibles à l’échelle de l’organisme. En effet, des souris Ifitm3−/−,chez qui IFITM3 est absente, présentent une susceptibilité accrue à l’infection par le virus de l’influenza A (IAV) (Everitt et coll., 2012), le virus du Nil occidental (WNV) (Gorman et coll., 2016), le virus du Chikungunya (CHIKV), le virus de l’encéphalite équine vénézuélienne (VEEV) (Poddar et coll., 2016), le virus respiratoire syncytial (RSV) (Everitt et coll., 2013) et le cytomégalovirus (CMV) (Stacey et coll., 2017). Cette susceptibilité se traduit par une forte perte de poids et une mortalité augmentée chez les souris Ifitm3−/− infectées.
Chez l’être humain, deux polymorphismes mononucléotidiques (ou SNP, single nucleotide polymorphism) ont été identifiés dans le gène IFITM3 et sont associés à des infections virales plus sévères par le virus de l’influenza A. L’allèle rs34481144-A, chez qui une mutation dans la région 5’ non codante du gène IFITM3 induit une diminution de l’expression de la protéine, est associé à une grippe plus sévère dans la population afro-américaine, et à un taux de décès plus élevé à la suite de l’infection dans la population européenne (Allen et coll., 2017). L’allèle rs12252-C (issu d’une délétion de 21 résidus dans le domaine N-terminal d’IFITM3) induit à l’état homozygote une augmentation de la mortalité et morbidité induites par le virus influenza A (Yang et coll., 2015) et par le VIH (Zhang et coll., 2015) dans la population chinoise, mais aucune conclusion ne peut être tirée quant aux effets de ce polymorphisme sur la population européenne à cause de sa rareté (Everitt et coll., 2012).
Certains virus résistent aux IFITM
Plusieurs virus sont résistants aux IFITM tels que le virus de l’hépatite C. Généralement, les virus résistants à une IFITM résistent aussi aux deux autres, même s’il existe des exceptions : par exemple, le virus Dugbe (DUGV) est sensible aux IFITM 1 et 2 mais pas à IFITM3, et le virus Eppstein Barr (EBV) est sensible aux IFITM 2 et 3 mais pas à IFITM1 (Tartour et coll., 2017).
Le virus de Lassa résiste à IFITM3 en étant endocyté majoritairement dans des endosomes ne contenant pas IFITM3 (Suddala et coll., 2019). Les mécanismes par lesquels ce virus évite les endosomes qui contiennent IFITM3 restent encore inconnus. Chez d’autres virus, la résistance semble provenir d’un défaut d’incorporation des IFITM dans l’enveloppe virale lors du bourgeonnement (Tartour et coll., 2017). La résistance des virus aux IFITM n’est généralement pas liée à leur glycoprotéine de fusion : en effet, des cellules exprimant à leur membrane plasmique la protéine de fusion d’un virus résistant aux IFITM sont incapables de fusionner avec des cellules exprimant des IFITM (Suddala et coll., 2019 ; Li et coll., 2013), ce qui montre que les IFITM sont bel et bien capables d’inhiber l’action de la glycoprotéine quand les autres composants du virus sont absents. Toutefois, pour le VIH, la glycoprotéine de fusion détermine indirectement la sensibilité du virus aux IFITM (Foster et coll., 2016 ; Wang et coll., 2017). En effet, il a été observé que lors d’une infection de novo, la souche infectieuse (nommée transmitted founder) résiste généralement à la restriction par les IFITM (Foster et coll., 2016). Cependant, la résistance est en grande partie perdue environ six mois après la primo-infection, sous l’effet de mutations de la glycoprotéine de fusion, qui empêchent la reconnaissance des particules virales par des anticorps neutralisants et donc protègent le virus de l’immunité adaptative qui s’est mise en place contre lui, au prix d’une sensibilité à la restriction par les IFITM.
VIH et résistance aux protéines IFITM
Le fait que les premiers virions à infecter un nouvel hôte soient résistants aux IFITM mais qu'ils deviennent plus tard sensibles à ces protéines peut paraître étonnant. Comment expliquer dans ce cas que les virions transmis à un nouvel hôte soient à nouveau résistants aux IFITM ? Plusieurs facteurs pourraient expliquer ce constat. Tout d'abord, il est possible que les virions résistants soient transmis avant de muter en formes sensibles. Par ailleurs certaines données suggèrent qu’en phase terminale de l’infection, la pression des anticorps neutralisants se relâche et les virus redeviennent résistants (Fenton-May et coll., 2013). Enfin, même en considérant une population de virus entièrement sensible, il faut prendre en compte que l'infection d'une cellule par un virus n'est pas un processus déterministe mais revêt au contraire une dimension probabiliste. Ainsi, même si la majorité de la population est inhibée par les IFITM, quelques virions peuvent leur échapper et parvenir à infecter un hôte.
Les IFITM sont induites par les interférons produits en contexte infectieux. Elles sont exprimées au niveau du réticulum endoplasmique avant d’être acheminées à la membrane plasmique en subissant des modifications post-traductionnelles. IFITM1 reste à la membrane tandis que les IFITM 2 et 3 sont redirigées vers les endosomes. Les IFITM entravent le cycle viral en inhibant la fusion virale au niveau de la membrane plasmique ou des endosomes, ou en étant incorporées dans les nouvelles particules virales, ce qui diminue la capacité de celles-ci à infecter de nouvelles cellules. Les virions qualifiés de transmitted founder du VIH sont généralement résistants aux IFITM mais certaines mutations de la protéine d’enveloppe peuvent induire une perte de résistance à ces protéines.
Conclusion
Les IFITM 1, 2 et 3 font partie des facteurs de restriction les plus importants dans le contrôle des infections virales, en inhibant la fusion membranaire de nombreux virus enveloppés. Leur activité est fortement contrôlée et fait intervenir conjointement plusieurs mécanismes.
Les fonctions des IFITM ne se limitent cependant pas à la restriction des infections virales, puisque IFITM5 participe à l’ostéogenèse (Moffatt et coll., 2008) et que les IFITM 1, 2 et 3 sont impliquées dans la migration des cellules germinales lors de l’embryogenèse (Lange et coll., 2003) et dans l’adhérence cellule-cellule (Deblandre et coll., 1995). De plus, une expression trop importante des IFITM durant la gestation peut interrompre la formation du placenta via une inhibition de la fusion des cellules du syncytiotrophoblaste (Buchrieser et coll., 2019).
Références
- Allen E, Randolph A, Bhangale T, Dogra P, Ohlson M, Oshansky C, Zamora A, Shannon J, Finkelstein D, Dressen A, DeVincenzo J, Caniza M, Youngblood B, Rosenberger C. SNP-mediated disruption of CTCF binding at the IFITM3 promoter is associated with risk of severe influenza in humans. Nat Med 2017 ; 23 : 975–983
- Amini-Bavil-Olyaee S, Choi Y, Lee J, Shi M, Huang I, Farzan M, Jung J. The antiviral effector IFITM3 disrupts intracellular cholesterol homeostasis to block viral entry. Cell host microbe 2014 ; 13(4) : 452-464
- Bailey C, Kondur H, Huang I, Farzan M. Interferon-induced transmembrane protein 3 is a type II transmembrane protein. J Biol Chem 2013 ; 288(45) : 32184–32193
- Bailey C, Zhong G, Huang I, Farzan M. IFITM-family proteins: the cell’s first line of antiviral defense. Annu Rev Virol 2014 ; 1 : 261-283
- Brass A, Huang I, Benita Y, John S, Krishnan N, Feeley E, Ryan B, Weyer J, van der Weyden L, Fikrig E, Adams D, Xavier R, Farzan M, Elledge S. The IFITM proteins mediate cellular resistance to influenza A H1N1 virus, west Nile virus, and dengue virus. Cell 2009 ; 139 : 1243–1254
- Buchrieser J, Degrelle S, Couderc T, Nevers Q, Dission O, Manet C, Donahue D, Porrot F, Hillion K, Perthame E, Arroyo M, Souquere S, Ruigrok K, Dupressoir A, Heidmann T, Montagutelli X, Fournier T, Lecuit M, Schwartz O. IFITM proteins inhibit placental syncytiotrophoblast formation and promote fetal demise. Science 2019 ; 365 : 176-180
- Chesarino N, McMichael T, Hach J, Yount J. Phosphorylation of the antiviral protein Interferon-inducible transmembrane protein 3 (IFITM3) dually regulates its endocytosis and ubiquitination. J Biol Chem 2014 ; 289 : 11986-1199
- Compton A, Bruel T, Porrot F, Mallet A, Sachse M, Euvrard M, Liang C, Casartelli N, Schwartz O. IFITM proteins incorporated into HIV-1 Virions impair viral fusion and spread. Cell 2014 ; 16 : 736-747
- Deblandre G, Marinx O, Evans S, Majjaj S, Leo O, Caput D, Huez G, Wathelet M. Expression cloning of an interferon-inducible 17-kDa membrane protein implicated in the control of cell growth. J Biol Chem 1995 ; 270 : 23860–23866
- Desai T, Marin M, Chin C, Savidis G, Brass A, Melikyan G. IFITM3 restricts influenza A virus entry by blocking the formation of fusion pores following virus-endosome hemifusion. PLoS Pathog 2014 ; 10(4) : e1004048
- Everitt A, Clare S, Pertel T, John S, Wash R, Smith S, Chin C, Feeley E, Sims J, Adams D, Wise H, Kane L, Goulding D, Digard P, Anttila V, Baillie K, Walsh T, Hume D, Palotie A, Xue Y, Colonna V, Tyler-Smith C, Dunning J, Gordon S, Smyth R, Openshaw P, Dougan G, Brass A, Kellam P. IFITM3 restricts the morbidity and mortality associated with influenza. Nature 2012 ; 484 : 519-523
- Everitt A, Clare S, McDonald J, Kane L, Harcourt K, Ahras M, Lall A, Hale C, Rodgers A, Young D, Haque A, Billker O, Tregoning J, Dougan G and Kellam P. Defining the Range of Pathogens Susceptible to Ifitm3 Restriction Using a Knockout Mouse Model. PLoS One2013; 8(11): e80723.
- Feeley E, Sims J, John S, Chin C, Pertel T, Chen L, Gaiha G, Ryan B, Donis R, Elledge S, Brass A. IFITM3 inhibits influenza A virus infection by preventing cytosolic entry. PloS Pathog 2011 ; 7(10) : e1002337
- Foster TL, Wilson H, Iyer SS, Coss K, Doores K, Smith S. Resistance of Transmitted Founder HIV-1 to IFITM-Mediated Restriction. Cell Host Microbe 2016 ; 20(4) : 429–442.
- Gorman M, Poddar S, Farzan M, Diamond M. The Interferon-Stimulated Gene Ifitm3 Restricts West Nile Virus Infection and Pathogenesis. J Virol 2016 ; 90 : 8212–8225
- Huang I, Bailey C, Weyer J, Radoshitzky S, Becker M, Chiang J, Brass A, Ahmed A, Chi X, Dong L, Longobardi L, Boltz D, Kuhn J, Elledge S, Bavari S, Denison M, Choe H, Farzan M. Distinct patterns of IFITM-mediated restriction of filoviruses, SARS coronavirus, and influenza A virus. PLoS Pathog 2011 ; 7 : e1001258
- Jia R, Pan Q, Ding S, Rong L, Liu S, Geng Y, Qiao W, Liang C. The N-terminal region of IFITM3 modulates its antiviral activity by regulating IFITM3 cellular localization. J virol 2012 ; 86 : 13697–13707
- John S, Chin C, Perreira J, Feeley E, Aker A, Savidis G, Smith S, Elia A, Everitt A, Vora M, Pertel T, Elledge J, Kellam P, Brass A. The CD225 domain of IFITM3 is required for both IFITM protein association and inhibition of influenza A virus and dengue virus replication. J Virol 2013 ; 87(14) : 7837–52
- Lange U, Saitou M, Western P, Barton S, Surani M. The fragilis interferon-inducible gene family of transmembrane proteins is associated with germ cell specification in mice. BMC Dev Biol 2003 ; 3 : 1–11
- Li K, Markosyan R, Zheng Y, Golfetto O, Bungart B, Li M, Ding S, He Y, Liang C, Lee J, Gratton E, Cohen F, Liu S. IFITM Proteins Restrict Viral Membrane Hemifusion. PLoS Pathog 2013 ; 9(1) : e1003124
- Lin T, Chin C, Everitt A, Clare S, Perreira J, Savidis G, Aker A, John S, Sarlah D, Carreira M, Elledge S, Kellam P, Brass A. Amphotericin B increases influenza A virus infection by preventing IFITM3-mediated restriction. Cell Reports 2013 ; 5 : 895–908
- Moffatt P, Gaumond M, Salois P, Sellin K, Bessette M, Godin E, Tambasco P, Atkins G, Nanci A, Thomas G. Bril: a novel bone-specific modulator of mineralization. J Bone Miner Res 2008 ; 23 : 1497–1508
- Poddar S, Hyde J, Gorman M, Farzan M, Diamond M. The Interferon-Stimulated Gene IFITM3 Restricts Infection and Pathogenesis of Arthritogenic and Encephalitic Alphaviruses. J Virol 2016 ; 90 : 8780–8794
- Siegrist F, Ebeling M, Certa U. The small interferon-induced transmembrane genes and proteins. J Interferon Cytokine Res 2011 ; 31 : 183–197
- Spence J, He R, Hoffmann H, Das T, Thinon E, Rice C, Peng T, Chandran K, Hang H. IFITM3 directly engages and shuttles incoming virus particles to lysosomes. Nature chem Biol 2019 ; 15 : 259-268
- Stacey M, Clare S, Clement M, Marsden M, Abdul-Karim J, Kane L, Harcourt K, Brandt C, Fielding C, Smith S, Wash R, Brias S, Stack G, Notley G, Cambridge E, Isherwood C, Speak An Johnson Z, Ferlin W, Jones S, Kellam P, Humphreys I. The antiviral restriction factor IFN-induced transmembrane protein 3 prevents cytokine-driven CMV pathogenesis. J Clin Invest. 2017 ; 127(4) : 1463–1474
- Suddala C, Lee C, Meraner P, Marin M, Markosyan R, Desai T, Cohen F, Brass A, Melikyan G. Interferon-induced transmembrane protein 3 blocks fusion of sensitive but not resistant viruses by partitioning into virus-carrying endosomes. PLoS Pathog 2019 ; 15 (1) : e1007532
- Tartour K, Appourchaux R, Gaillard J, Nguyen X, Durand S, Turpin J, Beaumont E, Roch E, Berger G, Mahieux R, Brand D, Roingeard P, Cimarelli A. IFITM proteins are incorporated onto HIV-1 virion particles and negatively imprint their infectivity. Retrovirology 2014 ; 11 : 103
- Tartour K, Nguyen X, Appourchaux R, Assil S, Barateau V, Bloyet L, Burlaud-Gaillard J, Confort M, Escudero-Perez B, Gruffat H, Hong S, Moroso M, Reynard O, Reynard S, Decembre E, Ftaich N, Rossi A, Wu N, Arnaud F, Baize S, Dreux M, Gerlier D, Paranhos-Baccala G, Volchkov V, Roingeard P, Cimarelli A. Interference with the production of infectious viral particles and bimodal inhibition of replication are broadly conserved antiviral properties of IFITM. PLoS Pathog 2017 ; 13(9) : e1006610
- Wang Y, Pan Q, Ding S, Wang Z, Yu J, Finzi A. The V3 Loop of HIV-1 Env Determines Viral Susceptibility to IFITM3 Impairment of Viral Infectivity. J Virol. 2017 ; 91(7). pmid:28100616.
- Wilkins C, Woodward J, Lau D, Barnes A, Joyce M, McFarlane N, McKeating J, Lorne D, Gale M. IFITM1 is a tight junction protein that inhibits hepatitis C virus entry. Hepatology 2013 ; 57 : 461-469
- Yang X, Tan B, Zhou X, Xue J, Zhang X, Wang P, Shao C, Li Y, Li C, Xia H, Qiu J. Interferon-inducible transmembrane protein 3 genetic variant rs12252 and influenza susceptibility and severity: a meta-analysis. PLoS One 2015 ; 10 : e0124985
- Yount J, Karssemeijer R, Hang H. S-palmitoylation and ubiquitination differentially regulate Interferon-induced Transmembrane Protein 3 (IFITM3)-mediated resistance to Influenza virus. J Biol Chem 2012 ; 287 : 19631-19641
- Zhang Y, Makvandi-Nejad S, Qin L, Zhao Y, Zhang T, Wang L, Renani E, Taylor S, McMichael A, Li N, Dong T, Wua H. Interferon-induced transmembrane protein-3 rs12252-C is associated with rapid progression of acute HIV-1 infection in Chinese MSM cohort. AIDS 2015 ; 29 : 889–894
- Zhao S, Qinglin H, Jia N, Xuezhi C, Zuojia C, Shuying Y, Yayi G, Fang L, Xiaohui Z, Ke X, Huimin F, Zhikang Q, Bing S, Jin Z, Bin L, Tsun A. Negative regulation of Interferon-induced Transmembrane Protein 3 by SET7-mediated Lysine monomethylation. J Biol Chem 2013 ; 288 : 35093–35103
- Zhao X, Sehgal M, Hou Z, Cheng J, Shu S, Wu S, Guo F, Le Marchand S, Lin H, Chang J, Guo J. Identification of residues controlling restriction versus enhancing activities of IFITM proteins on entry of human Coronaviruses. J Virol 2018 ; 92 : e01535-17