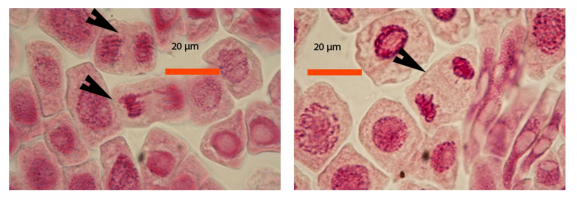
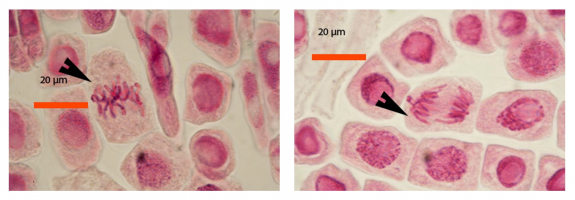
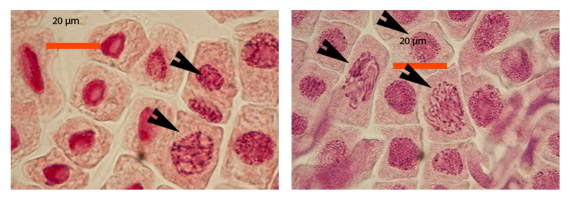
Médiathèque

Médiathèque
2462 médias
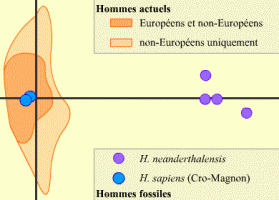
Le graphique correspond à une représentation (Multidimensional scaling, ou MDS) dans laquelle la distance entre deux points est proportionnelle à la distance génétique entre les deux séquences correspondantes. Sont représentés les résultats de la comparaison de la région hypervariable 1 de la boucle...
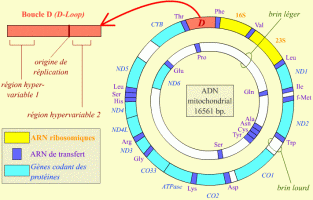
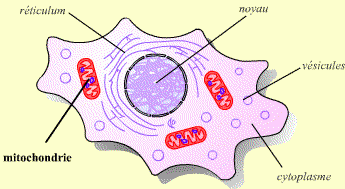
Le génome d'une mitochondrie code pour des ARN de transfert, les ARN ribosomiques et quelques protéines comme le cytochrome b (CYB) et l’ATP synthase (ATPase). Les deux brins complémentaires de l’ADN mitochondrial sont représentés par convention, et en référence à une légère différence de poids molé...
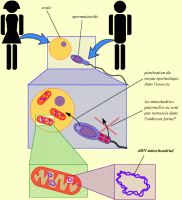
Lors de la fécondation d’un ovule, les mitochondries du spermatozoïde ne sont pas retrouvées dans l’œuf fécondé. Ainsi, les mitochondries du futur organisme sont d’origine maternelle. Or, les mitochondries possèdent un ADN qui leur est propre, présent en de nombreux exemplaires au sein de leur matri...
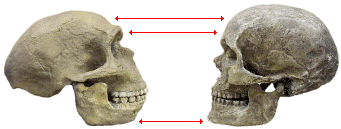
Le crâne d’H. sapiens est celui de droite. Les flèches soulignent trois points de comparaison possibles entre ces deux crânes : le front, l’arcade sourcilière et le menton. Noter aussi la présence du « chignon occipital », excroissance du crâne au niveau le plus postérieur, chez H. neanderthalensis....
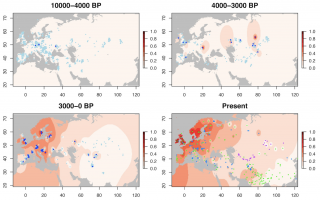
L’allèle –13.910*T est responsable du phénotype de persistance de la lactase en Eurasie. Les points représentent des individus anciens (trois premières cartes, 1434 individus au total) et modernes (dernière carte) chez lesquels cet allèle a été étudié. Ces données ont ensuite été extrapolées pour re...
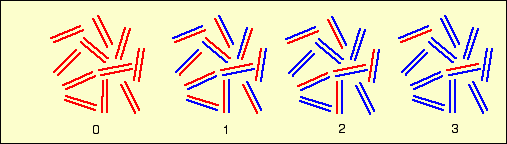
Au début de l'expérience tout l'ADN des bactéries est formé de deux brins d'ADN lourd (rouge). A la première génération, après une réplication en milieu contenant 14N, tout l’ADN est « hybride » et constitué d’un ancien brin « lourd » (15N, ici en rouge) et d’un nouveau brin « léger » (14N, ici en b...
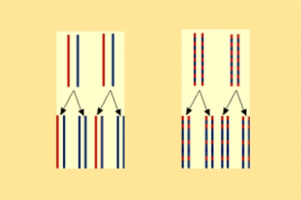
Ces schémas permettent de confronter les résultats obtenus après deux générations, à savoir de l'ADN hybride et de l'ADN léger, avec les résultats attendus selon les deux modèles encore possibles suite aux observations précédentes (après une génération).A gauche, résultat attendu pour le modèle semi...
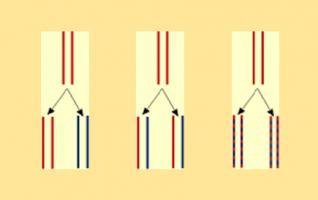
Ces schémas permettent de comparer le résultat obtenu après une génération, soit de l'ADN hybride, avec les résultats attendus selon les trois modèles de réplication de l'ADN envisagés.A gauche, résultat attendu pour le modèle conservatif : ADN lourd (15N) et ADN léger (14N)Au centre, résultat atten...