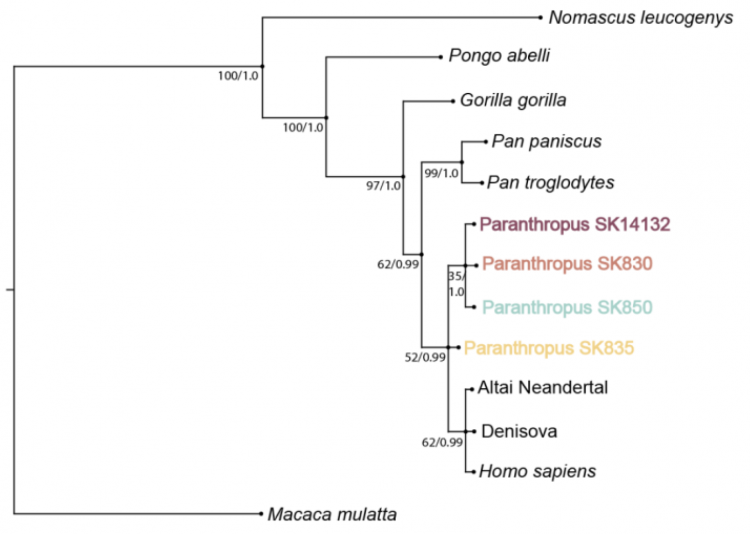
Les nombres aux nœuds représentent à gauche le bootstrap de 0 à 100 des réplicats de l’analyse par maximum de vraisemblance et à droite la probabilité à postériori de 0 à 1 de l’analyse par inférence bayésienne. Le bootstrap est une mesure permettant d’évaluer la stabilité d’un clade lorsque l’analyse est effectuée à plusieurs reprises (généralement au moins 1000 fois). Pour chaque réplicat on rééchantillonne aléatoirement les données, c’est-à-dire qu’on tire au sort (avec remise) les positions des résidus dans l’alignement des séquences qui sont conservées ou éliminées. Cela revient à perturber les données en attribuant un poids aléatoire à ces différentes positions, puisque certaines peuvent ne pas être choisies alors que d’autres peuvent être choisies une ou plusieurs fois. On observe alors le pourcentage de réplicats dans lesquels le meilleur arbre contient le clade d’intérêt. Un score de bootstrap faible (inférieur à 80 %) est le signe d’une grande sensibilité des données aux perturbations. Cela ne signifie pas que ces clades sont nécessairement faux, ni même improbables, mais cela rend leur position moins fiable, on ne peut pas leur accorder la même confiance qu’à ceux qui sont plus stables.
Arbre phylogénétique reconstruit par Madupe et coll 2023
Arbre phylogénétique reconstruit par Madupe et coll., 2023
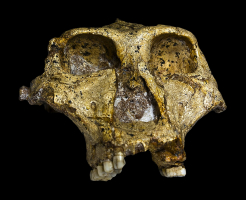
Autres médias
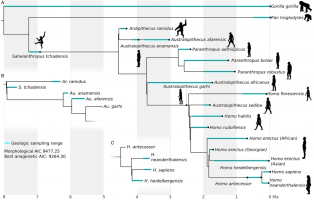
Cet arbre représente la meilleure estimation de Parins-Fukuchi et coll., 2018. À la différence d’autres méthodes de construction d’arbres phylogénétiques, n’utilisant que des données morphologiques, les auteurs estiment, dans l’étude dont est tirée cette figure, qu’il est préférable de recourir en p...
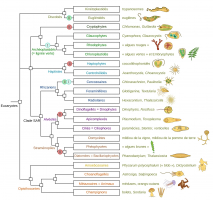
L’arbre montre les principaux groupes d’Eucaryotes (en termes de nombre d’espèces, d’importance écologique ou sanitaire). Les relations de parenté sont données d’après Keeling et Burki, 2019 1. Des exemples ainsi que des illustrations de membres des différents taxons sont donnés à droite. Les évène...
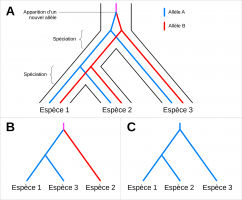
A. Histoire phylogénétique réelle d’une lignée montrant les évènements de spéciation et de diversification génétique avec l’exemple des allèles A et B issus d’un même allèle ancestral. Les allèles A et B sont deux versions d’un même gène qui coexistent chez les trois espèces. B. Arbre phylogénétique...