Explications de certains mécanismes épigénétiques : méthylation de l'ADN et modifications post-traductionnelles des histones. Lien entre ces modifications, l'expression des gènes, et les cancers.
Introduction
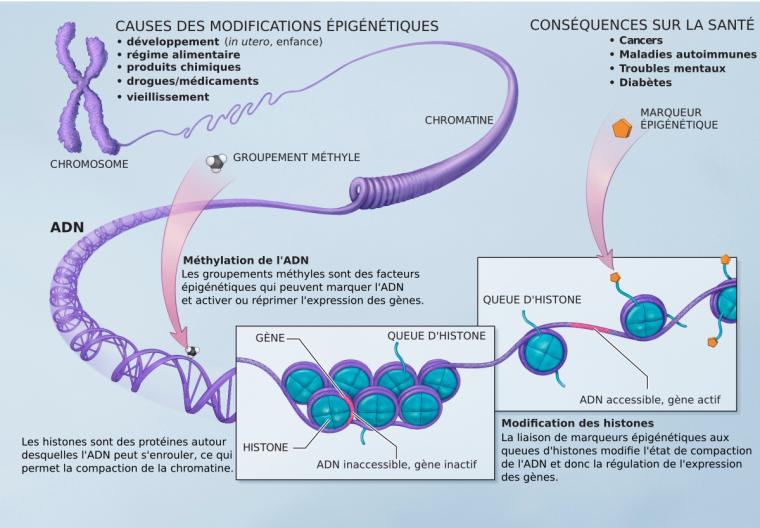
L’épigénétique est définie comme l’étude des changements transcriptionnels transmissibles lors de la division cellulaire, mais qui ne découlent pas de modifications dans la séquence d’ADN.
La plupart des processus épigénétiques sont basés sur des modifications de la chromatine, au niveau de l’ADN ou des histones, qui sont régulées de façon dynamique.
Les modifications chromatiniennes au sein du génome ont donc un rôle clé dans le processus d’expression des gènes et constituent un maillon de régulation complexe de l’expression génique. Elles sont particulièrement importantes dans la différenciation cellulaire pour établir et maintenir un « programme génétique donné » afin d’aboutir à un type cellulaire spécifique. Toutes les cellules d’un même organisme ont le même patrimoine génétique, la même séquence d’ADN nucléaire, sans pour autant avoir forcément la même fonction ni la même forme par exemple. Une cellule neuronale et une cellule épidermique n’ont pas la même fonction et sont très différentes morphologiquement. Pour cela, elles ont besoin d’avoir des informations épigénétiques différentes [1, 2].
Il est largement admis que des mutations de nucléotides au sein de l’ADN peuvent provoquer des cancers. Mais des anomalies épigénétiques contribuent également au développement et à la progression de certaines maladies et en particulier des cancers.
De plus, des modifications chromatiniennes pourraient être associées au développement de maladies neurodégénératives (Alzheimer, Parkinson, Huntington, etc.) ou métaboliques (obésité, diabète de type 2, etc.) ; de ce fait l’épigénétique est de plus en plus étudiée [1].
Compaction de l’ADN et rôle des histones
L’ADN comporte deux brins constitués de chaînes de nucléotides. Chaque nucléotide est formé d’un groupe phosphate, d’un sucre appelé désoxyribose et d’une base azotée : adénine (A), thymine (T), cytosine (C) ou guanine (G).
La structure en double hélice de l’ADN est liée à l’appariement des bases azotées grâce à des liaisons hydrogènes.
La figure ci-dessous présente différents états de condensation de l’ADN.
Les histones sont des protéines nucléaires associés à l’ADN et participent de façon primordiale à la formation de la chromatine, substance de base des chromosomes eucaryotes. L’ADN s’enroule autour d’un octamère d’histones fait de paires des histones H3, H4, H2A et H2B, ce qui forme l’unité de base de la chromatine : le nucléosome. Ce dernier est donc un complexe constitué d’histones et d’ADN. C’est le premier niveau de compaction de l’ADN. Des niveaux de compaction supérieurs sont obtenus à l’aide d’autres protéines.
La transcription de l’ADN en ARN est facilitée lorsque la chromatine est décondensée, cet état permettant aux protéines du complexe de transcription d’accéder à l’ADN.
La méthylation de l’ADN contrôle l’expression des gènes, car elle joue un rôle dans la compaction de la chromatine, empêchant l’accès des protéines du complexe de transcription à leur site de liaison à l’ADN [3].
La méthylation de l’ADN
La méthylation de l’ADN est impliquée dans la régulation de l’expression des gènes, la formation de la chromatine, le développement embryonnaire et d’autres processus fondamentaux. Elle constitue un élément clé de la régulation épigénétique de l’expression des gènes.
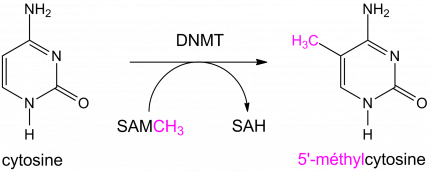
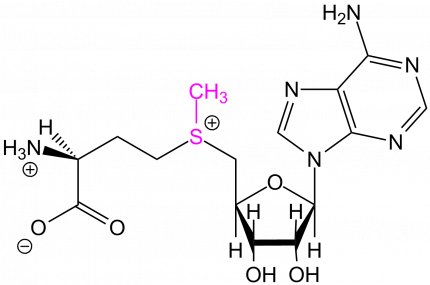
Les cystéines peuvent être modifiées par l’ajout d’un groupement méthyl (-CH3). Cette réaction est effectuée par des enzymes particulières : les ADN méthyl-transférases (DNMT). Ces enzymes catalysent le transfert d’un groupement méthyle d’une S-adénosylméthionine (SAM) sur des résidus cytosine ou adénine.
La S-adénosylméthionine est un métabolite présent dans les cellules, qui agit comme coenzyme dans les transferts de groupement méthyle. Chez les Mammifères, les cytosines méthylées se trouvent souvent dans des zones riches en cytosines et guanines, appelés îlots CpG [1].
La méthylation des cytosines de l’ADN est transmise au cours des divisions cellulaires et peut aussi être réversible. La déméthylation (la perte du groupement méthyle sur les cytosines de l’ADN) est un processus actif faisant intervenir une enzyme nommée déméthylase [4] mais peut aussi se produire de façon passive lors de la réplication de l’ADN si les méthyltransférases ne sont pas présentes ou actives.
Les modifications post-traductionnelles des histones
La méthylation de l’ADN n’est pas le seul vecteur de l’information épigénétique. Les modifications post-traductionnelles des histones (méthylation, acétylation, phosphorylation, ubiquitination…) affectent aussi l’état de la chromatine, et permettent ainsi de réguler l’expression d’un gène [4].
La méthylation des histones se fait principalement sur des résidus lysine et arginine par des enzymes appelées histones méthyltransférases (HMT). Selon le résidu modifié, la méthylation des lysines joue souvent un rôle dans la répression ou dans l’activation de la transcription ; la méthylation des arginines semble plutôt favoriser la transcription. De plus, le nombre de groupements méthyles présents sur une même lysine peut se traduire par différents niveaux de régulation de l’expression d’un gène [4].
Par ailleurs, les histones peuvent être acétylées (ajout d’un groupement acétyle –CO-CH3) sur des résidus lysines par les histones acétyltransférases (HAT). Il existe également une famille d’enzymes capable de désacétyler les lysines des histones : les histones désacétylases (HDAC). L’acétylation des lysines a pour conséquence de stabiliser la chromatine décondensée, ce qui est associé à une activation transcriptionnelle. L’acétylation des histones joue aussi un rôle dans la progression du cycle cellulaire, la réparation de l’ADN et la mort cellulaire programmée [4].
Le rôle de la méthylation dans les cancers
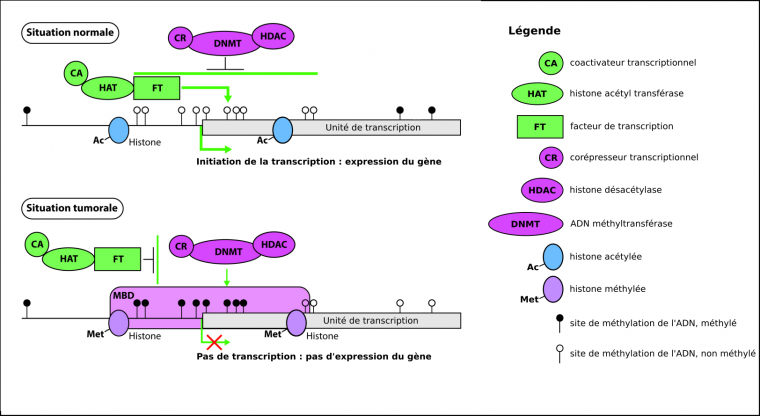
La transcription est une étape nécessaire à l’expression des gènes et fait l’objet d’un contrôle rigoureux. En particulier, l’accessibilité du promoteur est un élément clé dans la régulation de l’expression d’un gène. Toute modification au niveau du promoteur (ensemble de séquences d’ADN essentielles à l’initiation de la transcription) peut entraîner des changements d’état de la chromatine, avec des conséquences sur l’expression des gènes. Une modification de l’équilibre entre activation et répression des gènes peut conduire à la formation de tumeurs.
Chez les Mammifères, la plupart des gènes présente, au niveau de leur promoteur, un nombre conséquent d’îlots CpG.
En situation saine, les îlots CpG situés au niveau des promoteurs ne sont pas méthylés, et les histones sont acétylées. La chromatine est alors dans un état dit « relâché », accessible pour la transcription. Les îlots CpG situés en dehors des zones promotrices sont, quant à eux, méthylés. Lors de dérèglements de l’organisme, cette situation est susceptible de s’inverser : les îlots CpG situés au niveau du promoteur sont méthylés alors que ceux situés ailleurs dans le génome sont plutôt hypométhylés.
Les CpG méthylés du promoteur sont reconnus par des protéines présentant un Methyl-CpG Binding Domain (MBD). Ces protéines recrutent d’autres protéines, qui sont des enzymes de modifications post-traductionnelles des histones, dont par exemple des histones désacétylases (HDAC). Cette modification compacte un peu plus l’ADN qui est alors moins accessible pour la transcription. Les protéines MBD recrutent également des enzymes qui méthylent l’ADN (nommées DNMT, pour ADN méthyltransférases), ce qui a également pour conséquence de rendre l’ADN moins accessible pour la transcription. Le gène est alors inactivé : il ne sera pas transcrit. Si cette inactivation touche un gène suppresseur de tumeurs (gènes notamment impliqués dans le contrôle du cycle cellulaire ou dans la mort cellulaire programmée), cela peut mener à des cancers [5].
À l’inverse, les cancers peuvent également être dus à une hypométhylation globale ou locale du génome, en particulier des proto-oncogènes. Les proto-oncogènes sont impliqués dans la division et la croissance des cellules. Dans certains cas, ces gènes sont hypométhylés, conduisant à leur surexpression. Les cellules se divisent alors de manière anarchique, pouvant conduire à l’apparition de cancers.
Les chercheurs ont également observé que l’hypométhylation des gènes peut rendre l’ADN plus sensible à des cassures ou à des remaniements chromosomiques. C’est le cas par exemple des tumeurs de Wilms (tumeur du rein chez l’enfant) dans laquelle se produisent des translocations chromosomiques au niveau de l’ADN péricentromérique situé au niveau du centromère, qui constitue la zone de contact entre les deux chromatides d’un chromosome. Par ailleurs, l’hypométhylation globale du génome augmente avec l’âge, ce qui pourrait expliquer le nombre important de cancers observés chez les personnes âgées [5].
Conclusion
Des modifications épigénétiques activant des oncogènes (gènes dont la surexpression favorise la cancérogenèse) ou inhibant des gènes suppresseurs de tumeurs ont pu être mis en évidence. De ce fait, l’équilibre entre hyper et hypométhylation de l’ADN au sein de la cellule est particulièrement important et doit être finement régulé pour éviter d’aboutir à des maladies.
Les modifications chromatiniennes étant réversibles, il doit donc être possible de corriger celles associées à des maladies. C’est de cette idée qu’est né le concept d’épidrogues ou épimédicaments, c’est-à-dire le développement de médicaments qui agissent sur des mécanismes épigénétiques pour éliminer les régulations anormales. Les deux familles de molécules qui ont été développées jusqu’à présent sont les inhibiteurs des ADN méthyltransférases et les inhibiteurs des désacétylases d’histones. Mais ces molécules manquent de spécificité d’action et sont rapidement toxiques pour l’organisme. De nombreux autres épimédicaments sont en cours de développement [1].
Références
[1] Institut national de la santé et de la recherche médicale. Epigénétique. [en ligne]. (modifié en février 2015) Disponible sur : <http://www.inserm.fr/thematiques/genetique-genomique-et-bioinformatique/dossiers-d-information/epig…; (Consulté le 20/09/2016)
[2] http://www.epigenesys.eu/en/homepage
[3] Edith Heard. Epigénétique, comment se joue la partition du génome. Science & Santé, novembre-décembre 2012, n°11, Grand angle p22.
[4] Deltour Sophie, Chopin Valérie, Leprince Dominique, Modifications épigénétiques et cancer, Médecine Sciences, Vol 21, n°4, avril 2005, p.405-411
[5] Fucks François, Les méthyltransférases de l’ADN : du remodelage de la chromatine au cancer, Médecine sciences, Vol 19, n°4, avril 2003, p.447-480