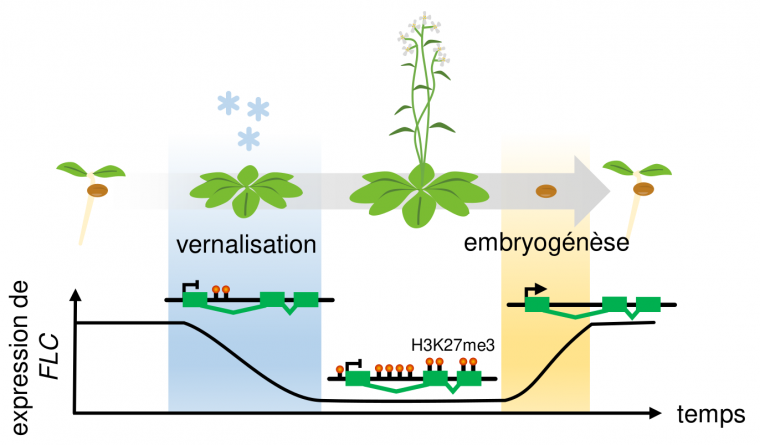
Les marques épigénétiques, qu'il s'agisse de modifications d'histones ou de méthylation de l'ADN, jouent un rôle clé dans la modulation de l'expression des gènes selon les types cellulaires et les conditions environnementales. Chez la plante modèle Arabidopsis thaliana, ces mécanismes sont essentiels dans le contrôle de la floraison qui repose notamment sur la régulation de l'expression du gène FLC. En effet, ce gène est un inhibiteur majeur de la floraison et est fortement exprimé chez les jeunes plants, ce qui les empêche de fleurir avant le printemps. Pendant l'hiver, l'exposition au froid et aux courtes photopériodes déclenche une cascade épigénétique complexe qui, par des modifications d'histones répressives, inactive progressivement l'expression de FLC, permettant ainsi la floraison au retour des beaux jours.
Au cours de sa vie, un organisme peut être amené à faire face à de nombreux changements de son environnement. Certains, comme le cycle des saisons, sont récurrents et relativement prévisibles et des mécanismes synchronisant les cycles de vie avec ces fluctuations environnementales ont donc pu évoluer. Chez les plantes, cette contrainte est particulièrement accrue par leur caractère sessile et on peut donc trouver, au sein d’une même espèce végétale, des populations aux cycles de vie significativement différents en fonction des exigences des habitats où elles se trouvent. Dans des environnements très fluctuants où les stress hydriques, thermiques, ou associés à l’activité humaine sont courants mais imprévisibles, des stratégies de cycle court et de floraison rapide sont communément favorisées (voir par exemple [1]). Par contre, lorsque les changements environnementaux sont relativement prévisibles (par exemple l’alternance des saisons dans les zones tempérées), un cycle court peut se révéler défavorable puisqu’il réduit la période d’accumulation de ressources et ne maximise donc pas la production de graines pour la génération suivante [2]. La sélection naturelle a ainsi favorisé l’émergence de mécanismes de synchronisation des périodes de croissance végétative et de reproduction avec les variations environnementales. C’est pourquoi de nombreuses espèces végétales passent l’hiver sous forme végétative et synchronisent leur floraison avec l’arrivée du printemps, comme c’est le cas chez l’espèce modèle Arabidopsis thaliana.
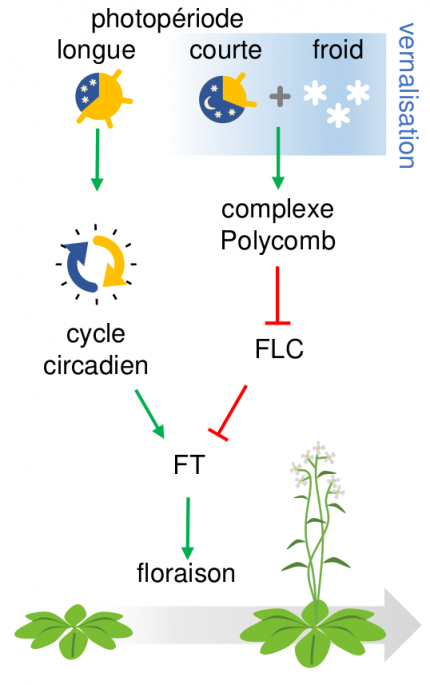
En réprimant FLC, la vernalisation libère l’expression de FT qui une fois activée par le cycle circadien va coder la protéine FT, transmissible des feuilles au méristème, et qui va y induire la transition de l’état végétatif à reproductif et déclencher ainsi la floraison.
Chez cette espèce, les mécanismes moléculaires responsables de la synchronisation de la floraison avec la fin de l’hiver ont été finement disséqués et un gène clé a été identifié : FLOWERING LOCUS C (FLC). Ce gène code un répresseur central de toute la cascade moléculaire régulant la transition d’un état végétatif à un état reproductif, et sa forte expression dès la germination bloque toute floraison. Ainsi, il maintient la plante dans un état végétatif tant qu’il n’est pas lui-même inhibé par exposition combinée au froid et aux courtes photopériodes hivernales (vernalisation1) qui va ainsi permettre l’activation par le cycle circadien du facteur déclencheur de la floraison (codé par FLOWERING LOCUS T ou FT ; Fig. 1).
L’importance de FLC dans la synchronisation de la floraison avec la fin de l’hiver, et l’impact adaptatif de cette transition développementale chez les populations naturelles d’A. thaliana ainsi que chez les espèces cultivées a permis de mieux comprendre les mécanismes de régulation de l’expression des gènes en réponse à l’environnement et le rôle central que la chromatine y joue.
Les nucléosomes, briques élémentaires de la chromatine
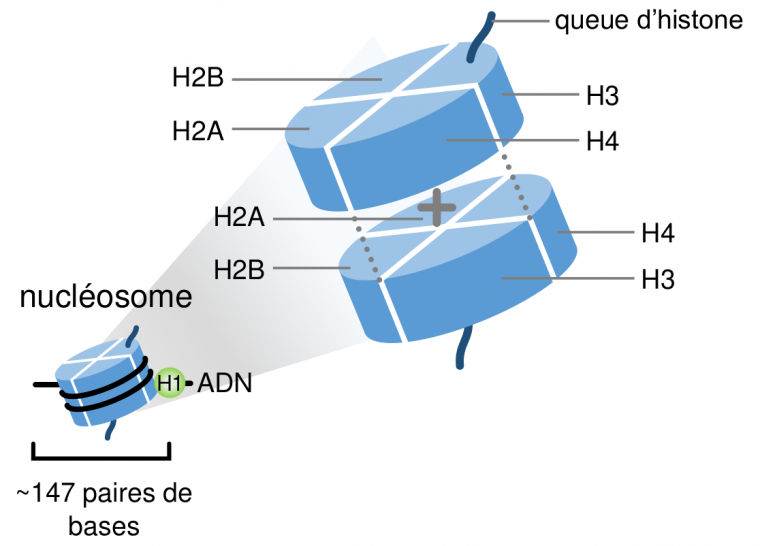
L’ADN est un long polymère de nucléotides, fortement condensé jusqu’à tenir dans les noyaux cellulaires de quelques micromètres de diamètre. Le premier niveau de condensation de l’ADN est obtenu par son enroulement autour des histones, protéines propres aux Eucaryotes et aux Archées. Ces histones sont présentes dans le génome sous différentes formes, les principales étant, chez les Eucaryotes, les histones H1, H2A, H2B, H3 et H4. Ces histones s’assemblent en octamères constitués de deux tétramères H2A+H2B+H3+H4 autour desquels l’ADN s’enroule sur presque deux tours (environ 147 paires de bases) formant ainsi les nucléosomes (Fig. 2). L’histone H1, appelée histone de liaison, ne fait pas partie du nucléosome à proprement parler, mais se joint aux brins d’ADN en sortie du nucléosome et semble protéger ainsi l’ADN entre deux nucléosomes (aussi appelé ADN de liaison) même si son rôle exact reste à élucider.
Le rôle de la chromatine dans la régulation de l’expression des gènes
Ces nucléosomes, constitués donc à la fois d’ADN et de protéines, représentent la brique élémentaire de la chromatine qui s’organise ensuite en des structures tridimensionnelles plus complexes. Ainsi, les nucléosomes jouent un rôle clé dans l’accessibilité de l’ADN à des fins de transcription ou de réplication. En effet, ces deux processus nécessitent que l’ADN soit temporairement dissocié des histones ce qui est rendu mécaniquement plus difficile dans les régions où la densité de nucléosomes est élevée. Or, cette densité des nucléosomes, et donc l’accessibilité à l’ADN, peut être modulée par différentes modifications post-traductionnelles des histones qui les composent. En effet, plusieurs des histones composant un nucléosome, notamment les histones H2B et H3, possèdent une extrémité N-terminale non structurée, appelée queue d’histone, qui peut être modifiée en plusieurs endroits par différents groupements chimiques. Parmi ces modifications, on peut citer par exemple l’ubiquitinylation de la queue d’histone H2B (appelée H2Bub), l’ajout de deux groupements méthyl sur la lysine 9 de l’histone H3 (H3K9me2) ou l’ajout d’un groupement acétyl sur la lysine 27 de l’histone H3 (H3K27ac). Au-delà de ces modifications d’histones, l’ADN lui-même peut être modifié chimiquement, notamment par l’ajout d’un groupement méthyl à la position 5 des anneaux pyrimidiques de ses cytosines (5mC).
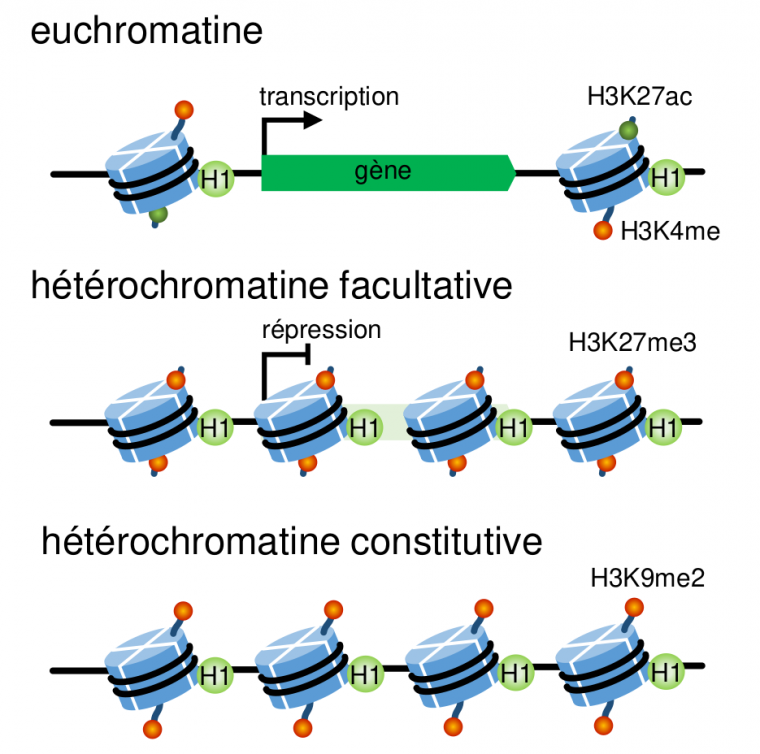
En fonction de l’impact que ces modifications apportent à la charge apparente du nucléosome, la densité locale des nucléosomes avoisinants va être modulée. De plus, comme les enzymes à l’origine de ces modifications agissent généralement sur plusieurs nucléosomes adjacents, on obtient des régions aux nucléosomes plus ou moins denses et donc à la chromatine plus ou moins compacte. Une chromatine lâche, très facilement accessible et donc favorable à l’expression des gènes, est appelée euchromatine, alors qu’une chromatine fortement condensée, très peu accessible et ne permettant donc pas l’expression des gènes est appelée hétérochromatine. Par exemple, la méthylation de la lysine 4 de l’histone H3 (H3K4me) ou l’acétylation de la lysine 27 de la même histone (H3K27ac) sont des marques activatrices caractéristiques de l’euchromatine, au contraire de la triméthylation de la lysine 27 de l’histone H3 (H3K27me3) qui est généralement répressive (en augmentant la condensation). Cependant, en raison de boucles de rétroaction entre modifications d’histones, certaines sont moins labiles que d’autres ce qui peut empêcher toute transition entre états euchromatiques et hétérochromatiques. Notamment, la diméthylation de la lysine 9 de l’histone H3 (H3K9me2) est répressive et, étant associée via des boucles de rétroaction positives à la méthylation de l’ADN, est très stable au cours du développement de l’organisme. Au contraire, la marque H3K27me3 n’apparaît que très rarement sur des régions d’ADN méthylé [3], et est donc beaucoup plus labile. Les régions réprimées par H3K27me3 peuvent donc être plus facilement réactivées que celles marquées par H3K9me2. Les premières forment l’hétérochromatine facultative et les secondes l’hétérochromatine constitutive (Fig. 3).
La transcription est possible au niveau de l’euchromatine, mais impossible dans l’hétérochromatine. Le degré de condensation de la chromatine dépend de marques chromatiniennes, en particulier des modifications post-traductionnelles des queues d’histones.
L’hétérochromatine constitutive est principalement trouvée dans les régions répétées et pauvres en gènes du génome, notamment à proximité des centromères et des télomères. Elle est maintenue au cours des divisions cellulaires par des mécanismes de lecture/écriture qui permettent la propagation des marques chromatiniennes répressives aux nouveaux nucléosomes intégrés dans les molécules d’ADN filles. En effet, au cours de la réplication, les nucléosomes de l’ADN mère sont recyclés [4] et attribués aléatoirement [5] à chacune des molécules filles à une position proche de celle d’origine [6]. Les marques chromatiniennes étant divisées par deux à chaque réplication, la stabilité des états chromatiniens à travers les divisions cellulaires requiert donc un certain nombre de nucléosomes portant la même modification pour qu’au moins un soit retrouvé sur chaque molécule d’ADN fille, ainsi que l’action de mécanismes de lecture/écriture qui reconnaissent les modifications d’histones des nucléosomes hérités de l’ADN mère pour modifier similairement les nouveaux nucléosomes des molécules filles.
Dans les régions géniques au contraire, une certaine flexibilité des états chromatiniens est nécessaire afin de permettre de potentielles réactivations de gènes initialement réprimés ou de répression de gènes activés, comme ça peut notamment être le cas au cours du développement de l’organisme, ou afin de répondre à des changements environnementaux.
Comment l’environnement peut influencer les états chromatiniens
Au cours du développement, certains gènes responsables de programmes cellulaires spécifiques doivent être activés ou inactivés sur des intervalles temporels et spatiaux bien précis. Ces transitions entre états transcriptionnels requièrent l’action de régulateurs chromatiniens qui ont la capacité de mettre en place des états chromatiniens permissifs à la transcription ou au contraire temporairement répressifs (hétérochromatine facultative). Un groupe majeur de tels régulateurs a été découvert par les conséquences développementales considérables que sa perte induit chez la mouche Drosophila melanogaster. En effet, lorsque les facteurs de ce groupe sont mutés on observe la transformation homéotique des pattes postérieures en pattes antérieures qui développent des poils en forme de peignes (combs en anglais) et les protéines de ce groupe ont donc été nommées Polycomb. Les protéines Polycomb forment des complexes qui ont notamment la capacité de triméthyler la lysine 27 de l’histone H3 (H3K27me3), une marque répressive caractéristique de l’hétérochromatine facultative. Ces complexes, appelés PRC pour Polycomb Repressive Complex, ont également la capacité de reconnaître H3K27me3 sur un nucléosome (lecture) pour modifier ensuite similairement les nucléosomes voisins (écriture), ce qui joue un rôle clé dans la stabilité de l’hétérochromatine facultative au cours des divisions cellulaires [7].
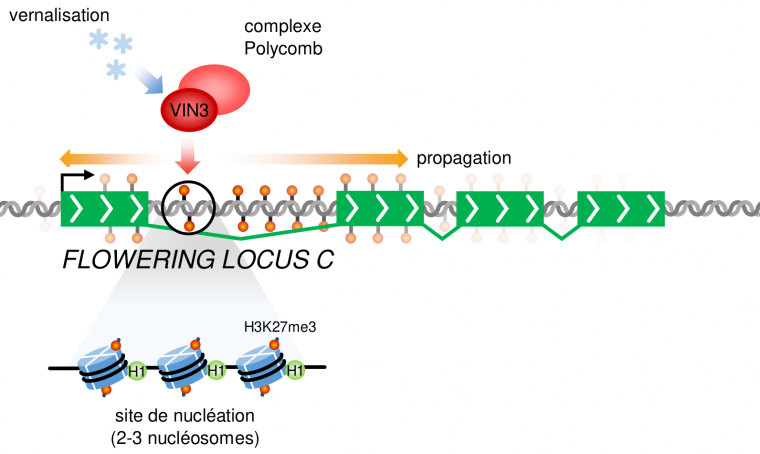
Au locus FLC, qui est initialement fortement exprimé et marqué par des marques de chromatine active comme H3K4me, un tel complexe Polycomb est recruté au niveau de son premier intron lorsque l’un de ses composants (codé par le gène VERNALIZATION INSENSITIVE 3 ou VIN3) est activé par l’exposition au froid. L’activation par le froid de VIN3 reste elle-même mal comprise, mais semble dépendre d’une protéine activatrice de VIN3 qui est relativement stable et s’accumule dans les cellules pendant la vernalisation du fait du ralentissement des divisions cellulaires qui limite sa dilution [8]. Une fois activé, le complexe Polycomb formé avec VIN3 va induire une méthylation de type H3K27me3 à un site de nucléation bien précis de 2 ou 3 nucléosomes tout au plus, au sein du premier intron de FLC [9]. Cet état chromatinien répressif va ensuite, à la sortie de l’hiver, se propager à l’ensemble du locus par l’action de nouveaux composants du complexe Polycomb afin de bloquer définitivement son expression et de permettre ainsi la floraison (Fig. 4) [10].
Cette propagation à l’ensemble du locus de l’état chromatinien répressif induit par Polycomb (représenté en particulier par H3K27me3) est essentielle à la stabilité de la répression de FLC et, si elle ne se fait pas, FLC est progressivement réactivé en moins de 20 jours après la remontée des températures [10]. En effet, si une modification d’histone est présente sur un nombre trop faible de nucléosomes, comme c’est le cas pour les trois nucléosomes du site de nucléation, cette modification est progressivement perdue au cours des divisions cellulaires, puisqu’à chaque division il y a une probabilité significative (0,53 soit 12,5 %) qu’un des brins filles n’hérite d’aucun des trois nucléosomes mères. Une fois les modifications d’histones perdues sur un brin elles ne peuvent plus être prises en compte et rétablies par les boucles de lecture/écriture assurées par Polycomb et l’allèle de FLC que ce brin porte est définitivement réactivé. Au contraire, si elle est propagée à l’ensemble du locus (environ 30 à 35 nucléosomes) les mécanismes de lecture/écriture associés aux marques H3K27me3 sont suffisants pour maintenir l’état chromatinien au travers des duplications cellulaires malgré une répartition aléatoire des nucléosomes à chaque division [11]. La floraison peut donc avoir lieu.
L’instabilité de la répression de FLC peut être induite expérimentalement par la mutation des facteurs responsables de la propagation des marques H3K27me3 [10], mais se retrouve également dans certaines populations naturelles d’A. thaliana provenant du nord de la Suède [12]. En effet, dans ces populations le site de nucléation du complexe Polycomb est porteur de plusieurs mutations et la répression de FLC est perdue stochastiquement dans les quelques jours qui suivent le retour à des plantes à la chaleur. Néanmoins, une répression plus stable, même si en définitive temporaire, de FLC peut être obtenue par une vernalisation prolongée de ces plantes (8 semaines ou plus contre 4 semaines pour les populations plus méridionales) ce qui permet la floraison le temps que FLC reste inactivé. Une telle variation génétique est probablement maintenue dans le nord de la Suède où les étés sont brefs et où une floraison ponctuelle avant un retour à un état végétatif peut être avantageuse afin de permettre à un individu de produire des graines sur plusieurs années. Ce cycle de vie pluriannuel, qui se distingue du cycle annuel des populations plus méridionales, se rapproche en effet de celui d’autres espèces d’Arabidopsis pérennes. Celles-ci occupent des habitats plus protégés, mais aussi à plus haute altitude qu’A. thaliana, et présentent également une répression temporaire de FLC, ce qui permet un retour à l’état végétatif durant l’hiver avant une deuxième saison de floraison [13]. Au-delà d’Arabidopsis, la répression temporaire de FLC ou de ses homologues a été décrite dans plusieurs autres espèces pérennes de Brassicaceae notamment Arabis alpina [14] qui, comme son nom l’indique, prospère également dans des environnements montagneux.
Une mémoire des changements environnementaux passés ?
À chaque nouvelle génération, l’expression de FLC est rétablie dès la germination, que les parents aient été exposés à l’hiver ou non. Cela implique une réinitialisation de l’état chromatinien du locus FLC à chaque génération, entre la floraison et la germination. Cette réinitialisation a lieu au cours de l’embryogenèse par l’action de déméthylases de H3K27 qui enlèvent donc toute marque répressive établie par Polycomb sur le locus [15] (Fig. 5).
Peu d’autres locus semblent ciblés par ces déméthylases, ce qui suggère une pression sélective très forte pour que de tels mécanismes évoluent afin de réinitialiser l’état chromatinien spécifiquement au locus FLC et donc d’empêcher la transmission d’une mémoire chromatinienne de la vernalisation des parents. En effet, une telle mémoire dans la nature résulterait en une mauvaise coordination du développement des descendants avec leur environnement et on comprend donc que de tels phénotypes seraient très rapidement contre-sélectionnés. De manière générale, il semble que les exemples d’une transmission au travers des générations d’une mémoire chromatinienne des environnements passés soient très rares voire inexistants chez les plantes étudiées jusqu’à présent [16]. Néanmoins, il est possible que ces conclusions soient biaisées par le cycle de vie rapide d’Arabidopsis thaliana où la transmission héritable d’une mémoire environnementale serait délétère, mais qui reste encore l’espèce modèle servant de base à notre compréhension des mécanismes chromatiniens impliqués dans la régulation des gènes en réponse à l’environnement. L’étude de ces mécanismes chez des espèces pérennes ou se propageant par reproduction clonale, ce qui contourne la réinitialisation chromatinienne lors de l’embryogenèse, pourrait donc éventuellement remettre en question l’absence constatée jusqu’à présent d’une mémoire chromatinienne des changements environnementaux passés.
Références
[1] M. C. Hall and J. H. Willis, “Divergent selection on flowering time contributes to local adaptation in Mimulus guttatus populations,” Evolution, vol. 60, no. 12. pp. 2466–2477, 2006. doi : 10.1111/j.0014-3820.2006.tb01882.x.
[2] T. Mitchell-Olds and J. Schmitt, “Genetic mechanisms and evolutionary significance of natural variation in Arabidopsis,” Nature, vol. 441, no. 7096, pp. 947–952, Jun. 2006.
[3] A. Déléris, F. Berger, and S. Duharcourt, “Role of Polycomb in the control of transposable elements,” Trends in Genetics, vol. 37, no. 10. pp. 882–889, 2021. doi : 10.1016/j.tig.2021.06.003.
[4] T. M. Escobar, A. Loyola, and D. Reinberg, “Parental nucleosome segregation and the inheritance of cellular identity,” Nature Reviews Genetics, vol. 22, no. 6. pp. 379–392, 2021. doi : 10.1038/s41576-020-00312-w.
[5] A. T. Annunziato, “Split Decision : What Happens to Nucleosomes during DNA Replication?,” Journal of Biological Chemistry, vol. 280, no. 13. pp. 12065–12068, 2005. doi : 10.1074/jbc.r400039200.
[6] G. Schlissel and J. Rine, “The nucleosome core particle remembers its position through DNA replication and RNA transcription,” Proceedings of the National Academy of Sciences, vol. 116, no. 41. pp. 20605–20611, 2019. doi : 10.1073/pnas.1911943116.
[7] C. Lövkvist, P. Mikulski, S. Reeck, M. Hartley, C. Dean, and M. Howard, “Hybrid protein assembly-histone modification mechanism for PRC2-based epigenetic switching and memory,” Elife, vol. 10, Sep. 2021, doi : 10.7554/eLife.66454.
[8] Y. Zhao, R. L. Antoniou-Kourounioti, G. Calder, C. Dean, and M. Howard, “Temperature-dependent growth contributes to long-term cold sensing,” Nature, vol. 583, no. 7818, pp. 825–829, Jul. 2020.
[9] J. Song, A. Angel, M. Howard, and C. Dean, “Vernalization – a cold-induced epigenetic switch,” Journal of Cell Science. 2012. doi : 10.1242/jcs.084764.
[10] H. Yang, S. Berry, T. S. G. Olsson, M. Hartley, M. Howard, and C. Dean, “Distinct phases of Polycomb silencing to hold epigenetic memory of cold in Arabidopsis,” Science, vol. 357, no. 6356. pp. 1142–1145, 2017. doi : 10.1126/science.aan1121.
[11] A. Angel, J. Song, C. Dean, and M. Howard, “A Polycomb-based switch underlying quantitative epigenetic memory,” Nature, vol. 476, no. 7358. pp. 105–108, 2011. doi : 10.1038/nature10241.
[12] J. I. Qüesta et al., “Noncoding SNPs influence a distinct phase of Polycomb silencing to destabilize long-term epigenetic memory at,” Genes Dev., vol. 34, no. 5–6, pp. 446–461, Mar. 2020.
[13] P. Baduel, B. Arnold, C. M. Weisman, B. Hunter, and K. Bomblies, “Habitat-Associated Life History and Stress-Tolerance Variation in Arabidopsis arenosa,” Plant Physiol., vol. 171, no. 1, pp. 437–451, May 2016.
[14] R. Wang et al., “PEP1 regulates perennial flowering in Arabis alpina,” Nature, vol. 459, no. 7245, pp. 423–427, May 2009.
[15] C. C. Sheldon, M. J. Hills, C. Lister, C. Dean, E. S. Dennis, and W. James Peacock, “Resetting of FLOWERING LOCUS C expression after epigenetic repression by vernalization,” Proceedings of the National Academy of Sciences, vol. 105, no. 6. pp. 2214–2219, 2008. doi : 10.1073/pnas.0711453105.
[16] P. Baduel and V. Colot, “The epiallelic potential of transposable elements and its evolutionary significance in plants,” Philos. Trans. R. Soc. Lond. B Biol. Sci., vol. 376, no. 1826, p. 20200123, Jun. 2021.