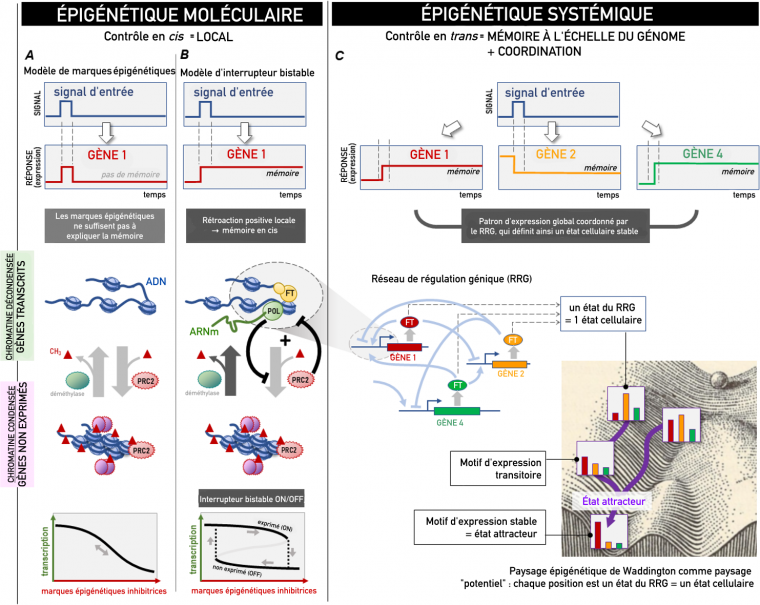
La notion de « marques » épigénétiques utilisée par les biologistes moléculaires est conceptuellement détachée de l’idée de « paysage » épigénétique de Waddington, qui est utilisée par les biologistes des systèmes et les biophysiciens. Des avancées récentes suggèrent que ces deux écoles de pensée distinctes pourraient être réunies.
Ce texte correspond à la traduction par Romane Petit de l’article intitulé Towards a unification of the 2 meanings of “epigenetics”, écrit par Sui Huang et paru en 2022 dans la revue PLOS Biology. La relecture de la traduction a été assurée par Pierre Baduel, chargé de recherche au CNRS dans l'équipe Dynamique du génome et variation épigénétique à l'Institut de biologie de l'École normale supérieure.
Bien que cela soit peu connu, le terme « épigénétique » est utilisé dans deux sens distincts, par deux écoles de pensée qui interagissent rarement. Je les appelle ici l’épigénétique moléculaire et l’épigénétique systémique. Ces deux écoles sont largement indépendantes l’une de l’autre, et ne pourront être unifiées qu’en évaluant simultanément leurs deux points de vue.
L’épigénétique moléculaire correspond au sens du mot « épigénétique » le plus répandu aujourd’hui. Il a été adopté par les biologistes moléculaires pour désigner la méthylation de l’ADN et les modifications post-traductionnelles des protéines histones, qui agissent comme des « marques » épigénétiques contrôlant l’activité d’un locus génétique (Figure 1, côté gauche). On pense que ces marques persistent et portent donc la mémoire de l’état d’activité d’un gène 12. L’épigénétique moléculaire s’intéresse également à la condensation de l’ADN génomique en chromatine compacte 3. Les marques épigénétiques recrutent des protéines qui remodèlent la chromatine, lui permettant de passer d’un état fermé à un état ouvert. Les gènes situés dans des structures chromatiniennes fermées étant inaccessibles aux facteurs de transcription, on pense que la (dé)condensation sélective des régions chromatiniennes ajoute un niveau de contrôle de l’expression génétique qui prévaut sur la régulation par les facteurs de transcription.
Le paradigme de l’épigénétique moléculaire comporte plusieurs incohérences logiques 456. Premièrement, si les enzymes qui remodèlent la chromatine (en ajoutant, en effaçant, et en lisant les marques épigénétiques sans reconnaître de séquences d’ADN spécifiques) contrôlent ainsi la régulation transcriptionnelle par les facteurs de transcription, qui contrôle ces contrôleurs ? Deuxièmement, si les facteurs de transcription doivent recruter ces enzymes, qui n’ont pas de spécificité de séquence, afin d’ouvrir la chromatine et de découvrir leurs motifs de liaison spécifiques, comment ces facteurs de transcription peuvent-ils se lier à leurs séquences cibles si celles-ci sont encore inaccessibles ? Pour résoudre cette énigme, on a proposé l’existence de facteurs de transcription dits « pionniers », capables de se lier à des séquences spécifiques dans la chromatine condensée [NdT : et de les rendre accessibles à des facteurs transcription non-pionniers]. Mais s’ils avaient accès aux sites de liaison quelle que soit la conformation de la chromatine, ils remettraient en cause le rôle absolument nécessaire de la chromatine dans la régulation des gènes. Et en effet, le concept de facteurs pionniers n’a pas pu être généralisé 7. Troisièmement, les enzymes qui apposent ou retirent les marques épigénétiques sont ubiquitaires et se neutralisent mutuellement. Comment une mémoire durable peut-elle être générée par des marques qui sont dynamiques et réversibles ? Quatrièmement, l’ouverture de la chromatine est-elle une cause ou une conséquence de la transcription ? Si la suppression des marques de méthylation inhibitrices active les gènes, l’activation transcriptionnelle peut également précéder la déméthylation 8. Ces paradoxes de l’œuf et de la poule sont le signe d’un circuit autorégulateur.
De nouvelles études suggèrent que la régulation épigénétique locale par l’ajout ou la suppression de marques épigénétiques, illustrée ici par la méthylation (triangle rouge ; panneau A), est convertie en un interrupteur local bistable (ON-OFF) (la mémoire agissant en cis ; panneau B), permettant une mémoire mais pour un seul locus de gène. Pour produire des patrons d’expression génétique stables (représentés ici par les gènes 1, 2 et 4), un contrôle en trans, incarné par le réseau de régulation génique (panneau C), est nécessaire. La dynamique du réseau de régulation génique dans son ensemble est illustrée par le « paysage épigénétique » de Waddington, dans lequel les vallées représentent des « états attracteurs » stables. RRG : réseau de régulation des gènes ; POL, polymérase ; PRC2, complexe Polycomb PRC2 ; FT, facteur de transcription.
En revanche, l’épigénétique systémique renvoie à une signification plus ancienne de l’épigénétique, antérieure à la biologie moléculaire et remontant au « paysage épigénétique » de Conrad Waddington. Utilisé aujourd’hui par les biologistes systémiques qui cherchent à déterminer la topographie réelle de ce paysage 1, ce terme désigne la création et le maintien de patrons d’expression génétique stables, caractéristiques de phénotypes cellulaires persistants tels que les types cellulaires. L’établissement d’un patron nécessite que l’expression de chaque gène soit étroitement coordonnée avec celle d’autres gènes, ce qui est permis par un réseau d’interactions régulatrices exercées par des facteurs de transcription, chacun modulant l’activité d’un ensemble spécifique de locus génétiques. Ensemble, ces interactions constituent le réseau de régulation génique (RRG) à l’échelle du génome. Une mémoire apparaît parce que la régulation mutuelle des gènes dans ce réseau peut « verrouiller » leurs états d’expression respectifs, produisant un patron d’expression stable : les « états attracteurs » illustrés par les vallées dans le paysage de Waddington 2 (Figure 1, partie droite).
L’épigénétique systémique est exempte des raisonnements circulaires qui minent l’épigénétique moléculaire. Mais ses partisans ignorent tacitement une question fondamentale : si le réseau de régulation génique est suffisant pour expliquer la persistance de différentes configurations d’expression génétique, pourquoi aurions-nous besoin de modifications épigénétiques locales ? Serait-ce uniquement pour la fonction structurelle de condensation de l’ADN ?
On peut facilement expliquer que la modification réversible de la chromatine par des marques épigénétiques ne puisse pas produire de mémoire. En ingénierie, un interrupteur avec mémoire doit, par définition, être capable de rester allumé après l’extinction du signal transitoire qui l’a activé, sinon il ne s’agit pas de mémoire (Figure 1A). La condition mathématique préalable à une telle dynamique ON-OFF (bistable) irréversible est que le circuit doit contenir au moins une boucle de rétroaction positive (ainsi qu’une non-linéarité dans ses caractéristiques de fonctionnement) 3. Le paradigme de l’épigénétique systémique n’a aucune difficulté à satisfaire cette exigence grâce à la pléthore de boucles de rétroaction positive dans la régulation agissant en trans des gènes du réseau de régulation génique (Figure 1C).
Deux nouveaux axes de recherche de chacune des deux écoles proposent un premier pas vers une unification logique (si elles peuvent être généralisées à d’autres systèmes de modifications épigénétiques). Des études d’épigénétique moléculaire ont identifié des boucles de rétroaction capables d’une transition bistable qui répond à la définition de la mémoire. Holoch et ses collègues 4 ont démontré dans une série d’expériences élégantes que le complexe Polycomb PRC2 responsable de la méthylation inhibitrice d’histone de type H3K27me, qui inhibe la transcription d’un gène cible X, est lui-même inhibé par la transcription continue de X (Figure 1B). Ce rétrocontrôle négatif d’un élément inhibiteur (PRC2) par l’entité régulée (gène X) constitue une boucle de rétroaction positive, répondant ainsi à l’exigence mathématique de bistabilité qui permet de produire une réponse stable se maintenant même après la disparition du signal d’entrée. Une boucle de rétroaction similaire a été observée pour le gène c-Kit, où une forte transcription empêche Polycomb de se lier au promoteur 5.
Mais ces « interrupteurs à mémoire épigénétique agissant en cis » fonctionnent au sein d’un locus génétique et ne sont pas sensibles au contrôle en trans entre différents locus. Du point de vue de l’épigénétique systémique, de tels interrupteurs ne semblent pas nécessaires, et certainement pas suffisants, pour établir et mémoriser des patrons d’expression entiers. Pourquoi alors l’évolution a mené à ces interrupteurs bistables élaborés agissant en cis 6 ? Que manque-t-il à l’épigénétique systémique pour expliquer l’existence des interrupteurs agissant en cis ?
Une nouvelle étude a mis en évidence un découplage entre la (dé)condensation de la chromatine et la mise en place de transcriptomes spécifiques, confirmant que le remodelage de la chromatine n’est pas suffisant à la coordination de l’expression génétique. En effet, Parmentier et ses collègues 7 observent que, comme chez l’embryon précoce, les cellules progénitrices sanguines CD34+, multipotentes, présentent une ouverture globale de leur chromatine avant de se différencier en lignages spécifiques. Comment des profils d’expression spécifiques à une lignée peuvent-ils apparaître de manière fiable à partir d’un accès si peu spécifique à la plupart des gènes ? Quelques heures après le début de la différenciation, 50 % des promoteurs du génome sont devenus accessibles, les sites accessibles variant beaucoup d’une cellule à l’autre. Puis on observe une augmentation stochastique de l’activité transcriptionnelle 8, ce qui permet, à partir de cellules multipotentes, d’initier la différenciation vers différents lignages 9. Le bruit transcriptionnel à l’échelle du génome se réduit ensuite en 48 heures, lorsque des patrons d’expression stables et spécifiques à chaque lignée ont émergé.
L’analyse informatique des transcriptomes de cellules uniques a révélé une dynamique de « transition critique » (point de basculement) 10 pendant la phase stochastique. Cela indique une transition irréversible vers un nouvel attracteur (une vallée du « paysage épigénétique » de Waddington), qui met en place le transcriptome stable et spécifique de la lignée, « prévu » par le réseau de régulation génique et partagé par toutes les cellules.
On peut maintenant faire l’hypothèse que sur des locus de gènes individuels, les interrupteurs bistables agissant en cis se comportent comme des transistors dans une puce d’ordinateur. Par rapport aux architectures analogiques, le codage numérique augmente la vitesse de traitement de l’information et offre une résistance au bruit. Un tel codage accélérerait la mise en œuvre de « programmes » spécifiques d’expression génétique par le réseau de régulation génique. On pourrait vérifier cette hypothèse si les études de Holoch et ses collègues 11 et de Parmentier et ses collègues 12, qui souscrivent chacun à l’une des deux écoles d’épigénétique, étaient étendues à des expériences guidées par les habitudes de pensée de l’autre école.
Ainsi, en théorie, les interrupteurs de mémoire agissant en cis (épigénétique moléculaire) facilitent fortement la mise en œuvre d’activités de contrôle en trans encodées dans le réseau de régulation génique (épigénétique systémique). Dans le formalisme de cette dernière, cela correspondrait au creusement des vallées dans le paysage de Waddington 13, ce qui augmente la stabilité des patrons d’expression à l’échelle du génome. Les deux écoles d’épigénétique opposées pourraient représenter les deux facettes du même principe de régulation de l’expression des gènes.