Les bactériophages constituent un type de virus probablement apparu en même temps que les premières bactéries, il y a quelques milliards d’années. Ils représentent les entités biologiques les plus abondantes de la Terre (leur nombre est estimé entre 1030 et 1032). Les bactériophages se retrouvent dans tous les biotopes, où ils jouent un rôle fondamental dans le fonctionnement des écosystèmes (y compris les microbiotes). Dès leur découverte, il y a près de cent ans, les bactériophages ont été utilisés par l’Homme en tant qu’agents thérapeutiques. Ces pratiques sont rapidement tombées en désuétude dans de nombreux pays, où les bactériophages ont davantage été utilisés en laboratoire en tant qu’outils biologiques. Actuellement, suite à l’augmentation inquiétante des cas d’antibiorésistance, les regards se tournent de nouveau vers ces virus.
Cette synthèse a pour objectif de comprendre en quoi l’évolution des connaissances sur les bactériophages a permis, au fil du temps, de les utiliser en tant qu’outils biologiques dans les laboratoires jusqu’à envisager aujourd’hui de les réutiliser en tant qu’agents thérapeutiques.
Caractéristiques des bactériophages
Découverte des bactériophages
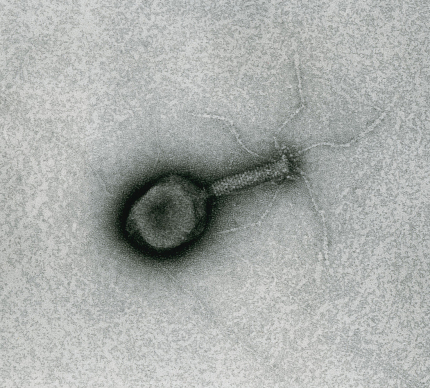
Le virion du bactériophage T4, observé ici en microscopie électronique à transmission, mesure environ 200 nm.
En 1896, Ernest Hankin fut le premier à remarquer que les eaux de la Jumna et du Gange, en Inde, possédaient un pouvoir bactéricide. Après filtration, elles avaient la capacité de soigner des patients atteints du choléra, une maladie causée par la bactérie Vibrio cholerae. Il supposa d’ailleurs que cette substance était responsable de la fin d’une épidémie de choléra par ingestion d’eau de ces fleuves.
Quelques années plus tard, des phénomènes de lyse dans des cultures bactériennes furent observés sans être expliqués. Ce n’est qu’en 1915 et 1917 que deux scientifiques, Frederick Twort et Félix d’Hérelle, étudièrent plus en détail ce phénomène et formulèrent des hypothèses quant à son origine. Twort imagina un « ultra microscopic virus » et D’Hérelle, un « microbe invisible ». Très vite, ce dernier isola le mystérieux agent infectieux et le nomma bactériophage, littéralement « le mangeur de bactéries ». Ce ne fut qu’en 1940 que le premier bactériophage fut observé au microscope électronique par Ernst Ruska. L’essor de la biologie moléculaire permit ensuite d’approfondir les connaissances sur leur structure, leur composition et leur fonctionnement.
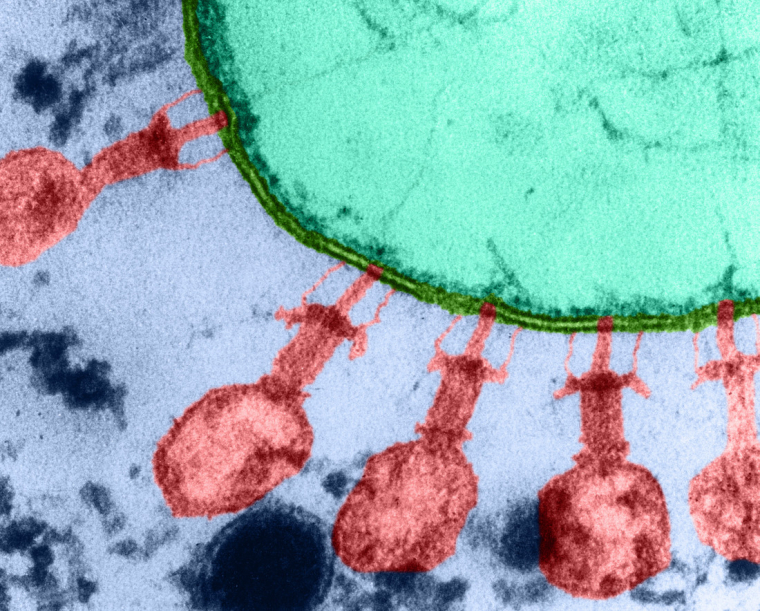
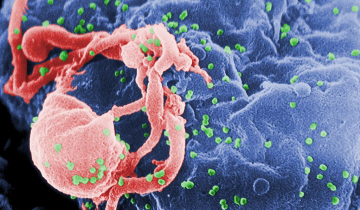
Des bactériophages T2 (en rouge) sont adsorbés sur la paroi (en vert) d’une cellule d’Escherichia coli (microscopie électronique à transmission, fausses couleurs).
Structure et composition des bactériophages
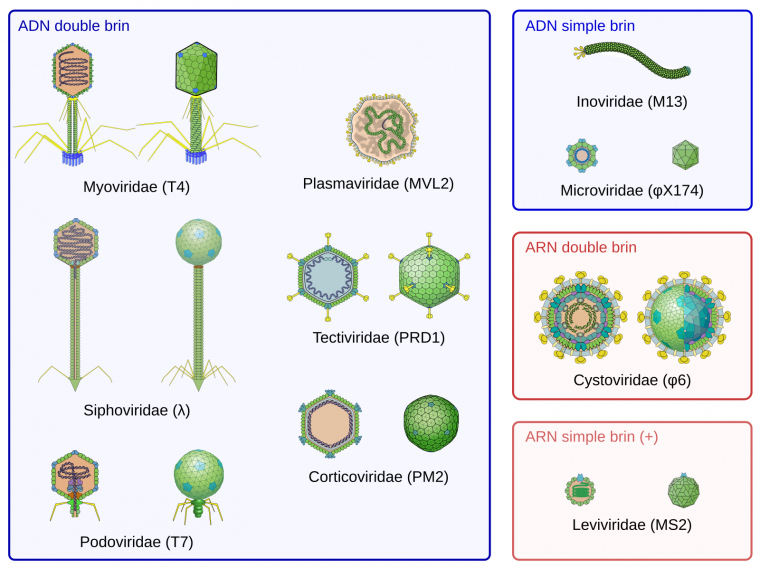
Les bactériophages ont une taille comprise entre 20 et 200 nm (la taille d’un virus est généralement comprise entre 10 et 400 nm). Leurs descriptions ont permis de mettre en évidence une importante diversité de virus classés selon leur morphologie (morphotype), leur composition (notamment la nature de l’acide nucléique qui peut être de l’ADN ou de l’ARN, simple brin ou double brin) et leur spectre d’hôte.
La classification des virus est établie par l’ICTV, International Committee on Taxonomy of Viruses. Pour chaque famille, à l’exception des Plasmaviridae et des Inoviridae, un membre (dont le nom est noté entre parenthèses) est représenté en coupe (à gauche) et en vue externe (à droite). Les Myoviridae, Podoviridae et Siphoviridae sont trois familles de l’ordre des Caudovirales. Cette liste des familles de bactériophages est non exhaustive.
Les schémas de virions proviennent de Philippe Le Mercier - ViralZone SIB Swiss Institute of Bioinformatics.
Environ 95 % des bactériophages actuellement décrits à partir d’observations en microscopie électronique appartiennent à l’ordre des Caudovirales. L’un des plus étudiés est le bactériophage T4 qui infecte la bactérie Escherichia coli. Il sera donc pris ici comme référence.
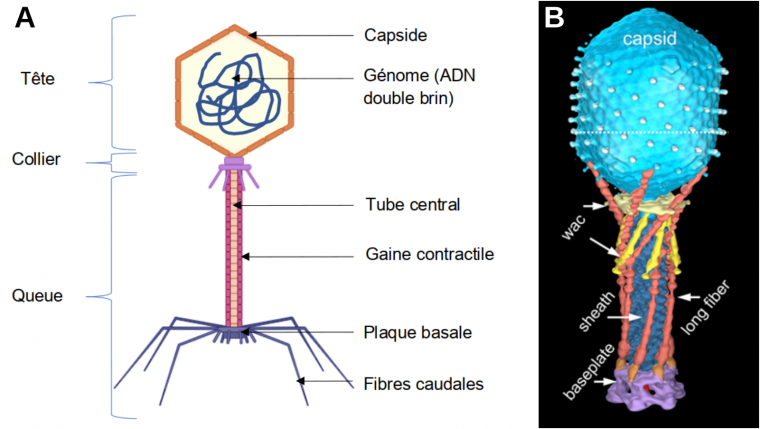
A. Schéma d’un virion de bactériophage T4. La plaque basale porte à la fois des fibres caudales longues et courtes. B. Reconstitution 3D d’un virion fondée sur des résultats de cryo-microscopie électronique. Cette image présente l’état d’une particule virale avant infection. La plupart des longues fibres caudales sont alors repliées contre la gaine.
Crédits : A. Marie Gontier, à partir de l'outil Biorender (qui autorise la reproduction dans un contexte éducatif). B : Hu et coll., 2015, PNAS (cet éditeur autorise la reproduction des figures dans un cadre éducatif et non commercial).
Le bactériophage T4 est constitué de deux parties : une tête et une queue reliées par un collier. La tête comprend une capside composée de protéines, elle renferme un acide nucléique (de l’ADN double brin). La queue, composée également de protéines, comprend un tube central creux entouré d’une gaine contractile. La lumière du tube est en connexion avec celle de la capside permettant le passage du génome du bactériophage vers la cellule hôte lors de l’infection. La queue se termine par une plaque basale à laquelle sont reliées des fibres caudales (ou fibres de la queue) responsables de la reconnaissance de la cellule hôte. On distingue des fibres longues et des fibres courtes. En absence de cellule hôte, seule une partie des fibres longues est étendue, le reste est rétracté et sera déployé lors de la rencontre avec la bactérie cible. En effet, comme tout virus, la propagation d’un bactériophage nécessite de parasiter une cellule cible et d’utiliser à son profit l’ensemble des molécules disponibles.
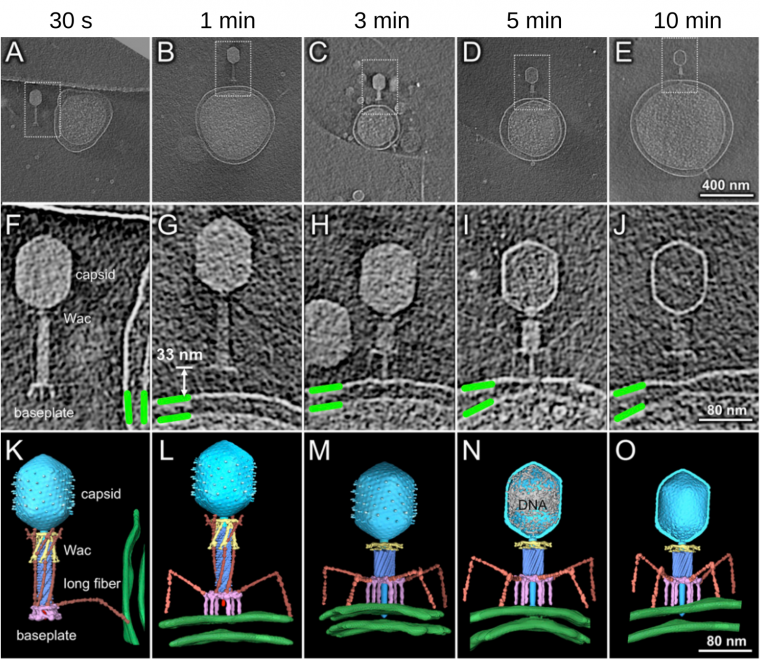
Différentes étapes de l’initiation de l’infection sont représentées de gauche à droite. Les images A à E montrent l’interaction entre un bactériophage et une cellule hôte (E. coli). Les parties encadrées des images A à E sont agrandies dans les images F à J et représentées en 3D sur les images K à O. Les membranes externes et internes de la bactérie hôte (E. coli) sont en vert. La plaque basale (rose, baseplate) passe d’une conformation hexagonale (A, F et K) à une conformation en étoile (B-E, G-J et L-O) par extension des fibres caudales courtes. La capside est en cyan, la gaine en bleu, le collier et ses moustaches (Wac, whisker antigen control) en jaune et les fibres caudales longues en orange. Sur la figure N, l’ADN présent dans la capside est en gris.
Propagation des bactériophages
Reconnaissance et pénétration de la cellule hôte
La rencontre entre un bactériophage et sa cellule hôte s’effectue de manière aléatoire et elle est donc favorisée par un rapport [bactériophages]/[bactéries] élevé.
La reconnaissance de la cellule cible est permise par la présence de ligands situés à la surface de la bactérie dont la nature varie selon le couple bactériophage / bactérie (lipopolysaccharides, protéines de la paroi, acides lipotéichoïques, flagelles, pili…). Il détermine le spectre d’hôte. Ainsi, chaque bactériophage est spécialisé dans l’infection d’un nombre limité d'espèces bactériennes. En revanche, une bactérie peut être reconnue par plusieurs types de bactériophages.
Prenons l’exemple du bactériophage T4 décrit précédemment, spécialisé dans l’infection d’E. coli. La fixation du bactériophage ou adsorption, commence avec le contact de quelques fibres de la queue avec leurs ligands spécifiques : des lipopolysaccharides ou des porines (OmpC) situés sur la membrane externe de la bactérie. La fixation déclenche une modification de la conformation de la plaque basale libérant les fibres courtes qui, à leur tour, adhèrent à leur ligand spécifique, une région particulière des lipopolysaccharides. Ces modifications conduisent à la contraction de la gaine puis à l’expulsion du tube de la queue dans le périplasme c’est-à-dire dans l’espace compris entre la membrane externe et la membrane plasmique. Cette dernière fusionne ensuite avec le tube central creux. Un canal se forme permettant le passage de l’ADN vers la cellule hôte.
Une fois le matériel génétique à l’intérieur de la cellule hôte, la relation entre le parasite et son hôte peut s’établir.
Développement de la relation hôte – parasite
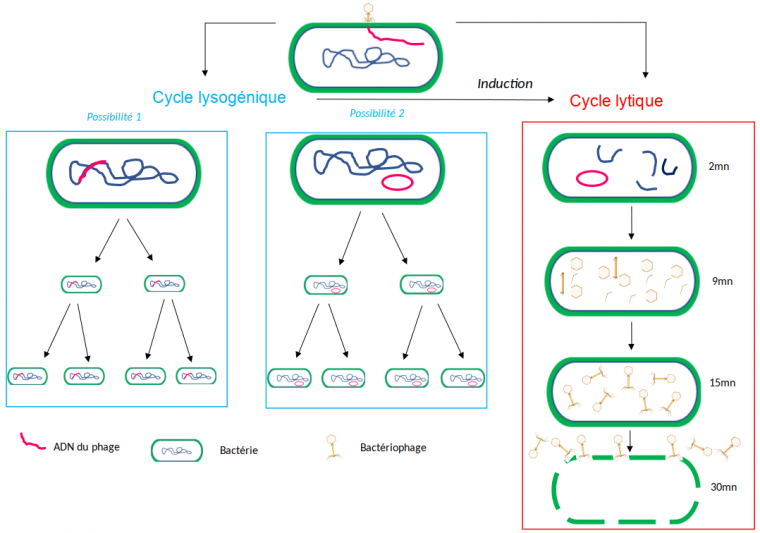
On distingue majoritairement deux grandes stratégies d’infection par les bactériophages :
- Soit l’infection de la bactérie conduit rapidement à sa destruction (lyse). Dans ce cas, le cycle est qualifié de « lytique » et le bactériophage de « virulent ». Les bactériophages T-pairs, comme les T2 ou T4, constituent des exemples de bactériophages lytiques.
- Soit l’infection ne conduit pas à la destruction rapide de la cellule hôte. Le génome du bactériophage s’insère durablement dans le chromosome bactérien sous forme de prophage, ou bien reste sous la forme d’un plasmide. Dans les deux cas, la bactérie n’est pas détruite et transmet ce matériel génétique supplémentaire à ses descendantes Le cycle est qualifié de « lysogénique » et le bactériophage de « tempéré » (c'est le cas, par exemple, du bactériophage λ). Sous certaines conditions physiologiques (stress), le prophage peut s’exciser et reprendre un cycle lytique aboutissant à la production et à la libération de nouvelles particules virales, ou virions, prêts à infecter d’autres bactéries.
À cela s’ajoute une troisième stratégie décrite pour les bactériophages filamenteux (par exemple le bactériophage M13), au cours de laquelle l’infection conduit à la sécrétion continue de virions sans provoquer la destruction de la bactérie. Dans cette partie, seules les deux premières stratégies seront détaillées.
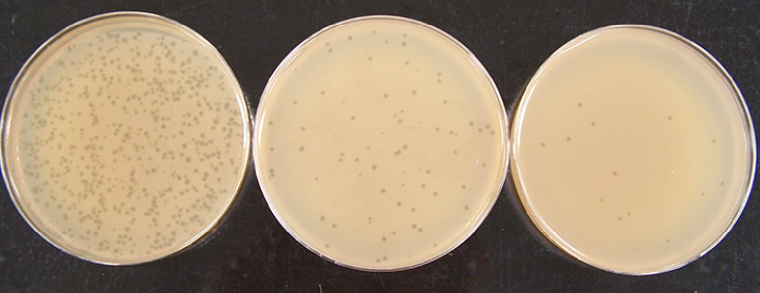
Max Delbrück (1906-1981) et Emory Ellis (1906-2003), qui travaillaient tous deux sur les bactériophages spécialisés dans l’infection d’E. coli ont caractérisé le cycle lytique du bactériophage T21. En 1939, ils mirent au point une expérience dite du cycle unique qui permit d’identifier les évènements moléculaires qui se déroulent pendant la multiplication des bactériophages. L’utilisation de la méthode des plages de lyse (Figure 6), mise au point par F. d’Hérelle, leur a permis de connaître l’évolution du nombre de bactériophages au cours du temps. Les résultats obtenus apparaissent ci-dessous (Figure 7).
Ces boîtes de Petri contiennent des bactériophages T4 mis en présence d’un tapis bactérien d’E. coli. Les plages de lyse, qui se présentent sous la forme de petits ronds, correspondent aux endroits où les bactéries ont été lysées. La concentration en bactériophages mis au contact du tapis bactérien décroît de gauche à droite. D’après un travail de l’American Society for Microbiology.
D’après Prescott et coll., Microbiologie, De Boeck Supérieur.
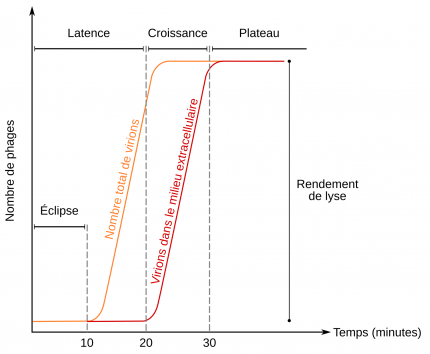
Trois phases ont été mises en évidence :
- une phase de latence ;
- une phase de croissance ;
- une phase de plateau.
Phase de latence
Au début de la phase de latence, seul l’ADN viral se trouve dans la cellule et le bactériophage, en tant que particule virale est indétectable, il n’est présent ni dehors ni à l’intérieur de la bactérie. C’est la phase d’éclipse.
Au laboratoire, dans les deux premières minutes qui suivent le début de l’infection, l’ARNm du bactériophage est synthétisé à partir de l’ARN polymérase de la cellule hôte. Cet ARNm code, entre autres, la production d’exonucléases, des enzymes qui dégradent l’ADN de l'hôte et libèrent ainsi des nucléotides. L’expression des gènes de la cellule hôte est donc stoppée.
Cinq minutes après la contamination, l’ADN viral est synthétisé à partir des nucléotides libérés précédemment. Certains sont modifiés, protégeant alors l’ADN viral des endonucléases (enzymes de restriction) de la bactérie. Par exemple, dans l’ADN du bactériophage T4 la cytosine est transformée en hydroxyméthylcytosine puis glycosylée.
Neuf minutes après l’injection du matériel génétique, l’assemblage des particules virales peut débuter. Des gènes tardifs sont exprimés. Ils codent trois catégories de protéines : des protéines de structure du virion, d’autres qui aident à l’assemblage des nouveaux virions et une dernière catégorie impliquée dans la lyse cellulaire et la libération des bactériophages.
Les protéines de structure s’assemblent spontanément pour former les différents composants du virion. Dans la bactérie, apparaissent alors têtes, queues et fibres caudales. L’assemblage de nouveaux virions s’effectue ensuite selon un ordre précis : il débute par la formation de la plaque basale, se poursuit par l’accrochage du tube de la queue et, enfin, de la gaine autour du tube. La tête finit par se combiner avec la queue tandis que les fibres caudales se fixent à la plaque basale.
Une protéine particulière située à la base de la capside permet l’empaquetage du matériel génétique dans la capside. Par exemple, pour le bactériophage T4, environ 500 µm d’ADN sont empaquetés dans la capside qui mesure moins de 0,1 µm de diamètre.
Après quinze minutes, les premières particules virales assemblées apparaissent dans la bactérie E. coli.
Phase de croissance
Après 22 minutes, les premiers bactériophages sortent de la cellule. On appelle rendement de la lyse le nombre de virus produits par une cellule infectée. Ce dernier dépend des souches de bactériophages. Par exemple, pour le bactériophage T4, il est d’environ 300 virions par cellule d’E. coli. Plusieurs gènes viraux dirigent le processus de lyse. L’un d’entre eux conduit à la production de la holine, une protéine qui assure la perforation de la membrane plasmique, un autre code l’endolysine, une protéine qui détruit le peptidoglycane entourant la bactérie. L’action combinée de ces deux protéines provoque une entrée d’eau massive dans la cellule bactérienne aboutissant à son éclatement et à la libération des virions.
Phase de plateau
Après 30 minutes, plus aucun virus n’est libéré car toutes les bactéries ont été lysées.
Dans les conditions optimales de croissance bactérienne au laboratoire, il faut donc au maximum 30 minutes au bactériophage T4 pour se multiplier. En comparaison, la multiplication des virus d’animaux prend généralement plusieurs heures. La reproduction des bactériophages est donc extrêmement rapide et constitue un avantage pour eux mais également pour les scientifiques qui les utilisent en tant qu’outils biologiques.
Les bactériophages tempérés, quant à eux, pourront basculer dans un cycle lytique et se multiplier activement selon les mêmes étapes que celles décrites précédemment. Ce phénomène de basculement se nomme induction et se manifeste uniquement sous l’influence de facteurs, qualifiés d’inducteurs, souvent liés à un stress : carences nutritives, modification du pH, rayons X, UV, ou encore substances chimiques. Contrairement au cycle lytique des bactériophages virulents, cette stratégie de multiplication décalée dans le temps peut sembler contre-productive et désavantageuse pour les bactériophages. Elle est pourtant bénéfique car ces bactériophages restent associés aux bactéries dans des situations où les bactériophages lytiques ne seraient pas productifs, comme lorsque les bactéries entrent en phase de latence. Les bactériophages tempérés peuvent ainsi attendre que les conditions de multiplication soient plus propices. De même, en cas d’infections par de multiples bactériophages, certaines bactéries contenant un ou des prophages, seront plus à même de survivre car ceux-ci empêcheront le phénomène de surinfection. Les bactéries survivantes permettront de perpétuer le matériel génétique viral.
Réalisé avec l'outil Biorender (qui autorise la reproduction des figures réalisées avec dans un contexte éducatif).
De par leur diversité, leur ubiquité et leurs modes de propagation, les bactériophages ont un impact majeur sur la dynamique et la diversification du monde microbien.
Rôles des bactériophages dans l’environnement
Bactériophages et dynamique des populations bactériennes
Les bactériophages lytiques assurent le renouvellement des populations bactériennes et jouent ainsi un rôle conséquent dans le cycle de la matière à l’échelle de notre planète. De plus, les bactériophages peuvent influencer le développement de maladies humaines. Par exemple certaines régions du monde sont soumises à des épidémies récurrentes de choléra. Leur déclenchement est lié à la multiplication du bacille cholérique dans l’eau qui, une fois ingéré par l’être humain, libère une toxine qui provoque des diarrhées importantes aboutissant à la déshydratation des individus, aussi bien chez les adultes que chez les enfants. Cette toxine est portée par un bactériophage tempéré, le bactériophage CTX. Autrement dit, tous les bacilles cholériques ne sont pas pathogènes. Ils le deviennent suite à l'infection par le bactériophage CTX.
Par ailleurs, dans l’environnement, d'autres bactériophages, quant à eux virulents, peuvent infecter ces souches pathogènes du bacille cholérique. Il en résulte une interaction complexe qui influence les épidémies comme l’a montré Shah Faruque lors d’une étude longitudinale des populations de bacille cholérique et de bactériophages dans les eaux du delta du Gange.
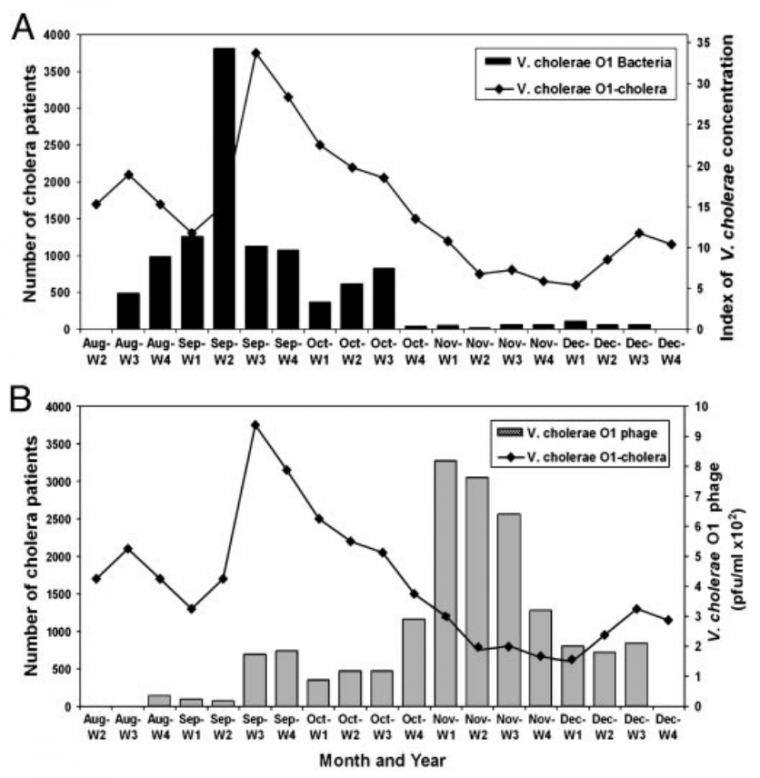
Son étude fut réalisée au Bangladesh et révéla que chaque augmentation de la population bactérienne dans l’eau était suivie d’une multiplication des bactériophages virulents spécialisés dans l’infection de cette bactérie. Ces variations, typiques de celles observées dans les relations de type proies-prédateurs, sont à l’origine des cycles épidémiques observés (Figure 9).
Les deux graphiques indiquent le nombre de cas de choléra recensés à Dhaka, au Bangladesh, entre août et décembre 2004 (courbe). Ce nombre de cas est à mettre en relation avec la concentration de bactéries Vibrio cholerae O1 échantillonnée dans les eaux de surface de la région de Dhaka (A) et la concentration d’un bactériophage lytique spécifique de cette bactérie, appelé JSF4 (B).
Ces interactions sont également à l’origine d’une diversification des bactéries et des bactériophages.
Bactériophages et diversification du monde microbien
Lorsqu’un bactériophage tempéré établit une relation durable avec une bactérie il apporte dans son génome un certain nombre de fonctions qui peuvent affecter la bactérie. Ce processus de « conversion lysogénique » a déjà été mentionné avec le cas de la toxine cholérique portée par le bactériophage tempéré CTX. De manière similaire, la toxine diphtérique est portée par le bactériophage tempéré β acquis par la bactérie Corynebacterium diphteriae. La production de cette toxine augmente le pouvoir infectieux de la bactérie et donc sa capacité de multiplication, ainsi que celle du bactériophage, dans les organismes infectés.
Au cours de l’évolution plusieurs échanges entre les matériels génétiques des bactériophages et des bactéries ont pris place et ceux procurant un avantage sélectif ont pu être conservés. C’est le cas de certains bactériophages infectant les cyanobactéries. Ces virus ont acquis de nombreux gènes bactériens codant notamment des éléments clés du système photosynthétique des bactéries. Lors d’une infection, alors que le matériel génétique de la cyanobactérie n’est plus fonctionnel suite à sa dégradation par les enzymes virales, les gènes viraux codant ces molécules sont exprimés et assurent le maintien du métabolisme photosynthétique chez la cyanobactérie. Le bactériophage peut donc profiter pleinement des ressources disponibles pour se multiplier activement jusqu’à lyse de la cellule hôte.
Aussi précis que le processus d’infection d’un bactériophage puisse être, il demeure soumis à de rares variations pouvant avoir des conséquences directes sur les bactéries. Par exemple, lorsque le matériel génétique du bactériophage est empaqueté dans la capside, il est possible que des gènes bactériens y soient aussi ajoutés. Ils pourront alors être transmis à une autre bactérie lors d’une nouvelle infection par ce bactériophage. Ce phénomène que l’on dénomme transduction est à l’origine de nombreuses modifications des propriétés bactériennes : métabolisme, résistance aux antibiotiques ou aux infections virales. On parle alors de transfert horizontal de gènes entre les bactéries. Un tel transfert peut aussi se produire lorsqu’un plasmide passe d’une bactérie à une autre.
La transduction
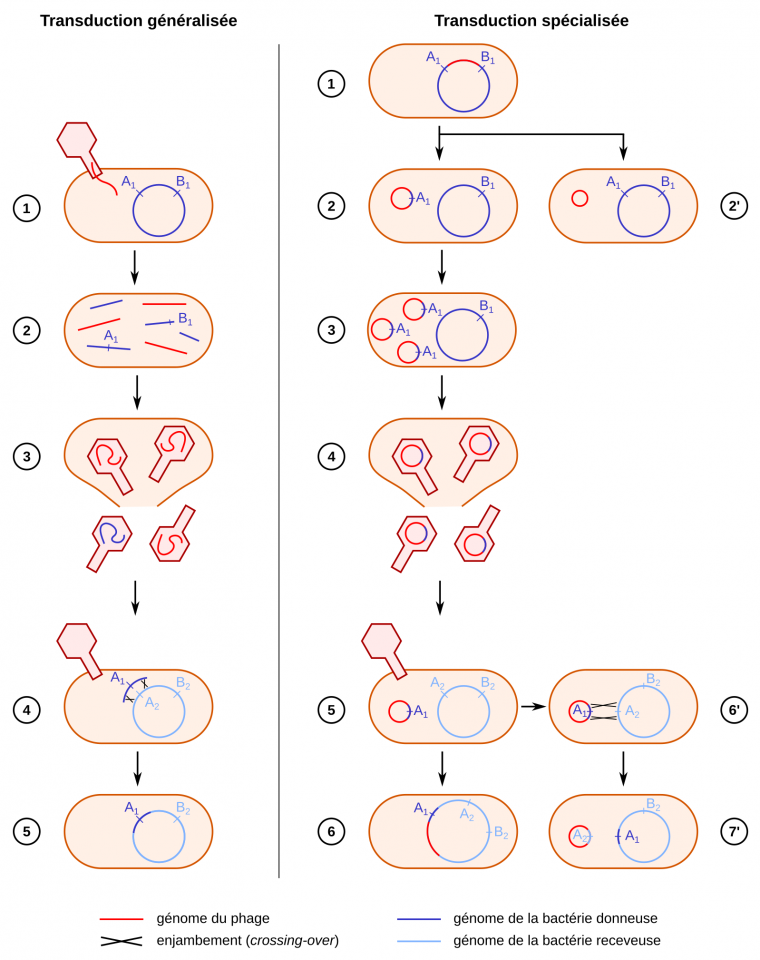
La transduction correspond au transfert de gènes bactériens par l’intermédiaire d’un virus. Au cours du cycle viral, les gènes bactériens se retrouvent enfermés dans la capside et seront transmis à une nouvelle cible bactérienne lors d’une infection ultérieure lui conférant de nouvelles caractéristiques. La transduction généralisée est à distinguer de la transduction spécialisée.
Le mécanisme de la transduction généralisée a été découvert en 1951 par Joshua Lederberg et Norton Zinder. Il est lié au cycle lytique et à la dégradation partielle du matériel génétique de la bactérie. Lorsque l’ADN viral est encapsidé, des fragments de n’importe quelle partie du génome bactérien pourront être incorporés, par erreur, dans la capside. Comme la quantité de matériel génétique incorporé est limitée, certains virions ne contiendront que de l’ADN bactérien, qui, une fois injecté dans une nouvelle bactérie peut, par recombinaison homologue, s’intégrer au chromosome bactérien.
La transduction spécialisée quant à elle se produit durant un cycle lysogène. Suite à l’induction, le matériel génétique viral est extrait du chromosome bactérien. Une excision imprécise peut emporter du matériel génétique bactérien voisin du site d’insertion du génome du bactériophage. L’ensemble sera ensuite injecté à une nouvelle cellule hôte. Lorsque le matériel génétique viral est incomplet et si, par exemple, les gènes nécessaires à l’excision sont absents, le prophage sera alors définitivement intégré au chromosome bactérien. De nombreuses bactéries renferment ainsi des prophages dits cryptiques.
Transduction généralisée. 1. Le bactériophage infecte une bactérie et y transfère son matériel génétique. 2. Le génome du bactériophage se réplique et l’ADN de l’hôte est hydrolysé en fragments de taille variable. 3. Lors de l’assemblage des nouveaux virions, il peut arriver que des morceaux d’ADN de la bactérie hôte soient encapsidés. 4. Lorsqu’un tel bactériophage infecte une autre bactérie, il peut y avoir, par recombinaison homologue, échange entre un fragment de l’ADN de la bactérie donneuse et la zone homologue dans le génome de la bactérie receveuse. 5. La bactérie receveuse peut ainsi présenter un génotype différent de son génotype initial.
Transduction spécialisée. 1. La bactérie donneuse contient de l’ADN d’un bactériophage lysogène incorporé sous forme de prophage. 2’. La plupart du temps, suite à l’induction, le prophage s’excise sans emporter d’ADN bactérien. 2. Cependant, il peut arriver que le prophage s’excise en emportant un fragment d’ADN bactérien adjacent. 3. Le génome viral, et l’ADN bactérien associé, sont répliqués. 4. La cellule hôte est lysée. 5. Une nouvelle bactérie est infectée. 6. Le génome du bactériophage lysogène, ainsi que le fragment d’ADN issu de la bactérie donneuse, s’intègre dans le génome de l’hôte. Le gène A est alors présent en deux exemplaires. 6’ et 7’. Alternativement, un évènement de recombinaison homologue peut intégrer l’ADN de la bactérie donneuse dans celui de la bactérie receveuse.
Enfin, par leurs interactions de type parasite-hôte, les bactéries et les bactériophages exercent entre eux des pressions de sélection menant à une coévolution de ces deux partenaires. Chez les bactéries, des mutations au niveau de gènes codant les récepteurs de surface peuvent leur permettre de devenir résistantes aux bactériophages (voir également l’encadré sur le système CRISPR/Cas). Les bactériophages, quant à eux, peuvent acquérir des mécanismes d’échappement face aux défenses de leurs hôtes [1]. Par exemple, la présence de nucléotides particuliers dans le génome viral peut empêcher la dégradation de celui-ci par les enzymes de restriction bactériennes (comme cela a déjà été évoqué pour le phage T4, où la cytosine est transformée en hydroxyméthylcytosine puis glycosylée).
Alors que cette course à l’armement est largement observée en laboratoire et dans la nature, elle ne semble pas être prédominante dans le microbiote intestinal de l’être humain. Des expériences ont montré que certaines bactéries du microbiote intestinal ne développent pas de résistance vis-à-vis des bactériophages et qu’elles y restent sensibles. Cela pourrait être lié à leur répartition spatiale : des études ont révélé que les bactéries sont plus abondantes dans le mucus externe de l’intestin et les bactériophages, plus abondants dans le lumen. À mesure que le mucus se renouvelle, les bactéries se retrouvent dans le lumen et deviennent des hôtes potentiels pour les bactériophages qui les utilisent pour se multiplier. Ensemble, ces entités biologiques suivent une dynamique qualifiée de « source – puits » qui pourrait être à l’origine de l’équilibre régnant au cours du temps entre les communautés microbiennes.
Le système CRISPR/Cas
Certaines bactéries sont dotées d’un système de défense contre les bactériophages nommé CRISPR/Cas (CRISPR pour Clustered Regularly Interspaced Short Palindromic Repeats et Cas pour CRISPR ASsociated). Le locus CRISPR produit de petits ARN qui, associés à une protéine Cas, sont capables de reconnaître et de dégrader des molécules d’ARN virales présentant une séquence complémentaire à ces petits ARN. À chaque nouvelle rencontre avec un bactériophage, des fragments d’ADN viral sont intégrés au locus CRISPR, augmentant ainsi la taille du répertoire de virus contre lesquels la bactérie peut réagir. Le système CRISPR/Cas constitue donc une forme de mémoire immunitaire permettant aux bactéries de neutraliser des bactériophages déjà rencontrés par elles ou leurs ancêtres (cette mémoire immunitaire étant héréditaire).
Dès leur découverte, les bactériophages ont attisé la curiosité. Ils ont d’abord été utilisés en tant qu’agents thérapeutiques puis, de par l’apparente simplicité de leur matériel génétique, leur diversité et la rapidité de leur cycle biologique, ils sont devenus des modèles biologiques très utilisés en laboratoire. Aujourd’hui face à l’augmentation des cas d’antibiorésistance, leur potentiel thérapeutique est de nouveau à l’étude.
Utilisations des bactériophages par l’Homme
La phagothérapie : les bactériophages utilisés en tant qu’agents thérapeutiques
Après avoir découvert en 1917 l’action bactéricide des bactériophages sur le bacille dysentérique, Félix d’Hérelle eut l’idée de les utiliser en tant qu’agents thérapeutiques pour soigner des enfants atteints de dysenterie. Le résultat fut un succès et, peu à peu, le traitement curatif à base de bactériophages se répandit dans le monde entier. Les bactériophages ont pu contribuer à limiter le développement de certaines maladies comme la peste en Égypte ou encore le choléra en Inde. Mais très vite, face au manque de connaissances sur la nature et le fonctionnement des bactériophages, cette pratique fut l’objet de controverses et soumise à de vives critiques. En 1928, la découverte de la pénicilline par Alexander Fleming puis celles d’autres antibiotiques annonça le déclin de la phagothérapie. Ces derniers, beaucoup plus simples à fabriquer, ont rapidement été produits en masse par les industries pharmaceutiques puis commercialisés. Peu à peu, dans les pays occidentaux, la phagothérapie tomba dans l’oubli et fut remplacée par l’antibiothérapie. Les bactériophages furent alors davantage utilisés à des fins de recherche fondamentale où ils participèrent d’ailleurs à l’essor de la biologie moléculaire. Dans certains pays, en revanche, la pratique se poursuivit. On y trouve d’ailleurs aujourd’hui des instituts spécialisés dans la recherche sur les bactériophages dont les plus renommés sont l’Institut Eliava situé à Tbilissi (Géorgie), créé en 1923, ou l’institut Hirszfeld à Wroclaw (Pologne), créé en 1961.
L’essor et le mésusage des antibiotiques a conduit au développement des cas d’antibiorésistance au point de devenir aujourd’hui l’une des menaces les plus importantes pour la santé publique mondiale. Ce phénomène ne concerne pas que les êtres humains mais plus largement, les animaux et autres formes de vie dans notre environnement. Si cette évolution se poursuit, il se pourrait qu’en 2050 les maladies infectieuses deviennent l’une des premières causes de mortalité humaine dans le monde. Face à l’urgence, des chercheurs se penchent actuellement de nouveau sur l’utilisation des bactériophages. En effet, contrairement aux antibiotiques, les bactériophages possèdent un spectre d’action très étroit permettant de cibler plus précisément des bactéries pathogènes. Leur utilisation, généralement sous forme de cocktail de bactériophages (association de plusieurs bactériophages) permettrait de combattre les bactéries infectieuses de manière plus ciblée et de pallier l’apparition de résistance. Enfin, les bactériophages ne s’attaquant pas aux cellules eucaryotes, ils sont éliminés une fois leur cible éradiquée. Actuellement, des start-ups apparaissent dans le monde entier. En France, Pherecydes Pharma, créée en 2006, a entamé plusieurs essais cliniques en vue d’utiliser des cocktails de bactériophages pour combattre des souches pathogènes : l’étude PhagoBurn (2013) pour combattre la bactérie pathogène Pseudomonas aeruginosa responsable d’infections cutanées chez les grands brûlés ; l’étude PHOSA (2014) en vue de lutter contre des infections ostéoarticulaires causées par Staphylococcus aureus et S. epidermidis ; l’étude PneumoPhage (2015) pour traiter les infections respiratoires aiguës à P. aeruginosa.
La généralisation de cette pratique nécessite cependant encore de franchir de nombreux obstacles notamment en ce qui concerne le procédé de fabrication. Ce dernier doit être encadré car actuellement, même si les bactériophages semblent répondre à la définition d’un médicament et plus précisément d’un biomédicament, aucun guide officiel de fabrication n’existe. Une autre difficulté concerne le manque de connaissances sur les effets des bactériophages à plus ou moins long terme sur les écosystèmes bactériens tant dans l’organisme que dans notre environnement. Enfin, l’utilisation des bactériophages nécessite un traitement personnalisé dont la mise en place peut être assez longue. Face à ces obstacles, l’utilisation des bactériophages en France reste rare et alimente un tourisme médical vers des instituts étrangers où ces traitements peuvent être dispensés. Jusqu’à aujourd’hui, en France, seuls quelques patients se trouvant dans une impasse thérapeutique ont pu bénéficier d’un traitement à base de bactériophages importés ou faisant l’objet d’essais cliniques et ayant bénéficié à titre exceptionnel d’une ATU (autorisation temporaire d’utilisation). Cette dernière est délivrée par l’Agence nationale de sécurité du médicament et des produits de santé (ANSM) dans le cadre d’une procédure compassionnelle.
Qu’est-ce qu’un médicament ?
« Un produit est qualifié de médicament s’il est présenté comme possédant des propriétés curatives ou préventives à l’égard des maladies humaines ou animales, ou s’il peut être utilisé ou administré en vue d’établir un diagnostic médical. Dans le développement d’un médicament, l'ANSM intervient dès la phase des essais cliniques. Elle autorise ensuite, au niveau national, sa mise sur le marché (AMM) en fonction de l’évaluation de son bénéfice et de ses risques. L’ANSM poursuit son action en assurant la surveillance de sa sécurité d’emploi. C’est elle également qui contrôle la publicité auprès du public et des professionnels de santé. »
« Un médicament biologique est un médicament dont la substance active est produite à partir d’une source biologique ou en est extraite et dont la caractérisation et la détermination de la qualité nécessitent une combinaison d’essais physiques, chimiques et biologiques ainsi que la connaissance de son procédé de fabrication et de son contrôle. » [2]
Recherche fondamentale, exemple du phage display
Les bactériophages peuvent être utilisés en tant que vecteurs de clonage. Un des modèles les plus utilisés est le bactériophage M13, appartenant à la catégorie des bactériophages filamenteux. Ces derniers ont une forme allongée (d’où leur appellation), leur matériel génétique se trouve au centre d’un tube composé de protéines de structure qui s’organisent en hélice. Ils possèdent un cycle de multiplication particulier puisque l’infection d’une cellule hôte (E. coli dans le cas du phage M13) conduit à une production continue de nouveaux virions sans détruire la bactérie. Lors d’une infection, la capside fusionne avec la membrane de la cellule hôte et les protéines qui la composent s’y insèrent. Lors de la fabrication de nouveaux virions, de nouvelles protéines de structure seront fabriquées puis insérées dans la membrane avant de se retrouver dans les nouveaux virions.
En 1985, George Smith mit au point une technique nommée phage display (ou exposition sur bactériophage en français) qui permet d’exprimer une protéine étrangère d’intérêt à la surface du bacteriophage M13. Par la suite, cette technique a été de plus en plus utilisée et trouve aujourd’hui de nombreuses applications dans le domaine médical où elle permet, par exemple, d’identifier des anticorps monoclonaux qui interviennent dans des stratégies thérapeutiques ou prophylaxiques.
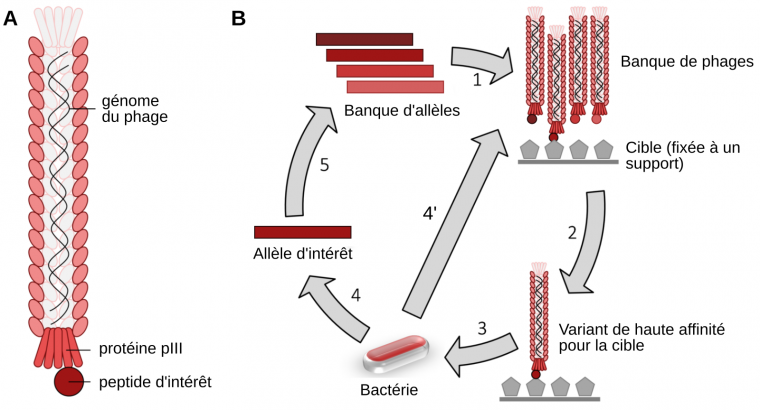
Le principe du phage display consiste à fusionner le gène codant la protéine PIII, responsable de l’adsorption du bactériophage, avec un gène d'intérêt. Le bactériophage est ensuite placé au contact de la cellule hôte dont l’infection conduit à la production de nombreux clones. Parmi ces derniers, certains exprimeront à leur surface la protéine d'intérêt associée aux protéines PIII. L’utilisation d’une cible (souvent un anticorps monoclonal) permet ensuite d’identifier les bactériophages porteurs de la protéine d’intérêt (c’est-à-dire capable de se lier à la cible) parmi les clones produits. Après sélection, ces derniers seront de nouveau amplifiés puis sélectionnés (Figure 12).
A. Le bactériophage M13 est un bactériophage filamenteux. En modifiant son génome par génie génétique, il est possible de produire une protéine de fusion associant la protéine de capside pIII à un peptide d’intérêt.
B. Une banque d’allèles d’intérêt est utilisée pour produire une banque de bactériophages (1). Ceux-ci sont mis en présence de la cible d’intérêt. Une étape de lavage (2) permet de ne retenir que les variants ayant une bonne affinité pour la cible. Ces bactériophages sont ensuite utilisés pour infecter des bactéries. Les bactériophages produits peuvent ensuite être directement utilisés pour recommencer une étape de sélection des variants ayant la meilleure affinité (4’). Les séquences d’ADN des meilleurs variants peuvent également être isolées et manipulées par génie génétique (4 et 5).
Au fur et à mesure, la méthode se perfectionna. Au lieu d’utiliser des gènes issus d’une banque génomique, des séquences furent produites de manière aléatoire de façon à générer de nombreux peptides parmi lesquels certains possèdent une forte affinité et spécificité vis-à-vis de protéines cibles.
Dans les années 1990, Gregory Winter et son équipe sont allés encore plus loin. Ils ont montré que la technique de phage display pouvait être utilisée pour créer des anticorps humains hautement spécifiques d’une molécule. Des gènes codant la partie variable des anticorps (regroupés sous le terme VH pour les chaînes lourdes, VL pour les chaînes légères) ont été introduits dans le génome du bactériophage M13 puis exprimés à sa surface. Ces gènes peuvent provenir de lymphocytes B naïfs (n’ayant jamais rencontré d’antigène), de lymphocytes B sélectionnés suite à une rencontre avec un antigène ou d’un réarrangement génétique réalisé in vitro. Leur expression conduit à l’exposition d’une fraction variable d’anticorps à la surface du bactériophage. Une phase de sélection par criblage permet ensuite de repérer les bactériophages portant la molécule d’intérêt, c’est-à-dire l’anticorps capable de reconnaître la cible souhaitée. L’amplification du gène en cause, par la technique de PCR mutagène (mise au point par Frances Arnold) permet ensuite de générer de nombreux variants de ce gène, et de reproduire l’effet des hypermutations somatiques qui se déroulent naturellement dans l’organisme et permettent d’améliorer l’affinité des anticorps pour leur antigène. De nouvelles phases d’amplification et de sélection permettent de sélectionner petit à petit des fragments possédant une forte spécificité vis-à-vis de la molécule ciblée. Par génie génétique, la partie variable extraite est ensuite associée à la partie constante d’un anticorps humain.
Ce procédé a par exemple permis d’obtenir un médicament, l’adalimumab, contenant un anticorps monoclonal capable de reconnaître et neutraliser le facteur de nécrose tumorale α ou TNF-α, une molécule pro-inflammatoire. Il est actuellement prescrit dans le cas de maladies inflammatoires comme le psoriasis, la maladie de Crohn ou encore la polyarthrite rhumatoïde [3]. Récemment, il a aussi permis la fabrication d’anticorps monoclonaux dirigés contre la protéine Spike (ou protéine S) du SARS-CoV-2, indispensable à la fusion entre le virus et ses cellules cibles. La technique du phage display a permis de sélectionner un fragment VH hautement spécifique à la protéine parmi une bibliothèque de fragments. La fusion entre ce dernier et un autre fragment codant la partie constante d’un anticorps humain a permis de produire des anticorps monoclonaux hautement spécifiques.
L’ensemble des techniques mises au point pour aboutir à cette nouvelle génération d’anticorps a valu à Frances Arnold, George Smith et Gregory Winter l’obtention du prix Nobel de chimie en 2018.
Conclusion
Cette synthèse sur les bactériophages, loin d’être exhaustive, nous rappelle à quel point le monde microbien est diversifié. Depuis des millions d’années, ces virus coévoluent avec leurs cellules hôtes, les bactéries. Durant les dernières décennies les bactériophages ont surtout été utilisés en tant qu’outils de biologie moléculaire. Aujourd’hui; de plus en plus d’applications sont envisagées notamment en ce qui concerne la lutte contre le développement des résistances bactériennes. L’avenir des bactériophages semble prometteur, ces virus, aussi discrets qu’ils paraissent, n’ont probablement pas fini de nous étonner.
Références
[1] M.-N. Cordonnier, Le puissant arsenal de guerre des bactériophages, Pourlascience.fr. (consulté le sept. 13, 2021).
[2] A. Dublanchet, La phagothérapie. Des virus pour combattre les infections. Favre, 2017.
[3] M.-N. Cordonnier, Le prix Nobel de chimie 2018 met à l’honneur l’évolution dirigée, Pourlascience.fr. (consulté le sept. 13, 2021).