Les antibiotiques exercent une pression de sélection naturelle sur les populations bactériennes à l’origine de l’émergence de plus en plus fréquente de bactéries résistantes. Ce problème majeur de santé publique est fortement lié au mésusage et à la surconsommation des antibiotiques en santé humaine et animale, accentués par la diffusion des antibiotiques et bactéries résistantes dans l’environnement. La prévention de la résistance doit donc être globale avec des leviers d’action multiples.
Découverte et mécanismes d’action des antibiotiques
Ernest Duchesne, étudiant en thèse à Lyon en 1897, met en évidence un effet antagoniste entre champignons et bactéries lorsqu’ils sont injectés à des cobayes, ceux-ci mourant moins de l’infection bactérienne (1). Plus tard, en 1928, Sir Alexander Fleming, médecin biologiste à Londres, observe une inhibition de la pousse bactérienne d’une culture de staphylocoques en présence d’une culture de Penicillium notatum. Il isole la molécule produite par le champignon et la nomme pénicilline. Plusieurs années de recherche seront nécessaires pour optimiser les conditions de production de la pénicilline et permettre son utilisation à grande échelle pendant la Seconde Guerre mondiale. En 1937, les premiers sulfamides, plus toxiques que la pénicilline, sont également découverts. Ces deux découvertes médicales majeures marquent le début de la découverte de nombreuses familles d’antibiotiques soit naturels (produits par des micro-organismes), soit synthétiques (produits chimiquement, sans l’intervention de micro-organismes), soit semi-synthétiques (antibiotiques naturels modifiés chimiquement afin d’en changer les propriétés) (tableau I).
| Antibiotiques | Classe | Première année d’utilisation en santé humaine |
|---|---|---|
| p-aminophénylsulfamide | Sulfamides | 1937 |
| Pénicilline | β-lactamines | 1943 |
| Streptomycine | Aminosides | 1947 |
| Tétracycline | Cyclines | 1952 |
| Méticilline | β-lactamines | 1960 |
| Acide nalidixique | Quinolones | 1964 |
| Gentamicine | Aminosides | 1967 |
| Vancomycine | Glycopeptides | 1972 |
| Céfotaxime | β-lactamines | 1981 |
| Linézolide | Oxazolidinones | 2000 |
| Daptomycine | Glycolipopeptides | 2003 |
| Ceftolozane-tazobactam | β-lactamines | 2017 |
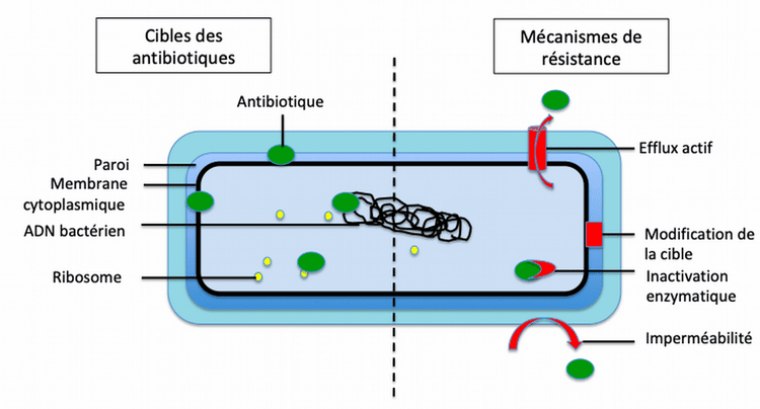
Les antibiotiques perturbent des mécanismes essentiels de la vie cellulaire (réplication, transcription, traduction, synthèse de la paroi…) ce qui limite la croissance bactérienne (effet bactériostatique) ou tue les bactéries (effet bactéricide) (Figure 1). Comme les cibles moléculaires des antibiotiques sont présentes uniquement chez les bactéries, ces substances n’interfèrent pas avec la vie des cellules eucaryotes et n’ont pas d’effet sur les virus.
Les principaux sites d’action des antibiotiques sont la paroi bactérienne, le ribosome, l’ADN bactérien, la membrane cytoplasmique. Les quatre principaux mécanismes de résistance sont l’imperméabilité, l’inactivation enzymatique, la modification de la cible de l’antibiotique et l’efflux actif.
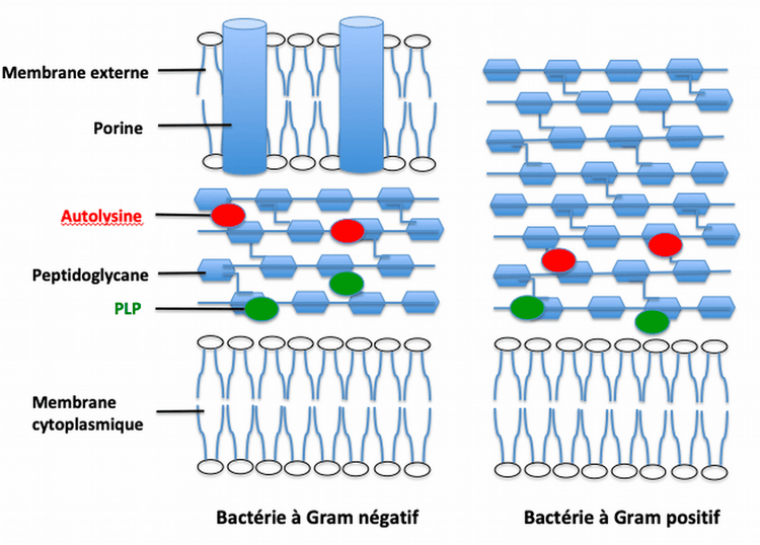
La cible des antibiotiques au niveau de la paroi bactérienne est un constituant indispensable à la bactérie : le peptidoglycane. Celui-ci est en perpétuel remaniement, résultat d’un équilibre dynamique fragile entre activité de synthèse par les PLP (protéines liant la pénicilline) et d’hydrolyse par les autolysines (Figure 2). Les β-lactamines (pénicillines, céphalosporines) forment une famille d’antibiotiques caractérisée par la présence d’un noyau β-lactame. Ces molécules sont capables de se lier aux PLP par liaison covalente et d’inhiber leur activité. L’équilibre entre lyse et synthèse du peptidoglycane est alors rompu, les bactéries deviennent incapables de résister à la pression osmotique exercée sur leur membrane plasmique et meurent par lyse osmotique.
Chez les bactéries à Gram positif, le peptidoglycane est la partie la plus externe de la bactérie. Il est plus épais que chez les bactéries à Gram négatif et entoure la membrane cytoplasmique de la bactérie. Chez les bactéries à Gram négatif, la paroi contient un élément supplémentaire, la membrane externe, laquelle entoure le peptidoglycane, plus fin que chez les bactéries à Gram positif. Le peptidoglycane constitue une structure tridimensionnelle spécifique au monde bactérien. Il est essentiellement composé de chaînes glucidiques reliées par des ponts tétrapeptidiques conférant à la bactérie sa forme, sa rigidité et lui permettant de résister à la forte pression osmotique intracytoplasmique. Il est synthétisé par les PLP (protéines liant la pénicilline correspondant à trois types d’enzymes : transpeptidases, carboxypeptidases et glycosyltransférases) et dégradé par les autolysines.
L’inhibition de la synthèse protéique est également l’un des principaux mécanismes d’action des antibiotiques. Les aminosides (amikacine, gentamicine) se fixent à l’ARN ribosomal 16S (de la sous-unité 30S du ribosome) et altèrent la synthèse protéique. En raison d’erreurs de lecture, des protéines anormales sont intégrées à la membrane cytoplasmique entrainant une perte de l’intégrité membranaire.
Les antibiotiques peuvent également inhiber la réplication de l’ADN. Lors de celle-ci, l’ADN gyrase crée des coupures dans le double brin d’ADN. Ces coupures normalement transitoires, sont suivies d’un recollage des deux brins par la gyrase. L’activité des quinolones (ofloxacine) est expliquée par la formation d’un complexe ternaire irréversible quinolone-ADN-gyrase. Le fonctionnement de l’enzyme est altéré et la coupure de l’ADN n’est pas réparée aboutissant à la mort cellulaire.
Enfin, il existe d’autres antibiotiques, aux mécanismes d’action variés : dépolarisation de la membrane plasmique par les glycolipopeptides cycliques, inhibition du métabolisme des folates par les sulfamides, etc (2).
Supports génétiques et mécanismes de résistance
La résistance d’une bactérie peut être naturelle, elle concerne alors toutes les souches d’un genre ou d’une espèce et détermine le phénotype sauvage de résistance. Elle est alors portée par le chromosome et se transmet verticalement lors de la division cellulaire. La résistance d’une bactérie peut également être acquise. Elle est alors retrouvée dans une proportion plus ou moins importante des souches d’une espèce et est variable dans le temps. Dans ce cas, la transmission est verticale ou horizontale (entre bactéries via des éléments génétiques mobiles).
La fréquence élevée des bactéries résistantes s’explique par la grande plasticité du génome bactérien. Il peut y survenir des mutations chromosomiques (phénomène rare de l’ordre d’une bactérie sur un milliard) ou plus fréquemment l’acquisition d’éléments génétiques mobiles porteurs de gènes de résistance (plasmides, transposons et intégrons) retrouvés à haute fréquence (jusqu’à une bactérie sur 100) (3). Les voies d’acquisition des éléments génétiques mobiles sont de plusieurs types : transformation bactérienne, transduction par bactériophages, conjugaison bactérienne pour le transfert de plasmides. La conjugaison, contrairement à la transduction, n’est pas spécifique d’espèce et est donc très impliquée dans la diffusion de gènes de résistance entre différentes espèces bactériennes notamment au sein des microbiotes1. L’acquisition de plusieurs gènes par une bactérie entraine une résistance à plusieurs classes d’antibiotiques habituellement appelée multirésistance.
Il existe quatre principaux mécanismes de résistance : imperméabilité bactérienne, modification de la cible, inactivation de l’antibiotique, efflux actif (Figure 1).
L’imperméabilité bactérienne est impliquée dans la résistance naturelle des bacilles à Gram négatif aux glycopeptides (vancomycine), molécules de grande taille ne pouvant pas entrer dans les porines de la membrane externe de ces bactéries (Figure 2). L’imperméabilité est également impliquée dans la résistance acquise des bactéries, par exemple celle de Pseudomonas aeruginosa à l’imipénème par perte de la porine D2 de la membrane externe, voie d’entrée de l’antibiotique (4).
Une modification de la cible de l’antibiotique entraine une perte d’activité de celui-ci. Un exemple incontournable est la résistance acquise de Staphylococcus aureus, pathogène humain très répandu, à la méticilline. La bactérie possède une nouvelle PLP, la PLP2A ayant très peu d’affinité pour les β-lactamines. La PLP2a est codée par le gène MecA, inclus dans un élément génétique mobile intégré dans le chromosome et appelé en anglais staphylococcal cassette chromosome mec ou SCCmec.
Un troisième mécanisme d’action très répandu est l’inactivation enzymatique de l’antibiotique. Par exemple, la bactérie peut acquérir des gènes de résistance codant des enzymes nommées β-lactamases et capables d’hydrolyser le noyau β-lactame des β-lactamines, les transformant en produits inactifs. Chez les bacilles à Gram négatif, il existe une grande diversité des β-lactamases impliquées dans des résistances naturelles (TEM, SHV) et acquises (CTX-M, TEM, SHV, KPC, OXA, NDM) aux antibiotiques (2). Quelques années après l’utilisation d’une nouvelle β-lactamine en thérapeutique (amoxicilline, céphalosporines, carbapénèmes), on voit toujours apparaitre la résistance à ces antibiotiques par production de β-lactamases.
Des systèmes de pompes à efflux permettent également d’éliminer l’antibiotique en dehors de la bactérie. Ce mécanisme de résistance est particulièrement impliqué dans les résistances naturelles et acquises de P. aeruginosa aux antibiotiques.
Les antibiotiques exercent inéluctablement une pression de sélection environnementale sur les populations bactériennes. Cette sélection de bactéries résistantes et pré-existantes peut avoir lieu en situation pathologique, au sein d’un foyer infecté (5). Elle peut également se faire en situation physiologique, par exemple au sein du microbiote intestinal, lieu privilégié présentant une densité élevée de bactéries (1014 unités formant des colonies/gramme de selles) et une diversité importante d’espèces, propice aux échanges horizontaux de gènes de résistance (6). Ainsi, la prise d’antibiotique, même de durée courte, a toujours des conséquences sur nos microbiotes ; il ne s’agit pas d’un acte anodin.
CMI et CPM
La concentration minimale inhibitrice ou CMI mesure l’effet bactériostatique d’un antibiotique sur une bactérie. Elle correspond à la plus petite concentration d’antibiotique inhibant la pousse bactérienne visible après 18 h de culture à 35 °C.
La concentration de prévention des mutants résistants ou CPM est une grandeur non mesurable, supérieure à la CMI, en dessous de laquelle la sélection de mutants résistants au sein d’une population bactérienne est possible.
Au-dessus de la CPM, même les bactéries résistantes à l’antibiotique sont tuées par celui-ci. En effet, l’antibiorésistance n’est jamais absolue. Au-delà d’une certaine concentration seuil en antibiotique, les mécanismes permettant la résistance à l’antibiotique sont saturés et même les bactéries dites « résistantes » meurent.
L’activité antibactérienne in vitro d’un antibiotique (détermination d’une CMI) considérée isolément n’est pas prédictive de son efficacité in vivo. En effet, un antibiotique actif in vitro peut être en incapacité à pénétrer de manière satisfaisante dans tel ou tel site infectieux. Seule la prise en considération des aspects bactériologiques (CMI) mais également pharmacocinétiques (concentration sérique de l’antibiotique en fonction du temps, diffusion de l’antibiotique dans les tissus, etc.) regroupés dans l’appellation pK/pD des antibiotiques (pharmacocinétique/pharmacodynamie des antibiotiques) permet de prédire l’efficacité clinique d’un antibiotique.
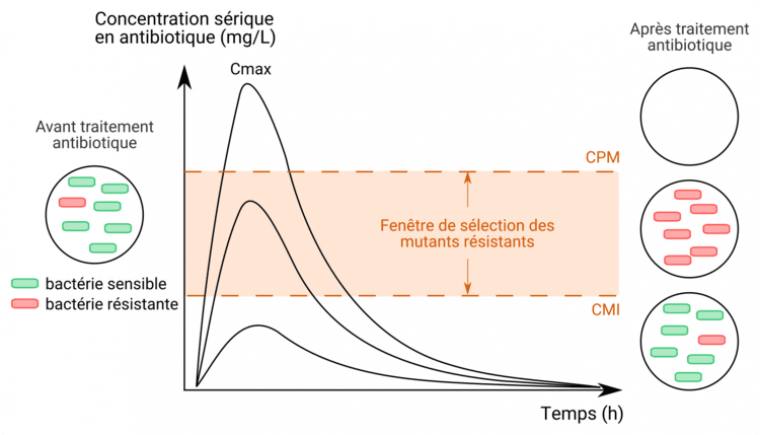
La concentration de prévention des mutants résistants (CPM) est un paramètre pharmacodynamique qui, confronté à la concentration sérique ou du site infecté en antibiotique, permet de prédire la sélection possible d’une sous-population résistante lors d’une infection (Figure 3). Les posologies, le rythme d’administration et la durée de traitement préconisés en thérapeutique reposent sur des études pK/pD et cliniques et visent notamment à éviter la fenêtre de sélection de mutants résistants. Lors d’un traitement antibiotique, leur respect est donc capital (7).
Une population bactérienne comprend de rares mutants résistants préexistants au traitement. Lors de la prise d’un antibiotique, il est possible de suivre l’évolution de la concentration sérique (sanguine) de celui-ci. Dans tous les cas, on observe une augmentation rapide de la concentration sérique en antibiotique suite à la prise de celui-ci, traduisant le passage des molécules du système digestif vers le système circulatoire. Puis, suite à la dégradation de l’antibiotique, notamment par le foie, la concentration sérique diminue.
Si la concentration sérique de l’antibiotique reste inférieure à la CMI, il n’a pas d’effet sur l’ensemble de la population bactérienne. Si la concentration sérique atteint des niveaux entre CMI et CPM, les rares bactéries résistantes (sous-population) présentes pourront persister et se multiplier. Il est donc important d’obtenir une concentration maximale sérique (Cmax) supérieure à la CPM et cela le plus longtemps possible pour éviter la fenêtre de sélection de mutants résistants.
Propagation de la résistance et conséquences
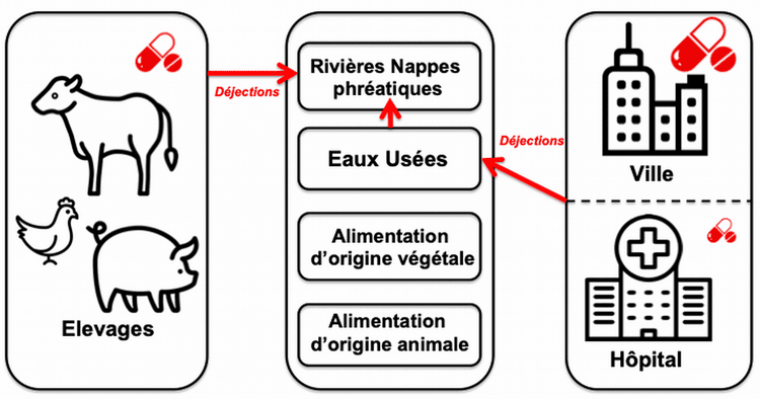
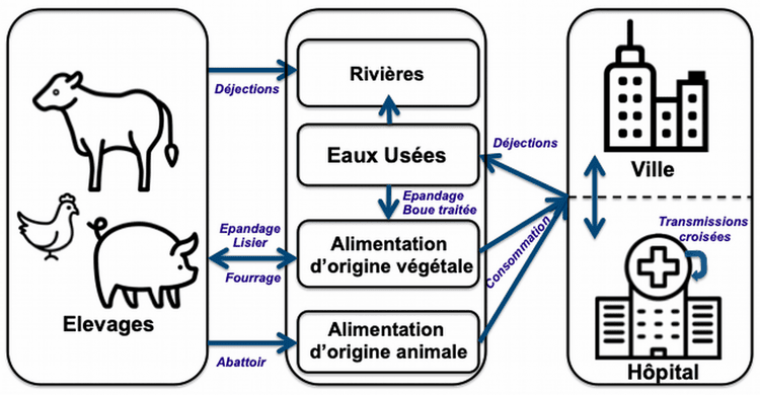
La consommation et la dissémination des antibiotiques, et donc des bactéries résistantes, se fait dans trois secteurs principaux : les élevages, l’environnement et en santé humaine (Figures 4 et 5) (8). Ces constats montrent l’importance de l’approche globale « Une seule santé ? » promulguée par l’Organisation mondiale de la santé dans son plan d’action contre la résistance aux antibiotiques, celle-ci mobilisant des acteurs variés.
Cette figure représente les trois grands secteurs de diffusion des antibiotiques : santé animale à gauche, environnement au milieu et santé humaine à droite. En 2017, la consommation d’antibiotiques est la plus élevée en santé humaine : 759 tonnes versus 514 tonnes en santé animale. En santé humaine, la consommation d’antibiotiques est essentiellement faite en ville (93 % contre 7 % à hôpital). Les antibiotiques ingérés sont éliminés dans l’environnement où certains antibiotiques y sont stables et persistent.
Les bactéries se propagent via l’alimentation et les déjections. L’environnement a un rôle de réservoir et de propagation. Le secteur hospitalier est un secteur propice aux transmissions croisées (d’individu à individu).
La principale cause de l’accélération de l’antibiorésistance est la surconsommation des antibiotiques, principalement en médecine de ville où la consommation a augmenté entre 2007 et 2017. Sur la même période, l’usage d’antibiotique est resté stable dans les hôpitaux et a diminué en santé animale grâce aux plans ÉcoAntibio, avec pour conséquence une baisse de la prévalence des résistances chez les animaux d’élevage (8).
À ce jour, même si des succès ont été observés (par exemple, diminution de la prévalence des pneumocoques de sensibilité diminuée à la pénicilline et de S. aureus résistant à la méticilline), l’inquiétude se concentre sur l’augmentation de la résistance des bacilles à Gram négatif responsables de nombreuses infections en santé humaine telles que des infections urinaires ou digestives.
La résistance aux antibiotiques a d’ores et déjà un impact considérable en santé publique. En France, l’un des pays d’Europe les plus consommateurs d’antibiotiques en ville, on estime que la multirésistance est responsable de 158 000 infections par an causant 12 500 décès. En effet, la résistance aux antibiotiques entraine des difficultés de traitement pour des infections autrefois sans gravité et peut mener parfois à des situations d’impasse thérapeutique (absence d’antibiotiques actifs) (exemple Figure 8 C). De plus, le coût annuel de l’antibiorésistance est estimé à 290 millions d’euros par an en France (9). La situation future pourrait s’aggraver en cas d’accélération de l’émergence de résistances ayant pour conséquence l’utilisation de molécules à spectre large, générant elles-mêmes plus de résistances (cercle vicieux) et en l’absence d’un développement suffisant de nouveaux antibiotiques. La problématique est mondiale, certains pays ayant une prévalence très élevée de bactéries résistantes. Des mesures d’hygiène strictes sont d’ailleurs mises en œuvre dans les établissements de santé français lors du rapatriement de patient hospitalisés préalablement à l’étranger.
Documentation de la résistance aux antibiotiques
La documentation des infections permet de mettre en évidence les bactéries impliquées et d’étudier leur sensibilité aux antibiotiques. Le traitement d’un prélèvement de microbiologie comprend les étapes suivantes : examen direct avec coloration de Gram et ensemencement du prélèvement sur des milieux de culture le premier jour (J0), identification des colonies bactériennes et ensemencement des antibiogrammes le lendemain (J1) et lecture des antibiogrammes à J2.
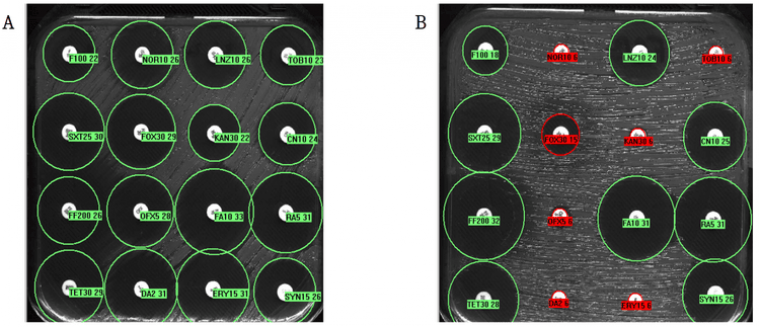
L’antibiogramme est une technique phénotypique permettant de catégoriser sensible (S), intermédiaire (I) ou résistant (R) un couple bactérie-antibiotique. L’antibiogramme par diffusion en milieu gélosé correspond à la croissance d’une bactérie sur un milieu solide comprenant une concentration croissante d’antibiotique (apposition d’un disque d’antibiotique sur le milieu de culture suivie de la diffusion de l’antibiotique dans la gélose). On obtient un diamètre d’inhibition après 16 h de culture à 35 °C (Figure 6). Il existe une corrélation entre CMI et diamètre d’inhibition d’un couple antibiotique-bactérie. L’utilisation de l’antibiotique en thérapeutique est possible pour les souches sensibles à la posologie standard et pour les souches intermédiaires à forte posologie (Figures 7 et 8) (10).
A. S. aureus sensible à l’ensemble des antibiotiques testés. Phénotype sauvage. B. S. aureus résistant à la méticilline (disque FOX) et à d’autres classes d’antibiotiques telles que les fluoroquinolones (NOR et OFX), macrolides (ERY), lincosamides (DA) et les aminosides (KAN et TOB). Les noms des antibiotiques, les cercles délimitant les contours des disques d’inhibition, et les couleurs (vert : sensible, jaune : intermédiaire, rouge : résistant) sont ajoutés automatiquement sur l’image par une machine spécialisée (Sirscan 2000 Automatic (I2A)).
Abréviations : F : furanes, NOR : norfloxacine, LNZ : linézolide, TOB : tobramicine, SXT : sulfaméthoxazole/triméthoprime, FOX : céfoxitine, KAN : kanamycine, CN : gentamicine, FF : fosfomycine, OFX : ofloxacine, FA : acide fusidique, RA : rifampicine, TET : tétracycline, DA : clindamycine, ERY : érythromycine, SYN : synergistine
A. Phénotype sauvage, production d’une pénicillinase naturelle de bas niveau, permettant la résistance à l’amoxicilline (une pénicilline). B. Résistance acquise aux β-lactamines (AM, AMX, CTX, FEP, CAZ) par production d’une β-lactamase à spectre étendu (BLSE). Résistance acquise associée aux fluoroquinolones (NA et OFX), à la fosfomycine (FF) et au triméthoprime/sulfaméthoxazole (SXT). C. Résistance acquise à l’ensemble des β-lactamines testées (AM, AMC, CTX, FOX, FEP, CAZ, TPZ, MEC, ETP) par production d’une β-lactamase nommée carpabénèmase. Résistance acquise associée aux fluoroquinolones (NA et OFX), aux aminosides (CN et AK), aux furanes (F), à la fosfomycine (FF), au triméthoprime/sulfaméthoxazole (SXT) (Antibiotique vert : S, jaune : I, rouge : R).
Abréviations : AX : amoxicilline, FOX : céfoxitine, CTX : céfotaxime, NA : acide nalidixique, FF : fosfomycine, CN : gentamine, AMC : amoxicilline/acide clavulanique, FEP : céfépime, SXT : triméthoprime/sulfaméthoxazole, AK : amikacine, CAZ : ceftazidime, OFX : ofloxacine, ETP : ertapénème, MEC : mécillinam, TPZ : pipéracilline/tazobactam, F : furanes.
Les techniques diagnostiques récentes, de plus en plus performantes et rapides, visent à pouvoir rapidement adapter l’antibiothérapie avant l’obtention de l’antibiogramme. Les techniques d’identification des bactéries se sont améliorées ces dernières années grâce à l’utilisation de la spectrométrie de masse, technologie basée sur l’analyse du profil protéique ribosomal des bactéries, se substituant aux techniques biochimiques dans de nombreux laboratoires (11). Ainsi, à partir d’une culture de bactéries sur un milieu solide, il est possible d’obtenir une identification en 15 minutes seulement. Les techniques de biologie moléculaire permettent de mettre en évidence les gènes de résistance des bactéries directement sur prélèvement (12). Sur les colonies bactériennes, des tests colorimétriques (détection de β-lactamases) ou immuno-chromatographiques (détection de la PLP2A) sont réalisés en 20 minutes et permettent d’obtenir des résultats précocement (13) (14). De plus, des améliorations de la technique de l’antibiogramme par diffusion en milieu gélosé permettent une lecture plus précoce de l’antibiogramme et sont actuellement en cours d’évaluation.
À l’avenir, la mise en place des techniques de séquençage du génome entier permettra d’avoir facilement la cartographie des gènes de résistance des bactéries d’un patient et pourra peut-être permettre un choix d’antibiotique plus pertinent et adapté aux potentielles émergences de souches résistantes chez le patient. Ce type de technique pourra également permettre de documenter facilement les transmissions croisées de bactéries résistantes (15).
Prévention de la résistance aux antibiotiques en ville et à l’hôpital
La prévention de l’antibiorésistance doit être menée conjointement en santé animale et humaine car les bactéries résistantes diffusent entre ces deux secteurs (Figure 5).
En santé humaine, lutter contre l’antibiorésistance consiste en premier lieu à éviter de prescrire des antibiotiques aux patients qui n’en ont pas besoin. Cela peut s’avérer complexe lors d’examens cliniques difficiles, chez des patients ayant des maladies inflammatoires (cancer) pouvant mimer une infection ou lorsque les résultats de certains examens de laboratoire ne sont pas disponibles. De plus, le suivi des recommandations de prescription médicale est capital. Ces recommandations sont produites par la Haute autorité de santé (16) et les sociétés savantes telles que la Société de pathologie infectieuse de langue française (17).
La réalisation de prélèvements microbiologiques permet également d’adapter l’antibiothérapie en fonction de l’antibiogramme en évitant les antibiotiques les plus générateurs de résistance (antibiotiques à spectre large ayant une activité anti-anaérobies et/ou un impact sur les microbiotes) et en évitant les molécules de dernier recours (18). Un effort particulier doit être réalisé sur le raccourcissement des durées d’antibiothérapie, la majorité des infections bactériennes pouvant être traitées en moins de 8 jours.
À l’hôpital, les mesures d’hygiène visent à diminuer les infections nosocomiales ou les transmissions croisées de bactéries résistantes et sont définies et adaptées des recommandations nationales par les équipes d’hygiène des hôpitaux. Lutter contre l’antibiorésistance nécessite la coopération de plusieurs praticiens hospitaliers et de leurs équipes : médecin en charge du malade, microbiologiste, infectiologue, pharmacien hospitalier, praticien en hygiène. De plus, dans chaque établissement hospitalier, est nommé un référent antibiotique (praticien ayant une compétence en antibiothérapie). Il a un rôle de conseil en antibiothérapie, rédige des protocoles de prescription, fait des formations et des audits. Il suit la consommation des antibiotiques et confronte celle-ci aux données épidémiologiques locales de résistance. Il est ainsi possible d’évaluer l’efficacité des actions de prévention menées dans l’établissement hospitalier.
En médecine de ville, plusieurs outils d’aide au bon usage des antibiotiques sont disponibles tels que Antibioclic, un logiciel d’aide à la décision thérapeutique en antibiothérapie (19). La disponibilité d’une cellule de conseil en antibiothérapie pour tout médecin prescripteur pourra également améliorer les pratiques. Le médecin doit utiliser des tests de diagnostic rapide en cas d’angine pour discriminer les angines virales, les plus fréquentes et ne nécessitant pas d’antibiotiques, des angines bactériennes. Il doit aussi promouvoir la vaccination.
La mauvaise utilisation des antibiotiques en ville a des répercussions à l’hôpital. Si un traitement antibiotique mal adapté a été instauré en ville pour un patient donné, une molécule plus génératrice de résistance sera utilisée si celui-ci se présente ensuite aux urgences de l’hôpital.
Rôle des patients et des citoyens
Les dernières prérogatives en matière de santé placent le patient en tant qu’ « acteur de sa prise en charge ». Il est donc indispensable que celui-ci connaisse les enjeux de la résistance aux antibiotiques pour sa propre santé et celle des autres. Les patients doivent éviter de solliciter leur médecin pour la prescription d’antibiotiques. Ils doivent également consulter des professionnels de santé et ne pas faire d’automédication. Faire connaître l’antibiorésistance est donc un des objectifs des politiques mondiale et nationale de lutte contre l’antibiorésistance car celle-ci reste à ce jour peu ou mal connue des Français (sondage IFOP 2017). Ces derniers ne perçoivent pas encore la problématique dans sa globalité et surtout le phénomène n’est pas une préoccupation majeure à leurs yeux. Pour les citoyens, lutter contre l’antibiorésistance consiste à s’impliquer en se vaccinant, en prévenant les infections sexuellement transmissibles et en respectant les mesures d’hygiène des mains et alimentaire. Pour éviter la dissémination des antibiotiques dans l’environnement, les citoyens doivent rapporter leurs médicaments non utilisés en pharmacie pour élimination selon la filière dédiée (Cyclamed). Enfin, les influenceurs (réseaux sociaux, etc.) ont la responsabilité de ne pas diffuser de fausses informations en matière santé (automédication, vaccination).
Références
- Duckett S. 1999. Ernest Duchesne and the concept of fungal antibiotic therapy. Lancet 354 :2068–2071.
- Jehl F, Chomarat M, Tankovic J, Gérard A. 2012. De l’antibiogramme à la prescription. bioMérieux S.A.
- Institut Pasteur https://www.pasteur.fr/fr/centre-medical/fiches-maladies/resistance-aux-antibiotiques.
- Yoneyama H, Nakae T. 1993. Mechanism of efficient elimination of protein D2 in outer membrane of imipenem-resistant Pseudomonas aeruginosa. Antimicrob Agents Chemother 37 :2385–2390.
- 1948. STREPTOMYCIN treatment of pulmonary tuberculosis. Br Med J 2 :769–782.
- Murray BE, Rensimer ER, DuPont HL. 1982. Emergence of high-level trimethoprim resistance in fecal Escherichia coli during oral administration of trimethoprim or trimethoprim--sulfamethoxazole. N Engl J Med 306 :130–135.
- Cantón R, Horcajada JP, Oliver A, Garbajosa PR, Vila J. 2013. Inappropriate use of antibiotics in hospitals : the complex relationship between antibiotic use and antimicrobial resistance. Enferm Infecc Microbiol Clin 31 Suppl 4 :3–11.
- Santé Publique France. 2018. Consommation d’antibiotiques et résistance aux antibiotiques en France : une infection évitée, c’est un antibiotique préservé !
- Touat M, Opatowski M, Brun-Buisson C, Cosker K, Guillemot D, Salomon J, Tuppin P, de Lagasnerie G, Watier L. 2019. A Payer Perspective of the Hospital Inpatient Additional Care Costs of Antimicrobial Resistance in France : A Matched Case-Control Study. Appl Health Econ Health Policy 17 :381–389.
- Société Française de Microbiologie. 2019. Comité de l’antibiogramme de la Société Française de Microbiologie Recommandations 2019 V.2.0 Mai.
- Lavigne J-P, Riegel P. 2015. [Mass spectrometry in bacteriology]. Ann Biol Clin (Paris) 73 :113–125.
- Cattoir V, Merabet L, Djibo N, Rioux C, Legrand P, Girou E, Lesprit P. 2011. Clinical impact of a real-time PCR assay for rapid identification of Staphylococcus aureus and determination of methicillin resistance from positive blood cultures. Clin Microbiol Infect 17 :425–431.
- Farfour E, Si Larbi AG, Cardot E, Limousin L, Mathonnet D, Cahen P, Vasse M, Lesprit P. 2019. Impact of rapid diagnostic tests on the management of patients presenting with Enterobacteriaceae bacteremia. Med Mal Infect 49 :202–207.
- Garnier M, Gallah S, Vimont S, Benzerara Y, Labbe V, Constant A-L, Siami S, Guerot E, Compain F, Mainardi J-L, Montil M, Quesnel C, BLUE-CarbA study group. 2019. Multicentre randomised controlled trial to investigate usefulness of the rapid diagnostic βLACTA test performed directly on bacterial cell pellets from respiratory, urinary or blood samples for the early de-escalation of carbapenems in septic intensive care unit patients : the BLUE-CarbA protocol. BMJ Open 9 : e024561.
- Köser CU, Holden MTG, Ellington MJ, Cartwright EJP, Brown NM, Ogilvy-Stuart AL, Hsu LY, Chewapreecha C, Croucher NJ, Harris SR, Sanders M, Enright MC, Dougan G, Bentley SD, Parkhill J, Fraser LJ, Betley JR, Schulz-Trieglaff OB, Smith GP, Peacock SJ. 2012. Rapid Whole-Genome Sequencing for Investigation of a Neonatal MRSA Outbreak. N Engl J Med 366 :2267–2275.
- Haute Autorité de Santé Recommandations de bonne pratique https://www.has-sante.fr/portail/jcms/c_1101438/fr/tableau-des-recommandations-ou-travaux-relatifs-….
- Société de Pathologie Infectieuse de Langue Française (SPILF) http://www.infectiologie.com/fr/recommandations.html.
- Agence nationale de sécurité du médicament et des produits de santé. 2016. Liste des antibiotiques critiques Actualisation 2015 https://ansm.sante.fr.
- Site internet Antibioclic https://antibioclic.com/.