La traduction correspond à l'étape de décodage de l'information génétique contenue dans l'ARN messager pour produire une protéine. Cette étape essentielle dans l'expression du génome nécessite l'intervention de différentes protéines et de molécules d'ARN : ARN messager, ARN de transfert et ARN ribosomiques. Ensemble, ces différents acteurs permettent de faire correspondre, à chaque triplet de nucléotides de la séquence codante, un acide aminé.
D’un langage à l’autre : d’une séquence de nucléotides à une séquence d’acides aminés
Une grande partie du phénotype macroscopique d’un individu découle directement du phénotype moléculaire de ses cellules, c’est-à-dire de son protéome. Les protéines sont des polymères d’acides α-aminés, et l’information nécessaire à leur synthèse est contenue dans l’ADN, qui est un polymère de (désoxyribo)nucléotides. Il existe 20 acides α-aminés différents, mais seulement 4 nucléotides différents. L’expression génétique comporte donc une étape de changement de langage, soit le passage d’un langage à 4 lettres (l’ADN, et sa copie en ARN messager) à un langage à 20 lettres (les protéines). Le passage d’une molécule d’ARN messager (ARNm) à la protéine qu’elle code s’appelle la traduction.
Cette synthèse présente les modalités de la traduction chez les Bactéries et les Eucaryotes. Lorsqu’aucune précision n’est donnée, les informations présentées s’appliquent à ces deux taxons.
Où commencer ?
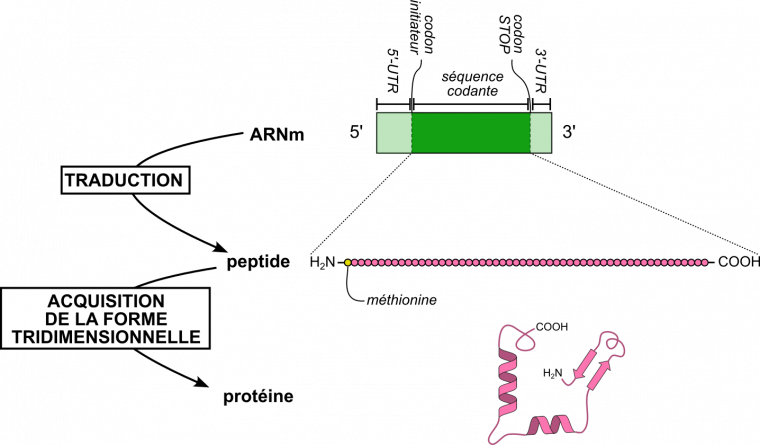
Si l’ARNm porte l’information génétique du gène transcrit, une partie de cet ARNm ne sera pas traduite (elle est dite non codante) : il s’agit des extrémités 5’-UTR (UTR pour UnTranslated Region, région non traduite) en amont de la phase codante et 3’-UTR en aval (Figure 1). La région traduite correspond donc uniquement à la partie de l’ARN messager comprise entre le codon initiateur indiquant le démarrage de la traduction et le codon stop de terminaison. Cette portion de l'ARN messager est appelée séquence codante ou cadre ouvert de lecture (Open Reading Frame en anglais).
Le codon initiateur et le codon stop sont indiqués : ils encadrent, dans l’ARN messager (ARNm), la séquence codante (représentée en vert foncé). Les extrémités 5’-UTR et 3’-UTR (représentées en vert clair) sont les régions non codantes situées respectivement avant le codon initiateur et après le codon stop.
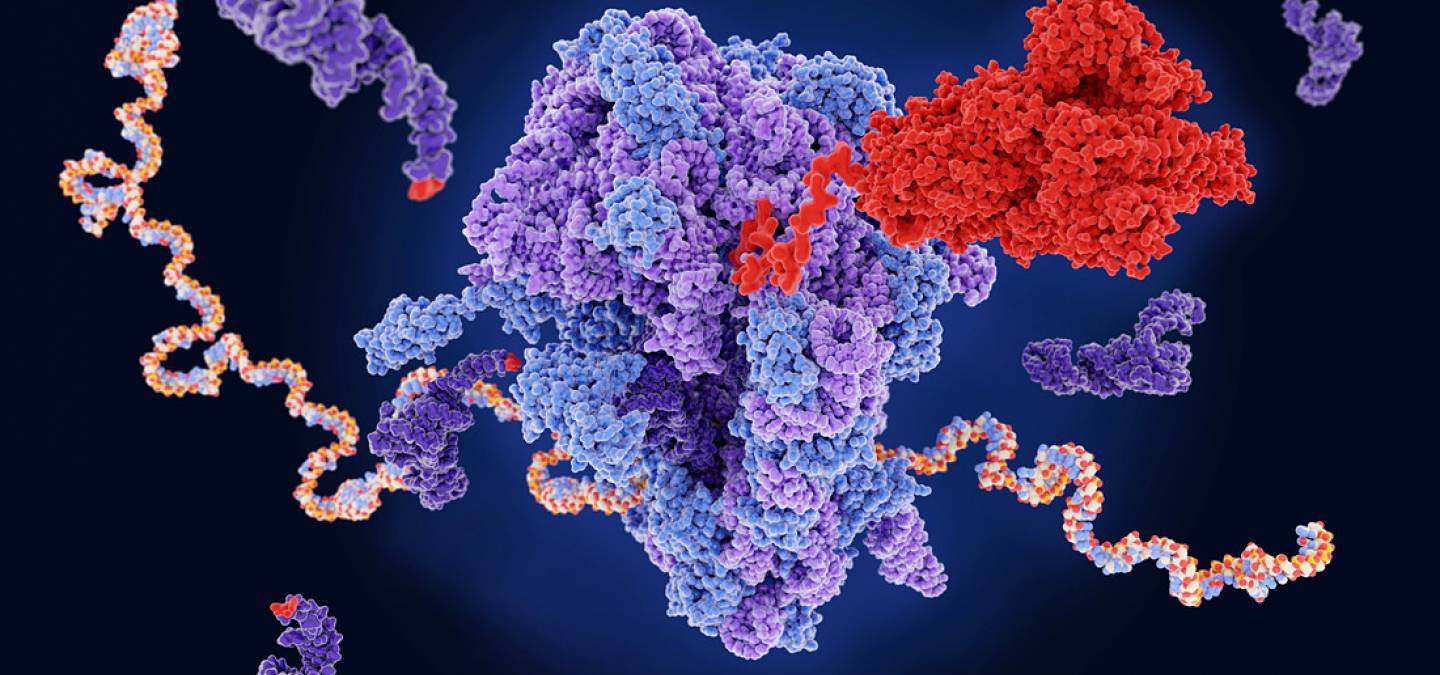
La lecture des informations portées par les ARN messagers est assurée par les ribosomes. Un ribosome est un complexe formé de deux sous-unités, chacune constituée de protéines et d’ARN ribosomiques (ARNr). La structure du cœur catalytique des ribosomes est globalement conservée chez tous les êtres vivants. Au cours de l’évolution, des composants ribonucléiques et protéiques se sont rajoutés autour de ce cœur catalytique commun. La masse des différents constituants des ribosomes est liée à leur temps de sédimentation, exprimé en Svedberg1 (S), lorsqu’ils sont soumis à ultracentrifugation (Tableau 1).
| E. coli (Bactérie) | H. sapiens (Eucaryote) | |
|---|---|---|
| Ribosome total | 70S | 80S |
| 2,3 MDa | 4,3 MDa | |
| 54 protéines | 80 protéines | |
| 3 ARNr | 4 ARNr | |
| Grande sous-unité | 50S | 60S |
| 33 protéines | 47 protéines | |
| ARNr 23S : 2904 bases | ARNr 28S : 5034 bases | |
| - | ARNr 5,8S : 156 bases | |
| ARNr 5S : 121 bases | ARNr 5S : 121 bases | |
| Petite sous-unité | 30S | 40S |
| 21 protéines | 33 protéines | |
| ARNr 16S : 1542 bases | ARNr 18S : 1870 bases |
La petite sous-unité du ribosome, qui porte l’ARN de transfert (ARNt) initiateur portant la méthionine2, est recrutée au niveau de l’ARNm par des facteurs d’initiation de la traduction (Initiation Factors, notés IF chez les Bactéries et eIF chez les Eucaryotes). La petite sous-unité se déplace le long de l’ARNm dans le sens 5’ → 3’, et s’arrête au niveau d’une séquence appelée site d’initiation, qui comporte le codon de démarrage AUG.
Le site d’initiation diffère chez les Bactéries et chez les Eucaryotes :
-
Chez les Bactéries à Gram négatif, il s'agit de la séquence de Shine-Dalgarno, située environ 8 nucléotides en amont de l’AUG initiateur, et ayant pour séquence consensus AGGAGGU (parfois seulement GAGG).
-
Chez les Eucaryotes, il s'agit de la séquence de Kozak GCCR3CCAUGG, qui contient l’AUG initiateur.
La fixation à l’AUG initiateur est primordiale pour au moins deux raisons :
-
La protéine produite à partir d’un autre AUG serait plus longue ou plus courte ;
-
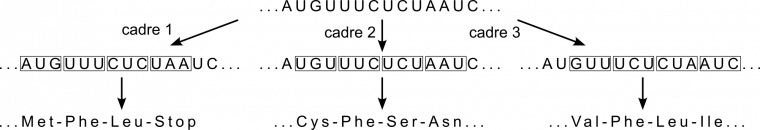
L'information génétique est lue par groupe de trois nucléotides, appelés codons. Chaque séquence de nucléotides peut donc potentiellement être lue de trois façons différentes, selon le cadre de lecture utilisé, ce qui donnera potentiellement des peptides totalement différents (Figure 2). La fixation de la petite sous-unité du ribosome à l’AUG initiateur impose le cadre de lecture.
L’extrait de séquence d’ARNm AGUUUCUCUAAUC peut être traduit en trois peptides totalement différents, selon le cadre de lecture, c’est-à-dire la façon de regrouper par trois les nucléotides.
Le code génétique désigne la correspondance entre codons et acides aminés
Propriétés du code génétique
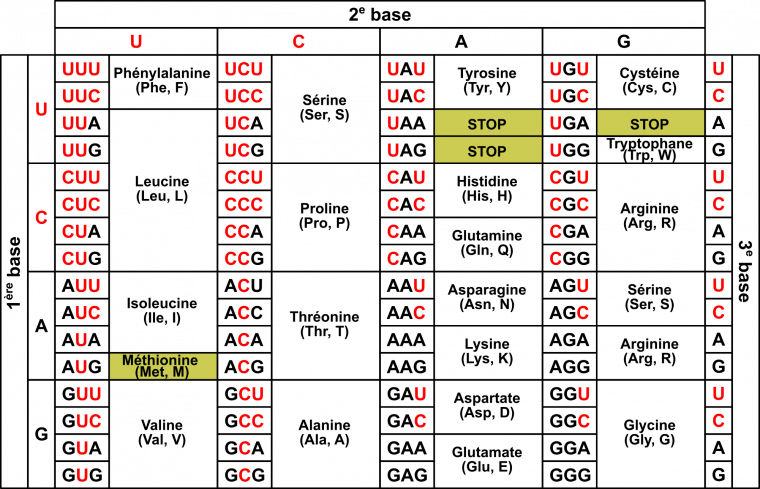
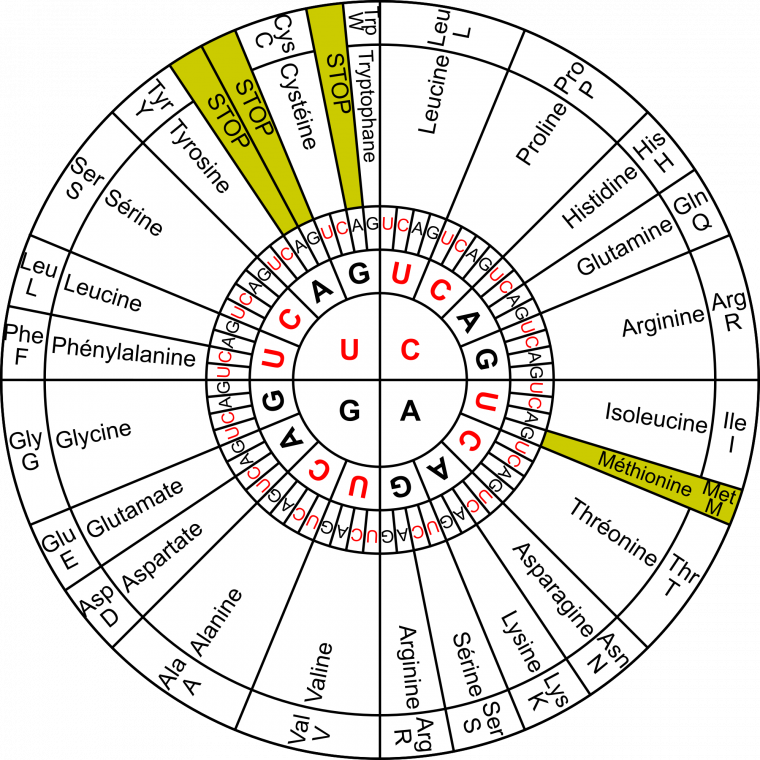
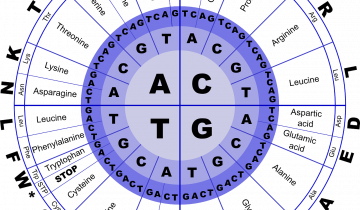
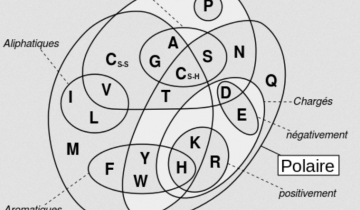
Le code génétique est la correspondance entre le « langage nucléotide » et le « langage acide α-aminé » (Figure 3). Il est généralement présenté sous la forme d’un tableau (Figure 4), qui donne la correspondance entre un triplet de nucléotides (aussi appelé codon) et l’acide α-aminé correspondant, mais on peut aussi le représenter sous la forme de cercles concentriques (Figure 5).
Le code génétique est souvent présenté comme universel, car commun à la plupart des êtres vivants. Il existe cependant quelques variations par rapport à ce standard, notamment chez les Bactéries, les Archées, et les organites semi-autonomes (mitochondries et chloroplastes) 1.
Code génétique ou information génétique ?
Il convient donc de bien distinguer la notion d’information génétique, qui correspond au message porté par une molécule d’acide nucléique, de celle de code génétique, qui désigne la correspondance entre codons et acides aminés.
Les pyrimidines (U et C) sont représentées en rouge et les purines (A et G) en noir. Les quatre codons particuliers (codon Met initiateur, et les trois codons stop) sont colorés en jaune.
Analysons ce code génétique :
-
Il existe 64 codons différents. En effet, chaque codon est composé de trois nucléotides et il existe quatre nucléotides différents. Il y a donc 43 = 64 combinaisons possibles.
-
Le code génétique est non ambigu : un codon a une seule et unique signification, et tous les codons possibles ont une signification.
-
Le code génétique est redondant (ou dégénéré) : un acide α-aminé peut être codé par plusieurs codons, qui comportent des variations sur la troisième position du codon:
-
pour les acides α-aminés codés par 4 codons, les deux premiers nucléotides sont identiques ;
-
pour les acides α-aminés codés par 2 codons, les deux premiers nucléotides sont identiques, et le 3e est toujours une pyrimidine (U ou C) ou toujours une purine (A ou G).
-
la leucine est codée par 6 codons.
-
-
Il existe quatre codons particuliers :
-
Le codon AUG code la méthionine. Comme il correspond également au codon initiateur, toute protéine débute a priori par une méthionine. Ce même codon est aussi utilisé pour coder des méthionines au milieu des protéines. Cependant, c’est alors un autre ARNt, l’ARNt élongateur qui est utilisé.
-
Les codons UAA, UAG et UGA sont des codons stop, qui ne correspondent pas à un acide aminé. Historiquement, ces codons étaient appelés ocre, ambre et opale.
-
Influence des mutations sur le phénotype
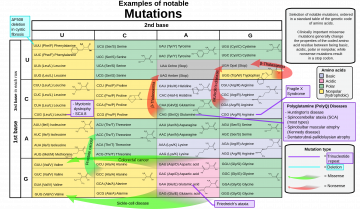
Des propriétés de ce code génétique, on peut déduire que les mutations génétiques ponctuelles de la séquence codante des gènes ont des conséquences inégales :
-
Lorsque la mutation est une addition (ajout d’un nucléotide) ou une délétion (suppression d’un nucléotide), le cadre de lecture est décalé à partir du site de la mutation (voir la figure 2 pour la notion de cadre de lecture). Dans ce cas, la séquence du peptide est profondément modifiée, en particulier si cette mutation se produit au début de la séquence codante (proche de l’extrémité 5’).
-
Lorsque la mutation est une substitution (remplacement d’un nucléotide par un autre), plusieurs cas de figure peuvent se poser :
-
La substitution est dite silencieuse si, par redondance du code génétique, le codon muté donne le même acide α-aminé. Exemple : UUA → UUG ; dans ce cas, l’acide α-aminé est toujours la leucine. La mutation constitue donc un élément de diversité génétique, mais sans aucune conséquence sur le phénotype.
-
La substitution est dite faux-sens si le codon muté donne un acide α-aminé différent. Exemple : UUA → UCA ; dans ce cas, l’acide α-aminé n’est plus la leucine, mais la sérine. Comme ces deux acides α-aminés ont des radicaux très différents (hydrophobe pour la leucine, hydrophile pour la sérine), la structure tridimensionnelle de la protéine mutée peut potentiellement être fortement modifiée par rapport à la protéine originelle, ce qui peut entrainer une modification, voire une perte de sa fonction.
-
Un cas particulier de la substitution faux-sens est le cas de la substitution synonyme, où l’acide α-aminé muté a des propriétés proches de l’acide α-aminé d’origine. Exemple : UUA → AUA ; dans ce cas, l’acide α-aminé n’est plus la leucine, mais l’isoleucine. Comme ces deux acides α-aminés ont des chaines latérales très proches (taille presque identique, hydrophobes tous les deux), la protéine mutée voit sa structure très légèrement modifiée par rapport à la protéine originelle et les modifications de son activité sont généralement très faibles.
-
Enfin, la substitution est dite non-sens si le codon muté devient un codon stop. Dans ce cas, la protéine mutée est tronquée et sa fonction généralement profondément modifiée, en particulier si cette mutation se produit au début de la séquence codante (proche de l’extrémité 5’).
-
Les aminoacyl-ARNt-synthéthases lient les acides aminés à leurs ARNt
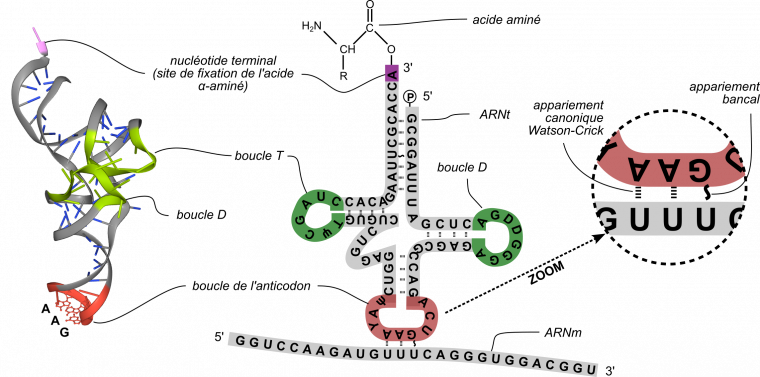
D’un point de vue moléculaire, la correspondance entre les codons portés par l’ARNm et les acides α-aminés présents dans la protéine synthétisée est réalisée par des ARN non codants, les ARN de transfert (ARNt). Il s’agit de molécules longues de 70 à 90 nucléotides dont la structure secondaire est qualifée de feuille de trèfle du fait de la complémentarité de bases entre de petites portions (Figure 6). Les ARNt comportent quelques bases et nucléotides modifiés (notamment, de la thymine, ce qui est rare pour de l’ARN, mais également de l’inosine, de la dihydrouridine, de la pseudouridine ainsi que de nombreuses autres modifications plus ou moins complexes). L’une des trois boucles comporte une séquence nommée anticodon, capable de s’apparier au codon complémentaire dans l’ARNm. Chacun des ARNt diffère (entre autres) par la séquence de l’anticodon. Au niveau de l’extrémité 3’ de l’ARNt, l’acide α-aminé correspondant est fixé de façon covalente par une liaison ester sur la séquence CCA, formant ainsi un aminoacyl-ARNt. Cette séquence CCA est conservée dans tout le monde vivant.
Gauche : structure tridimensionnelle de l’ARNt complémentaire du codon UUU, correspondant à la phénylalanine. Droite : représentation de la structure secondaire de ce même ARNt, la boucle de l’anticodon étant appariée au codon lui correspondant sur l’ARNm. Pour les explications concernant l’appariement bancal G-U, voir la partie Les mécanismes de la traduction. La dihydrouridine (D) est presque toujours présente dans la boucle D, tandis que la ribothymidine (T) et la pseudouridine (Ψ) sont presque toujours présentes dans la boucle T. L’inosine (Y) est un autre nucléotide original présent dans l’ARNt, mais généralement absent des autres ARN, notamment de l’ARNm. Figure de gauche obtenue avec LibMol, à partir du fichier 4TNA.
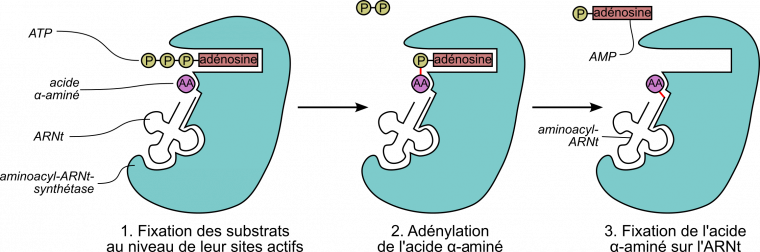
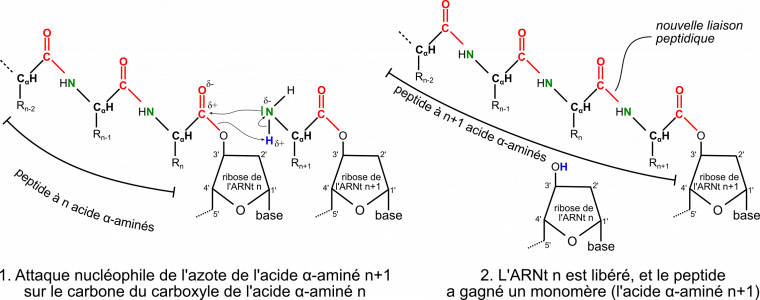
Les aminoacyl-ARNt-synthéthases sont les enzymes qui produisent les aminoacyl-ARNt en formant une liaison ester entre l’extrémité 3’OH d’un ARNt la fonction acide carboxylique COOH de l’acide aminé correspondant (Figure 7). Cette réaction, appelée aminoacylation, a bien entendu lieu en dehors de la traduction et elle permet à la cellule de disposer d’un stock suffisant d’aminoacyl-ARNt à tout moment.
Il existe 20 aminoacyl-ARNt synthétases différentes : chacune reconnaît un acide aminé et tous ses ARNt correspondants. La grande spécificité de substrat de ces enzymes est importante pour que chacune fixe le bon acide α-aminé sur les ARNt correspondants. Ces enzymes garantissent donc la fidélité de la traduction. Grâce à leur fonction de correction des erreurs, certaines aminoacyl-ARNt synthétases ne chargent un ARNt de manière erronée qu’une fois sur 10 000.
Par ailleurs, la formation de l’aminoacyl-ARNt consomme un ATP. L’énergie libérée par son hydrolyse est utilisée pour former la liaison ester entre l’ARNt et l’acide α-aminé. Ces substrats seront ensuite utilisés pendant la traduction proprement dite.
Ces enzymes ont la particularité de posséder deux activités enzymatiques distinctes : une activité adénosyl-transférase (fixation d’un AMP sur l’acide α-aminé) et une activité aminoacyl-transférase (fixation de l’acide aminé sur les différents ARNt associés à cet acide aminé).
Les mécanismes de la traduction
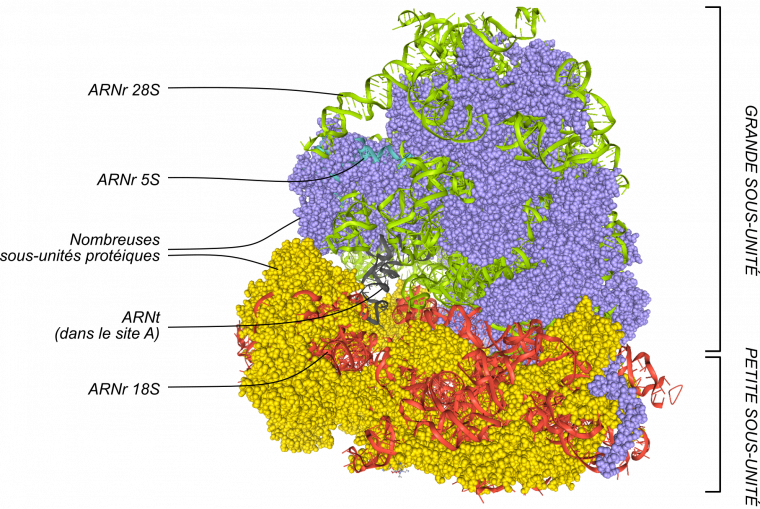
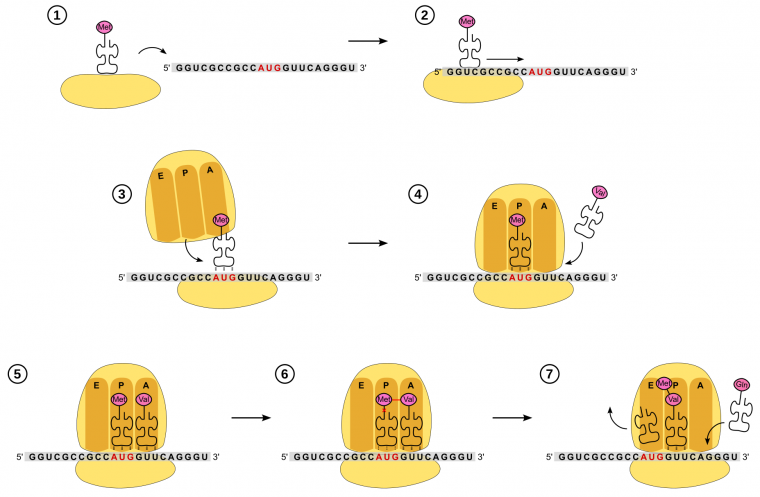
Une fois que le complexe formé par la petite sous-unité du ribosome et l'ARNt initiateur est positionnée sur le codon initiateur, la grande sous-unité va s’associer pour former un ribosome complet (Figure 8). Le ribosome comporte trois sites :
- le site A (A pour aminoacyl-ARNt). Il s’agit du site d’entrée, où les aminoacyl-ARNt arrivent dans le ribosome ;
- le site P (pour peptidyl-ARNt), où se trouve l’ARNt lié au peptide en cours d’élongation ;
- le site E (pour exit), où l’ARNt ayant donné son acide aminé quitte le ribosome.
Les parties protéiques des sous-unités sont représentées sous forme de boules, lilas pour la grande sous-unité, et jaune pour la petite sous-unité. Les ARN ribosomiques sont quant à eux représentés sous forme de rubans, vert pour la grande sous-unité, safran pour la petite. Un ARNt est représenté (en noir). Figure obtenue avec LibMol, à partir du fichier 4UG0.
Lorsque le ribosome complet se forme, l'ARNt initiateur est positionné au niveau du site P (Figure 9). Puis, l’aminoacyl-ARNt complémentaire du deuxième codon se met en position au niveau du site A.
C’est à ce moment que va avoir lieu la synthèse de la première liaison peptidique à proprement parler (Figure 10). La méthionine se détache de son ARNt (par rupture de la liaison ester) et se fixe au 2e acide aminé, au niveau de sa fonction amine, formant ainsi une liaison peptidique. La réaction nécessite de l’énergie : elle est indirectement apportée par l’hydrolyse d’un ATP s'étant produite lors de la formation de l’aminoacyl-ARNt par l'aminoacyl-ARNt-synthéthase (voir partie précédente et Figure 7).
1. La petite sous-unité du ribosome, déjà chargée avec l’ARN initiateur, se fixe à l'ARN messager
2. Ce complexe parcourt l’ARN messager dans le sens 5’ → 3’ jusqu’à rencontrer le site d’initiation.
3. La grande sous unité se fixe. L’ARNt initiateur est au niveau du site P.
4. Un ARNt entre au niveau du site A.
5. La reconnaissance codon-anticodon stabilise l’ARNt au niveau du site A
6. L’activité catalytique de l’ARNr de la grande sous-unité du ribosome permet la formation de la liaison peptidique entre les deux acides aminés adjacents. Dans le même temps, la liaison entre la méthionine et son ARNt est rompue.
7. Le ribosome se décale de trois nucléotides vers l’extrémité 3’. L’ARNt initiateur sort au niveau du site E. Un nouvel ARNt peut entrer au niveau du site A.
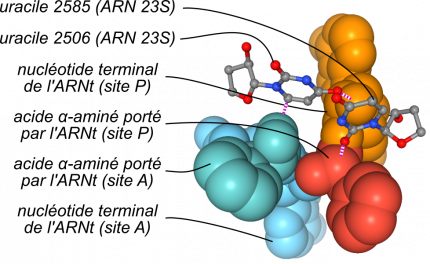
Cette activité enzymatique est appelée peptidyl-transférase. Elle a ceci d’original qu’elle est entièrement catalysée par l’ARN ribosomique, et non par une protéine 1. Un ARN à activité catalytique est appelé ribozyme (mot constitué par la contraction des mots « ribonucléique » et « enzyme »). La figure 11 présente le site actif responsable de l’activité peptidyl-transférase, composé de nucléotides de l’ARN ribosomique 23S chez les Bactéries et de l’ARNr 28S chez les Eucaryotes.
Il est important ici de revenir sur la redondance du code génétique. La phénylalanine, par exemple, est codée par les deux codons 5’-UUC-3’ et 5’-UUU-3’ (voir code génétique, Figures 4 et 5). Il n’existe pourtant qu’un seul ARNt correspondant à la phénylalanine, et son anticodon a pour séquence 3’-AAG-5’.
-
La complémentarité entre 3’-AAG-5’ (anticodon) et 5’-UUC-3’ (codon) est triviale.
-
L’anticodon 3’-AAG-5’ peut cependant aussi s’apparier avec le codon 5’-UUU-3’. L’appariement non canonique entre G et U est dit bancal (wobble pairing en anglais). Ceci explique le fait que la plupart des codons codant plusieurs acides aminés diffèrent entre eux seulement au niveau de la troisième base du codon.
Ce flottement, qui permet à un même ARNt de s’apparier à plusieurs codons, fait qu’il n’existe pas forcément autant de types d’ARNt que de codons. Par exemple, Helicobacter pylori (une bactérie parasite de l’estomac des mammifères) dispose de 36 ARNt différents, au lieu des 61 attendus pour décoder les 61 codons possibles (64 codons − 3 codons stop) 1.
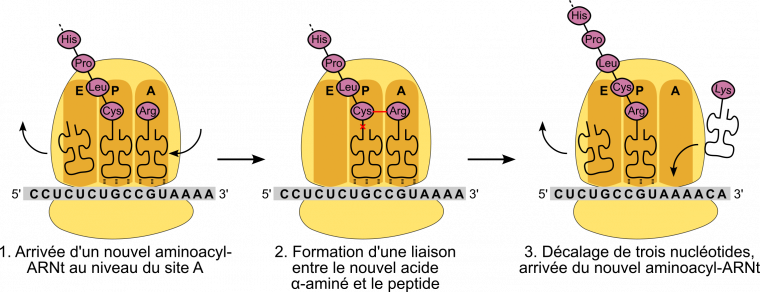
Le processus de traduction se répète codon après codon et la protéine s’allonge donc dans le sens extrémité N-terminale (portant la fonction amine NH2) vers l’extrémité C-terminale (portant la fonction acide carboxylique COOH) (Figure 12). La vitesse d’élongation est de l’ordre de 5 à 20 acides aminés par seconde, le processus étant globalement plus rapide chez les Bactéries que chez les Eucaryotes. Au fur et à mesure que le ribosome progresse et que de nouveaux acides α-aminés sont ajoutés au peptide en cours d’élongation, les ARNt non aminoacylés sont relâchés dans le cytosol via le site E. Ils seront rechargés en aminoacyl-ARNt par les aminoacyl-ARNt-synthétases pour être réutilisés par le ribosome pour d’autres protéines. Le bon déroulement de l’élongation nécessite des protéines appelées facteurs d’élongation (elongation factors en anglais, notés EF chez les Bactéries et eEF chez les Eucaryotes).
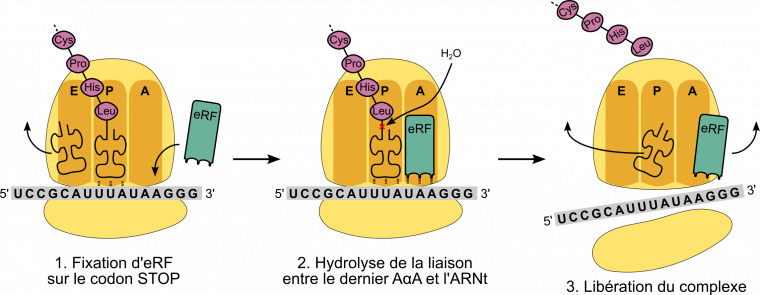
La traduction s’arrête lorsque le ribosome rencontre un codon stop (Figure 13). Il n’existe pas d’ARNt capables de décoder les trois codons stop. En revanche, une protéine reconnaît spécifiquement ces codons : chez les Eucaryotes, c’est la protéine eRF (pour eukariotic release factor, soit facteur de libération [du peptide]). Lorsqu’eRF se fixe sur le codon stop, le ribozyme catalyse l’hydrolyse de la liaison entre le peptide et l’ARNt et le peptide synthétisé peut donc être libéré par le ribosome. Les différents éléments – petite et grande sous-unité du ribosome, eRF, dernier ARNt, ARNm – se désolidarisent alors et la traduction est terminée. Chez les Bactéries, trois facteurs de terminaison (RF1, RF2 et RF3) sont impliqués dans l’arrêt de la traduction.
La reconnaissance eRF-codon stop (non détaillée ici) repose sur l’établissement de liaisons faibles entre des chaînes latérales des acides α-aminés d’eRF et les trois bases du codon stop.
Alors que les ARNm bactériens sont linéaires, les ARNm eucaryotes se circularisent grâce à l’interaction, par l’intermédiaire de différentes protéines, de leurs parties 5’ et 3’. Grâce à cette circularisation, un ribosome qui termine la traduction de l’ARNm peut se réassocier rapidement au même ARNm pour redémarrer un nouveau cycle de traduction. Par ailleurs, la circularisation augmente aussi la durée de vie des ARNm eucaryotes en réduisant leur accessibilité aux nucléases.
Une protéine fonctionnelle est une protéine bien adressée
La fonction d’une protéine ne peut être dissociée de sa localisation (transmembranaire, cytosolique, extracellulaire…). Dans la plupart des cas, les protéines sont adressées vers leur compartiment final au cours de la traduction.
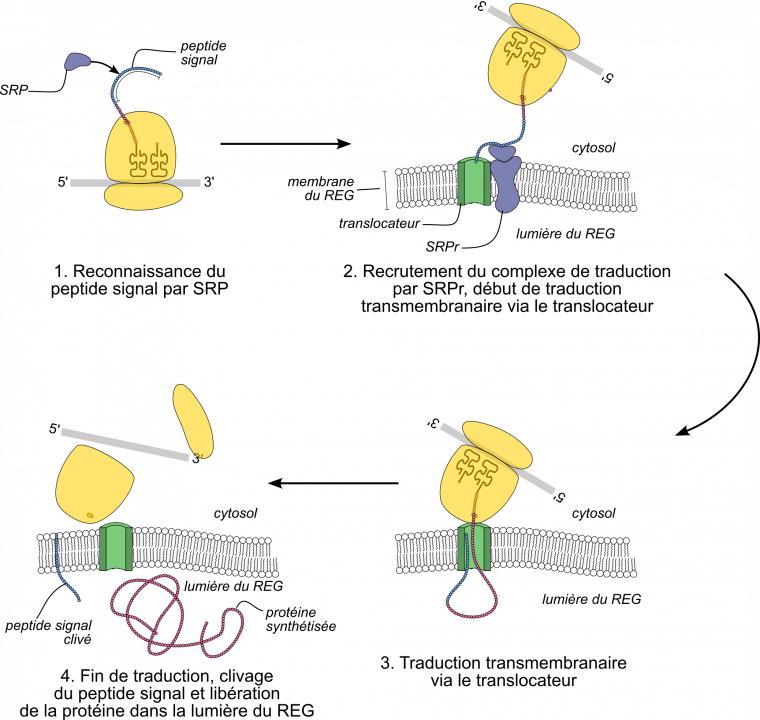
L’exemple développé ici est celui d’une protéine produite par une cellule eucaryote et sécrétée dans le milieu extracellulaire (Figure 14). Chez les Eucaryotes, les protéines sécrétées transitent par le réticulum endoplasmique granuleux (REG), puis sont libérées dans le milieu extracellulaire par exocytose. Une partie de la séquence peptidique (en l’occurrence, les 25 premiers acides aminés environ) constitue un peptide signal : elle porte le signal d’export vers le REG.
Dès que le peptide signal est synthétisé (au tout début de la traduction), il est reconnu par une protéine cytosolique (la protéine SRP, acronyme de Signal Recognition Particle, ou particule de reconnaissance du signal), et est adressé à une protéine de la membrane du REG, le translocateur (via le récepteur de SRP, SRPr). Dès lors, la traduction continue à travers la membrane du REG. À la fin de la traduction, le peptide signal (dont l’unique rôle était l’adressage) est clivé par hydrolyse. Comme l’adressage a lieu pendant la traduction, on parle d’adressage cotraductionnel.
Il existe de nombreux types d’adressage. Pour les protéines de la membrane plasmique, des compartiments (hors mitochondrie ou chloroplaste) ou les protéines sécrétées, il s’agit toujours d’un adressage cotraductionnel, via le translocateur du REG. Pour les protéines transférées au noyau, à la mitochondrie ou au chloroplaste, le transfert se fait après la traduction, et dépasse le cadre de cet article.
Les principales différences dans les modalités de traduction chez les Bactéries et les Eucaryotes
| Bactéries | Eucaryotes | |
|---|---|---|
| Moment de la traduction | En même temps que la transcription | Après transcription et export de l’ARNm du noyau |
| Lieu de la traduction | Cytosol | Cytosol, REG, organites semi-autonomes |
| ARNt initiateur | N-formylméthionine-ARNtiMet | Méthionine-ARNtiMet |
| Site d’initiation | Séquence de Shine-Dalgarno | Séquence de Kozak |
| Facteurs d’initiation (initiation factors) | IF1, IF2 et IF3 | eIF1, eIF1a, eIF2, eIF3, eIF4F (A,E,G), eIF4B, eIF5 |
| Facteurs d’élongation (elongation factors) | EF-Tu et EF-Ts, EF-G | eEF1α, eEF1βγ et eEF2 |
| Facteurs de terminaison (release factors) | RF1, RF2 et RF3 | eRF |
| Structure de l’ARNm pendant la traduction | Linéaire | Circularisé |
Résumé
La traduction consiste en la synthèse d’un peptide (polymère d’acides aminés) grâce à l’information contenue dans la séquence codante de l’ARNm (polymère de nucléotides), décryptée par un dictionnaire moléculaire (le code génétique). Ce code est constitué par les ARNt, qui sont liés de façon covalente à des acides α-aminés par des aminoacyl-ARNt-synthétases. L’anticodon de l’ARNt est complémentaire à un codon de l’ARNm. La catalyse enzymatique de l’élongation du peptide est réalisée par un ARNr à activité catalytique (ribozyme). La traduction commence au niveau du codon initiateur AUG, qui code la méthionine. Elle se poursuit jusqu’à un codon stop, reconnu par un ou plusieurs facteurs de terminaison, qui stoppe la traduction. Chez les Eucaryotes, les protéines du réseau endomembranaire et les protéines sécrétées sont traduites directement à travers un translocateur de la membrane du REG, grâce à un peptide signal faisant partie de la séquence traduite, et clivé ultérieurement.
Comme beaucoup de processus cellulaires, la traduction est régulée. Chez les eucaryotes, une pénurie de nutriments peut par exemple inhiber l’initiation de la traduction par dissociation du facteur eIF4E d’avec la molécule d’ARNm, empêchant ainsi le recrutement du ribosome. La stabilité de l’ARNm est également un facteur important de régulation ; le mode d’action des microARN est un exemple de ce mode de régulation.