En comparant les structures des ribosomes de différents organismes, nous pouvons explorer l'évolution de la vie.
Les premières structures à l’échelle atomique des ribosomes, déterminées en 2000, ont révélé les détails de la biosynthèse des protéines. La petite sous-unité ribosomique interagit avec l'ARN messager, en l'associant aux molécules d'ARN de transfert appropriées par correspondance entre codons et anticodons. La grande sous-unité ribosomique relie les acides aminés transportés par les ARN de transfert en une nouvelle chaîne protéique. Au cours des 25 dernières années, des milliers de structures supplémentaires de ribosomes ont été déterminées, ce qui a permis d'élucider de nombreuses étapes de la synthèse des protéines et, également, de comparer les structures des ribosomes de différents organismes.
Évolution de la complexité
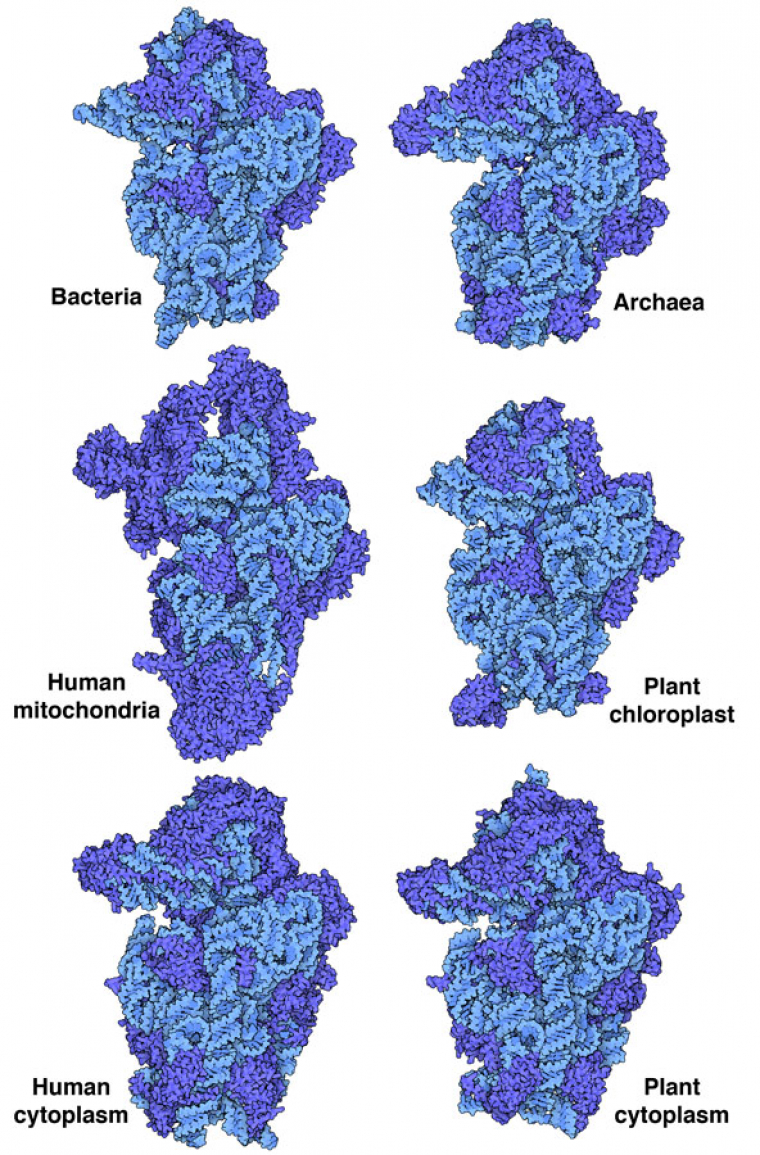
Comme tous les organismes vivants ont des ribosomes fonctionnels, les structures des ribosomes sont utiles pour étudier l'évolution moléculaire, ce qui nous permet de les comparer entre les différents taxons. La figure 1 représente la petite sous-unité ribosomique de différents organismes. Les bactéries ont des structures relativement simples. La petite sous-unité bactérienne, figurée dans l’entrée 1fjg de la banque de données sur les protéines (PDB), comprend un brin d'ARN d'une longueur de 1522 nucléotides et 20 protéines 1. De même, la petite sous-unité archéenne (entrée PDB 6tmf) comporte un petit brin d'ARN de 1485 nucléotides et 29 protéines 2. Cependant, les petites sous-unités humaines 3 et végétales 4 (entrées PDB 5a2q et 8auv respectivement) ont évolué vers une structure plus complexe, avec des brins d'ARN de plus de 1800 nucléotides et plus de 30 protéines.
L'ARN ribosomique est en bleu clair et les protéines en bleu plus foncé. Les illustrations montrent la partie de la sous-unité qui interagit avec la grande sous-unité ribosomale.
Travailler ensemble
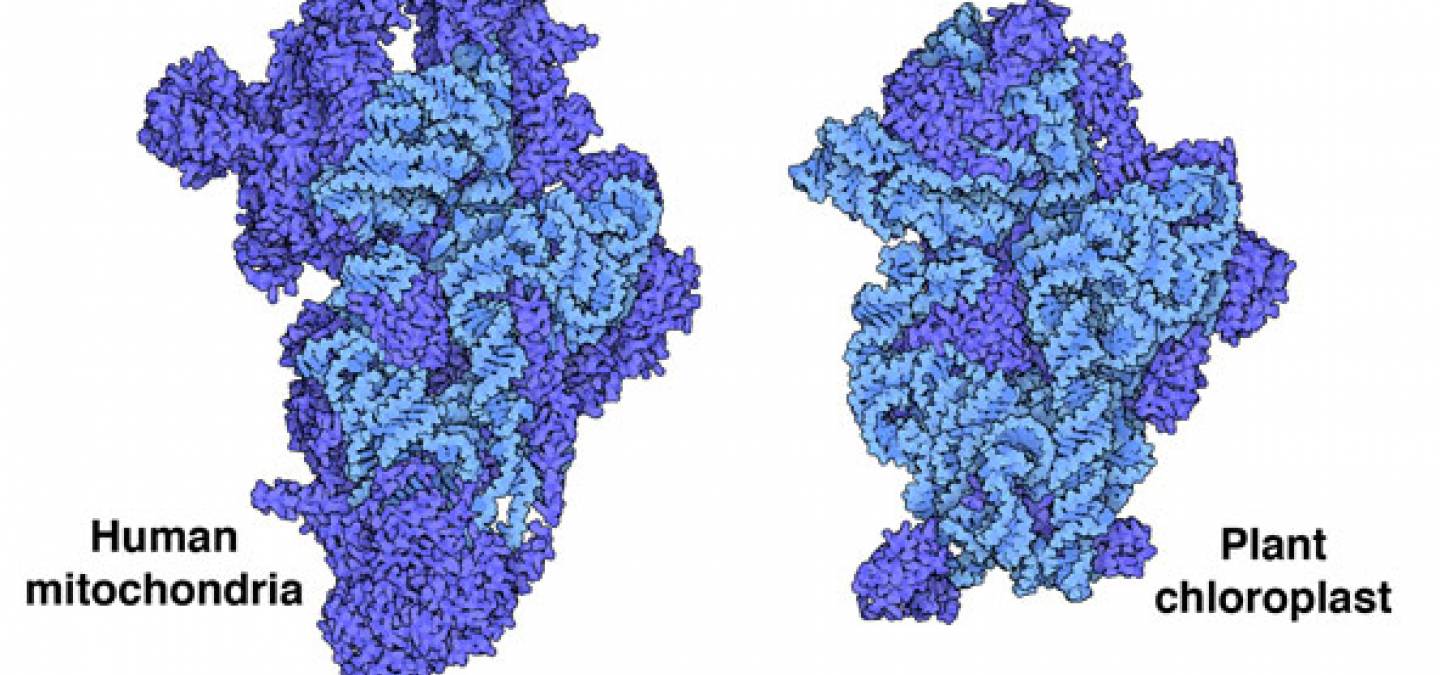
Les mitochondries et les chloroplastes fabriquent leurs propres ribosomes, qui diffèrent de ceux présents dans le cytosol des plantes, des animaux et des champignons. On pense que ces deux organites ont évolué à partir de bactéries qui sont devenues des organites symbiotiques dans les cytoplasmes des cellules à l’origine des cellules eucaryotes modernes. La structure des ribosomes du chloroplaste est l'un des éléments en faveur de cette théorie endosymbiotique. La petite sous-unité ribosomique du chloroplaste (entrée PDB 5mmj) est similaire à son homologue bactérienne, avec une courte chaîne d'ARN de 1491 nucléotides et 25 protéines 1. La structure de la petite sous-unité ribosomique mitochondriale (entrée PDB 6rw4) montre que les ribosomes mitochondriaux ont divergé de manière significative de leur ancêtre bactérien supposé, avec une chaîne d'ARN encore plus courte, de seulement 955 nucléotides, et davantage de protéines qu'un ribosome bactérien typique 2.
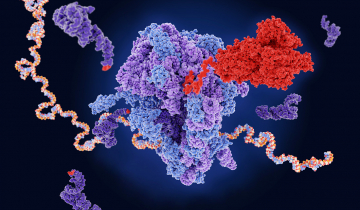
Complexité de l'initiation
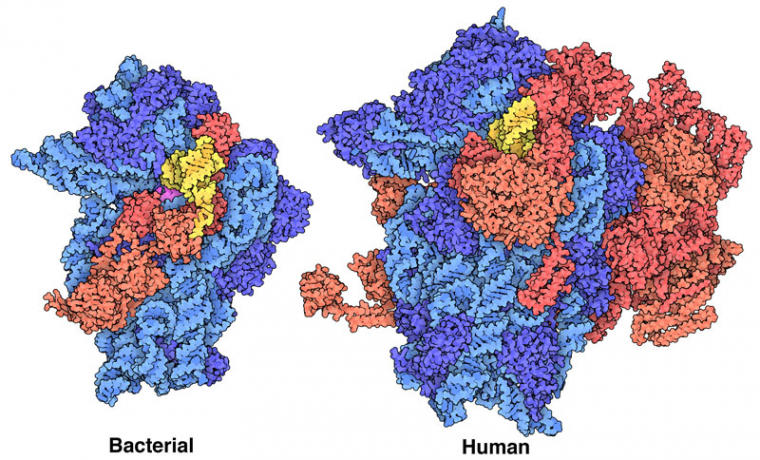
La complexité accrue des eucaryotes se manifeste également dans les nombreuses protéines qui assistent les ribosomes dans le processus de traduction. Par exemple, les deux structures représentées sur la figure 2 (entrées PDB 5lmv et 8oz0) montrent les protéines qui orchestrent l'initiation de la traduction. Ensemble, elles aident à capturer un ARN messager et à aligner un ARN de transfert initiateur sur le codon de départ de la traduction. Les cellules bactériennes n'utilisent que trois facteurs d'initiation pour accomplir cette tâche 3, mais les eucaryotes en recrutent environ 20 4.
ARN ribosomique
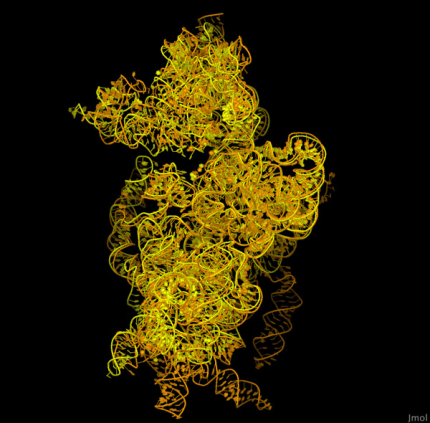
La figure 3 compare l'ARN ribosomique des petites sous-unités bactérienne et humaine (entrées PDB 1fjg et 5a2q). On remarque qu'il y a une partie centrale similaire pour les deux, qui accomplit les tâches de base en réunissant les ARN messagers et de transfert 1. L'ARN humain (en orange) comprend de nombreuses extensions autour de cette partie centrale, produisant une sous-unité ribosomique plus grande et plus complexe 2. Pour explorer ces structures plus en détail, vous pouvez utiliser par exemple le logiciel de visualisation de molécules disponible sur le site PDB-101.
Pour aller plus loin
- Des tendances similaires sont observées pour les grandes sous-unités ribosomiques. Par exemple, sur le site PDB-101, vous pouvez comparer la structure complète des ribosomes de la bactérie Escherichia coli (entrée PDB 4v4a) et de l'être humain (entrée PDB 4v6x).
- La comparaison des ARN présentés dans la figure 2 a été réalisée à l'aide du serveur en ligne gratuit RNA-align.
Ce texte correspond à la traduction par Cédric Bordi de l’article Molecule of the Month : Ribosome Diversity 1 écrit par David S. Goodsell et paru en juillet 2024 sur le site PDB-101, le portail éducatif de la base de données sur les protéines (PDB).