Depuis dix ans environ, l’identification des micro-organismes responsables de maladies infectieuses a été nettement améliorée par l’utilisation de la spectrométrie de masse de type MALDI-TOF (matrix-assisted laser desorption ionization-time of flight). Cette technique améliore la prise en charge des patients puisque les résultats sont connus environ 24 heures avant ceux obtenus par les méthodes biochimiques.
Contexte
L’identification des micro-organismes (bactéries, virus, champignons microscopiques et autres parasites eucaryotes) responsables d’infections est le rôle principal des laboratoires de microbiologie médicale. Cette documentation permet de faire le diagnostic d’infection et d’adapter le traitement anti-infectieux en fonction des résistances naturelles et acquises des micro-organismes isolés à partir des prélèvements des malades. Ces dernières années, les laboratoires de biologie médicale vivent une véritable révolution avec l’utilisation de milieux de culture de plus en plus performants (meilleure sensibilité dans la détection des bactéries, mise au point de milieux chromogènes1 et sélectifs permettant d’isoler un micro-organisme recherché), une importante informatisation et automatisation des tâches effectuées (pour la réalisation d’antibiogrammes ou, plus récemment, pour l’ensemencement des prélèvements) ainsi que le développement important des techniques de biologie moléculaire.
Pour l’identification des bactéries, les techniques conventionnelles utilisant les caractéristiques biochimiques des souches (galeries Api ou automates d’identification biochimique) ont été remplacées par la spectrométrie de masse de type MALDI-TOF (matrix-assisted laser desorption ionization-time of flight ; en français, désorption-ionisation laser assistée par matrice). Cette technologie est également déployée dans les laboratoires vétérinaires et certains laboratoires agroalimentaires (recherche et développement, contrôle qualité pour la recherche de pathogènes alimentaires).
Principe de la spectrométrie de masse
La spectrométrie de masse est une technique d’analyse physico-chimique permettant de détecter, d’identifier et de quantifier des molécules d’intérêt par mesure de leur masse. Son principe réside dans la séparation en phase gazeuse de molécules chargées (ions) en fonction de leur rapport masse/charge (m/z). Le spectromètre de masse se compose d’une chambre d’ionisation, d’un analyseur permettant la séparation des ions et d’un détecteur d’ions. Il existe de nombreux procédés d’ionisation et de séparation choisis en fonction de divers facteurs : volatilité, thermostabilité, capacité d’ionisation, taille, quantité et état physique (gaz, solide, liquide) des molécules à étudier.
Mise au point en 1912, la spectrométrie de masse a d’abord été utilisée pour la séparation de petites molécules avec de nombreuses applications (en chimie organique et inorganique par exemple). En 1975, une étude montre que cette technologie est potentiellement applicable à l’identification des bactéries [1]. Par la suite, l’optimisation de cette technique par réalisation d’une ionisation douce de l’échantillon (MALDI et ionisation par électronébulisation [electrospray en anglais]) a permis l’étude des macromolécules (jusqu’à 100 kDa) dont les protéines et a contribué à son essor dans les laboratoires de recherche [2]. Cette découverte a été honorée par le prix Nobel de chimie en 2002. Il a suivi une étape importante de standardisation (conditions expérimentales, conditions de culture) ayant permis d’améliorer la robustesse de la méthode et l’utilisation en routine pour l’identification des micro-organismes2 dans les laboratoires médicaux. Celle-ci est couramment employée dans les laboratoires de biologie médicale depuis une dizaine d’années.
Utilisation de la spectrométrie de masse MALDI-TOF en microbiologie médicale
Les prélèvements des patients sont généralement ensemencés sur divers milieux de culture dans différentes atmosphères (aérobie, anaérobie, 5 % CO2) à 35 °C pendant un à plusieurs jours.
L’identification MALDI-TOF est réalisée sur les colonies d’intérêt isolées à partir des milieux de culture. La colonie à identifier est prélevée à l’aide d’un cône et déposée sur une plaque en métal ou en plastique (comprenant plusieurs emplacements, appelés spots). Le dépôt, fin et régulier, est ensuite séché.
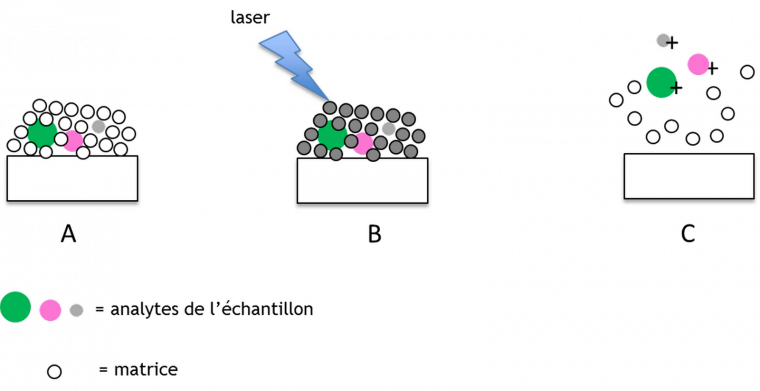
Une première étape cruciale et indispensable au procédé d’identification est celle de désorption/ionisation douce des protéines de l’échantillon. Cette étape, effectuée à l’aide d’une matrice et d’un laser se nomme MALDI (matrix-assisted laser desorption ionization). Une matrice photosensible (par exemple l’acide α-cyano-4-hydroxycinnamique) est reconstituée dans un mélange composé généralement d’eau, d’un solvant organique (acétonitrile) et d’acide trifluoroacétique. Elle est ensuite déposée sur le spot et va cocristalliser avec l’échantillon sous l’effet de l’évaporation. La plaque est introduite dans le spectromètre où un laser (généralement laser à azote de longueur d’onde 337 nm) va exciter les molécules de la matrice photosensible entraînant une ionisation généralement par transfert d’un proton depuis la matrice vers l’échantillon et une désorption (passage en phase gazeuse) de l’échantillon (Figure 1).
A. Cocristallisation de l’échantillon et d’une matrice photosensible sur la plaque MALDI.
B. Excitation des molécules photosensibles de la matrice par un laser.
C. Ionisation et désorption de l’échantillon.
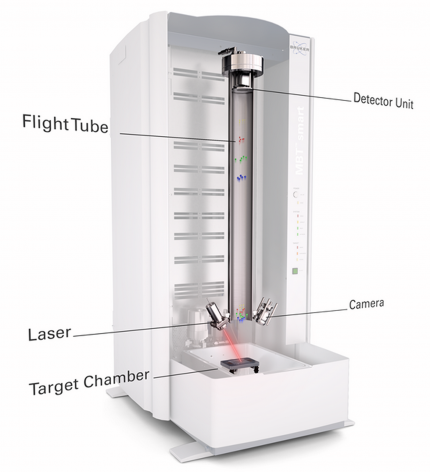
Le spectromètre de masse comprend une chambre d’introduction de la cible (target chamber), un laser, une caméra, un tube de vol permettant la migration des fragments protéiques ionisés (flight tube), un détecteur (detector unit). Les principaux spectromètres de masse actuellement utilisés sont les automates Microflex (Bruker, Allemagne) et Vitek-MS (BioMérieux, France).
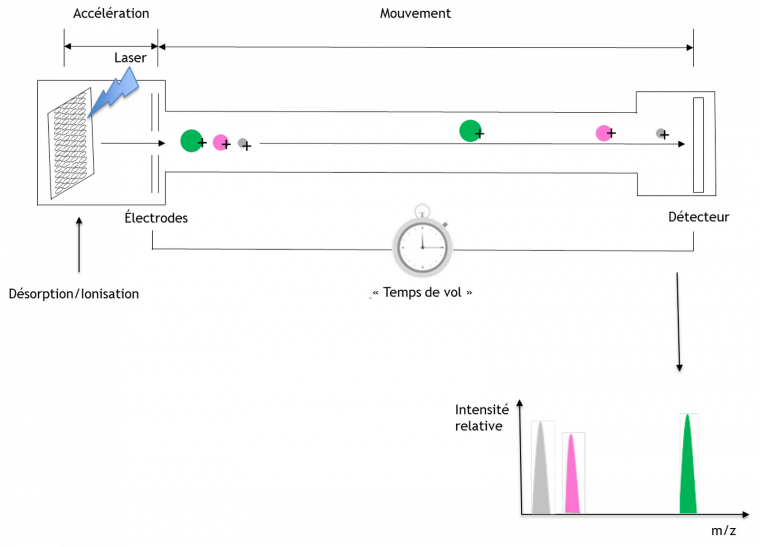
La technique d’ionisation MALDI est associée à la technologie TOF (time of flight en anglais ou temps de vol). Dans une zone d’accélération, une impulsion électrique (10 à 30 kiloélectronvolts) par application d’une différence de potentiel accélère les fragments ionisés jusqu’à l’entrée d’un tube de vol. Ceux-ci sont principalement mono-chargés et arrivent en même temps à l’entrée du tube de vol. Ils sont ensuite « lachés » dans le tube de vol dans un vide poussé (évitant les collisions avec l’air) et se déplacent jusqu’au niveau d’un détecteur (multiplicateur d’électrons). Le temps de vol de chaque fragment est déterminé. Plus la masse des fragments ionisés est élevée, plus le temps de vol est long. Le spectre obtenu correspond à un signal exprimé en temps de vol, qui est ensuite usuellement traduit en spectre de masse (m/z) (avec z = 1 dans le cas présent1). L’intensité des pics est proportionnelle au nombre d’ions détectés à un temps donné et donc également à la quantité de fragments protéiques ionisés du dépôt.
Relation fondamentale entre le rapport (m/z) et temps de vol t des analyseurs à TOF linéaires [3]
Au début de la phase d’accélération, les ions sont soumis à une différence de potentiel U et acquièrent une énergie correspondant principalement à une énergie potentielle Ep = zeU avec z = nombre de charge d’un ion (z = 1 pour les applications du MALDI-TOF en microbiologie), e = charge élémentaire de l’électron et U = tension totale.
À l’entrée du tube de vol, cette énergie potentielle est convertie en une énergie cinétique selon la relation :
Ec = zeU = ½ mv2
avec m = masse en kg, v = vitesse en m/s.
Le temps de vol t d’un ion dans le tube de vol de longueur L correspond à t = L/v
En remplaçant dans l’expression précédente, on obtient : m/z = (2eU/L2) t2
Ainsi, il est possible de convertir un temps de vol t en rapport masse/charge.
En haut : Les analytes ionisés présentent, en fonction de leur masse, des temps de vol différents (ou time of flight, TOF) dans le tube de vol. En bas : Les spectres des temps de vol sont traduits en spectre de masse/charge.
La catégorie de molécules ionisées dépend du type d’ionisation (positive ou négative), de la gamme de mesure de masse et surtout de la matrice utilisée. Par exemple, l’acide α-cyano-4-hydroxycinnamique utilisé dans l’identification des micro-organismes favorise très fortement l’ionisation des peptides et petites protéines bactériens (0,7 à 20 kDa). Certains spectromètres MALDI-TOF permettent également de travailler en ionisation négative avec utilisation d’une matrice différente (par exemple acide dihydroxybenzoïque) favorisant fortement l’ionisation et l’étude des lipides bactériens [4].
Dans le cadre de l’identification bactérienne, les protéines étudiées sont principalement des protéines ribosomales mais également d’autres protéines (cold shock proteins par exemple) [5]. Le spectre du micro-organisme d’intérêt est comparé aux spectres de référence d’une base de données selon un algorithme statistique, ce qui permet son identification. Par exemple, la base de données principale Biotyper version 8 (Bruker Daltonics) comprend 7712 spectres de référence de 2665 espèces différentes (bactéries et levures) permettant une identification facile de la quasi-totalité des espèces bactériennes impliquées en pathologie humaine. Le résultat est accompagné d’un score de confiance qui permet aux microbiologistes d’apprécier la fiabilité de l’identification du micro-organisme.
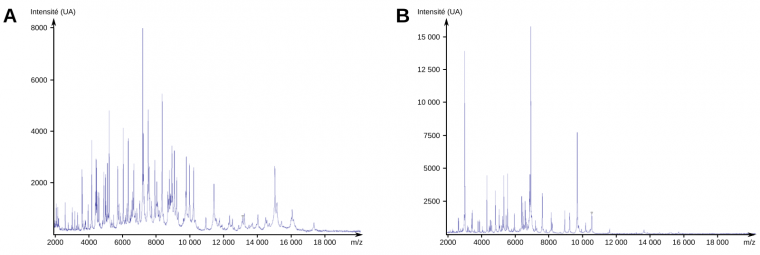
A. Spectre de Pseudomonas aeruginosa. B. Spectre de Staphylococcus aureus. Ces spectres ont été obtenus avec le MALDI-TOF Biotyper Microflex LT (Bruker). En ordonnées, l’intensité, exprimée en unité arbitraire, correspond à nombre d’ions détectés à un temps donné.
Lorsque l’identification ne donne pas un score satisfaisant ou dans le cas de micro-organismes ayant une paroi épaisse (par exemple pour les champignons microscopiques, qui ont une paroi composée de chitine et glucanes1), il peut être nécessaire de réaliser une extraction protéique soit directement sur la plaque MALDI-TOF ou en milieu liquide par mise en contact du micro-organisme à identifier avec, par exemple, de l’acide formique ou un mélange acide formique/acétonitrile entraînant une meilleure libération des protéines cellulaires.
Les spectres obtenus peuvent varier en fonction des conditions expérimentales et également parfois en fonction des sous-espèces ou des souches étudiées à l’intérieur d’une même espèce. Les conditions expérimentales influant sur la qualité des spectres sont le délai de culture, le milieu de culture utilisé, le type et la qualité du dépôt et/ou de l’extraction protéique et l’appareil MALDI-TOF utilisé [6,7]. L’utilisation de protocoles expérimentaux standardisés et la conception des bases de données de spectres de référence permettent de minimiser l’impact de ces facteurs sur les résultats d’identification. Les bases de données commercialisées sont construites avec des souches de collection et des souches identifiées au préalable par biologie moléculaire (séquençage de l’ADNr 16S et si besoin d’autres cibles : rpoB, sodA, recA). Les spectres de référence sont créés à partir de pics communs et reproductibles obtenus à partir de plusieurs passages des souches dans différentes conditions expérimentales. De plus, plusieurs souches d’une même espèce sont généralement représentées dans les bases de données. Pour une espèce donnée, le nombre de souches présentes dans les bases de données commercialisées dépend généralement de l’intérêt diagnostique que représente cette espèce, de sa diversité intraspécifique et de la disponibilité des différentes souches.
A. Prélèvement de la colonie bactérienne avec un cône
B. Dépôt de la colonie sur le spot de la plaque et ajout de la matrice sur chaque spot. La plaque comprend plusieurs spots permettant d’identifier séquentiellement plusieurs échantillons.
C. Insertion de la plaque dans le spectromètre de masse.
D. Lecture des résultats (ici, c’est l’espèce Staphylococcus aureus qui avait été prélevée en A) sur le poste de travail informatique associé au spectromètre et envoi informatisé des résultats au système informatique du laboratoire.
Avantages
L’intérêt majeur de la spectrométrie de masse est la diminution du temps d’identification des micro-organismes. Ce temps, réduit à moins d'une heure, est beaucoup plus court que celui nécessaire aux techniques biochimiques conventionnelles qui imposaient la croissance des micro-organismes en présence de divers substrats. L'utilisation de la spectrométrie de masse de type MALDI-TOF permet ainsi le rendu des résultats d’identification 24 h plus tôt et améliore la prise en charge des malades (modification de l’antibiothérapie, compréhension d’une pathologie infectieuse). Cet apport clinique est notamment démontré dans le rendu des résultats d’hémocultures (mises en culture de prélèvements sanguins en milieu liquide incubé à 35 °C). Celles-ci permettent de faire le diagnostic de bactériémie ou fongémie (présence de bactéries ou levures/champignons dans le sang, normalement stérile) à l’origine de sepsis ou de choc septique1, pathologies grevées d’une mortalité élevée [8]. La gravité clinique impose donc une prise en charge urgente des hémocultures. Lorsque celles-ci sont positives (flacon détecté positif par un automate pour hémocultures), il est possible d’identifier très rapidement la bactérie impliquée par MALDI-TOF et d’y associer des tests rapides de détection de résistance aux antibiotiques, générant une meilleure prise en charge des malades (antibiothérapie plus adaptée, moins d’hospitalisation en soins intensifs, baisse de la mortalité). L’impact clinique des tests rapides (identification, résistance) est généralement plus significatif lorsqu’il est associé à un conseil en antibiothérapie (par un médecin infectiologue par exemple) [9-11].
L’identification par MALDI-TOF présente d’autres avantages : elle ne prend que quelques minutes, est facile à mettre en œuvre et ne nécessite pas une formation longue des techniciens de laboratoire. De nombreux travaux montrent que cette technique présente de très bonnes performances d’identification par rapport aux techniques conventionnelles et permet même d’identifier des germes nécessitant sinon l’utilisation de techniques longues de biologie moléculaire. Par exemple, le MALDI-TOF remplace l’identification longue des mycobactéries habituellement réalisée par séquençage du gène hsp65 ou par des techniques d’hybridation de l’espace intergénique 16S-23S [12-14].
Les rares erreurs ou les absences d’identification sont en général attribuées au caractère incomplet des bases de données. Celles-ci sont cependant de plus en plus exhaustives car mises à jour régulièrement par les fabricants de spectromètres. Il est également possible d’alimenter les bases de données MALDI-TOF avec des spectres pour obtenir des bases de données « maison », ce qui peut présenter un intérêt diagnostique, par exemple pour identifier des pathogènes émergents et/ou non représentés dans la base de données [15,16]. De telles espèces doivent au préalable avoir été identifiées par séquençage de l’ADNr 16S ou d’autres cibles plus pertinentes en fonction de l’espèce bactérienne étudiée [17].
Le spectromètre de masse MALDI-TOF présente un coût d’achat non négligeable (155 000 à 170 000 euros hors taxes) mais cet investissement est finalement rentabilisé par un coût au test faible (15 à 30 centimes d’euros hors taxes par identification) [18].
Ces différents avantages font que la technique a rapidement été adoptée par les laboratoires de microbiologie pour une utilisation en routine.
Le diagnostic mycologique est également amélioré par l’utilisation de la technologie MALDI-TOF qui permet une identification précise des champignons, ce qui est fondamental pour utiliser les antifongiques adaptés. Cette technique supplante ainsi les examens microscopiques et macroscopiques, peu discriminants, ainsi que, dans de nombreux cas, l’identification par séquençage moléculaire, généralement long (séquençage des régions ITS de l’ADN ribosomal par exemple) [19,20]. Récemment, l’utilisation d’un nouveau milieu de culture nommé ID-FUNGI plate facilitant le prélèvement du champignon à partir de la gélose pour dépôt sur le spot de la plaque MALDI a amélioré les résultats d’identification des champignons [21,22].
Limites
Le principal désavantage de la technique est la nécessité d’avoir un inoculum bactérien ou fongique important à déposer sur le spot de la plaque MALDI-TOF. Cette nécessité exclut la réalisation possible d’un MALDI-TOF directement sur le prélèvement clinique à l’exception de certains prélèvements (urine par exemple) pouvant présenter d’emblée une charge bactérienne importante (>105 unités formant colonies/mL) [23]. Dans de nombreux cas, l’étape de mise en culture du prélèvement est donc toujours nécessaire.
Les performances analytiques, globalement satisfaisantes, dépendent toutefois de l’exhaustivité des bases de données qui sont mises à jour régulièrement. De plus, la technique ne différencie pas ou mal certaines espèces phylogénétiquement proches telles que Escherichia coli et Shigella spp, Streptococcus pneumoniae et Streptococcus mitis ou encore Mycobacterium intracellulare et Mycobacterium chimaera. Cependant des améliorations techniques des appareils ainsi que de l’interprétation des spectres pourraient permettre, dans certains cas, une analyse de plus en plus fine [24].
Perspectives
L’amélioration de l’exhaustivité des bases de données par les fabricants de spectromètres est continuelle. L’amélioration des derniers spectromètres disponibles vise également à l’obtention de cadences améliorées, ce qui est intéressant pour les grands laboratoires.
Pour certaines espèces bactériennes, cette technique pourrait permettre du typage de micro-organismes c’est-à-dire la reconnaissance, à l’intérieur d’une même espèce, des clones ayant une pathogénicité importante ou spécifique. Le typage permet également de comparer différents isolats retrouvés en clinique et/ou dans l’environnement dans le cadre d’études épidémiologiques. À ce jour, le typage par spectrométrie de masse MALDI-TOF doit être amélioré (discrimination, reproductibilité) afin de rendre possible une éventuelle utilisation en microbiologie médicale [25,26].
La spectrométrie de masse MALDI-TOF peut permettre également de détecter certaines résistances aux antibiotiques et antifongiques par des approches différentes : mise en évidence de profils de pics caractéristiques de sous-types bactériens résistants, détection de l’hydrolyse enzymatique d’un antibiotique ou détection de la croissance bactérienne en présence/absence d’antibiotique. Le délai de rendu varie entre une heure et quelques heures en fonction des techniques et permet l’obtention de résultats de résistance bien avant l’antibiogramme conventionnel [27-29]. À titre d’exemple, la détection de la dégradation enzymatique d’un antibiotique se réalise en incubant la bactérie à étudier en présence d’un antibiotique puis en observant par MALDI-TOF la disparition ou non du pic correspondant à l’antibiotique et l’apparition du pic correspondant au produit de dégradation de l’antibiotique. Des tests commercialisés utilisant cette approche permettent de détecter la résistance des entérobactéries aux bêta-lactamines (céfotaxime, imipénème) par production de bêta-lactamases. D’autres tests prometteurs sont en cours de développement [4]. À ce jour, les laboratoires utilisent souvent d’autres tests rapides disponibles (biologie moléculaire, méthodes immuno-chromatographiques ou colorimétriques) pour détecter de manière rapide la résistance bactérienne.
Depuis quelques années, la spectrométrie de masse de type MALDI-TOF est donc un outil indispensable pour l’identification des microorganismes dans les laboratoires de microbiologie médicale.
Références
- Anhalt J, Fenselau C. Identification of Bacteria Using Mass Spectrometry. Anal Chem 1975:219–25.
- Karas M, Hillenkamp F. Laser desorption ionization of proteins with molecular masses exceeding 10,000 daltons. Anal Chem 1988;60:2299–301. https://doi.org/10.1021/ac00171a028.
- Rouessac F, Rouessac A. Analyse chimique - Méthodes et techniques instrumentales modernes. 5ième édition. Dunod; 2000.
- Furniss RCD, Kostrzewa M, Mavridou DAI, Larrouy-Maumus G. The clue is in the lipid A: Rapid detection of colistin resistance. PLoS Pathog 2020;16:e1008331. https://doi.org/10.1371/journal.ppat.1008331.
- Ryzhov V, Fenselau C. Characterization of the protein subset desorbed by MALDI from whole bacterial cells. Anal Chem 2001;73:746–50. https://doi.org/10.1021/ac0008791.
- Carbonnelle E, Beretti J-L, Cottyn S, Quesne G, Berche P, Nassif X, et al. Rapid Identification of Staphylococci Isolated in Clinical Microbiology Laboratories by Matrix-Assisted Laser Desorption Ionization-Time of Flight Mass Spectrometry. J Clin Microbiol 2007;45:2156–61. https://doi.org/10.1128/JCM.02405-06.
- Walker J, Fox AJ, Edwards-Jones V, Gordon DB. Intact cell mass spectrometry (ICMS) used to type methicillin-resistant Staphylococcus aureus: media effects and inter-laboratory reproducibility. J Microbiol Methods 2002;48:117–26. https://doi.org/10.1016/s0167-7012(01)00316-5.
- Société Française de Microbiologie. Bactériémies et fongémies-hémocultures in: Rémic. Société Française de Microbiologie Ed ; 2018. n.d.
- Farfour E, Si Larbi AG, Cardot E, Limousin L, Mathonnet D, Cahen P, et al. Impact of rapid diagnostic tests on the management of patients presenting with Enterobacteriaceae bacteremia. Med Mal Infect 2019;49:202–7. https://doi.org/10.1016/j.medmal.2018.11.015.
- Clerc O, Prod’hom G, Vogne C, Bizzini A, Calandra T, Greub G. Impact of matrix-assisted laser desorption ionization time-of-flight mass spectrometry on the clinical management of patients with Gram-negative bacteremia: a prospective observational study. Clin Infect Dis Off Publ Infect Dis Soc Am 2013;56:1101–7. https://doi.org/10.1093/cid/cis1204.
- Osthoff M, Gürtler N, Bassetti S, Balestra G, Marsch S, Pargger H, et al. Impact of MALDI-TOF-MS-based identification directly from positive blood cultures on patient management: a controlled clinical trial. Clin Microbiol Infect Off Publ Eur Soc Clin Microbiol Infect Dis 2017;23:78–85. https://doi.org/10.1016/j.cmi.2016.08.009.
- Bille E, Dauphin B, Leto J, Bougnoux M-E, Beretti J-L, Lotz A, et al. MALDI-TOF MS Andromas strategy for the routine identification of bacteria, mycobacteria, yeasts, Aspergillus spp. and positive blood cultures. Clin Microbiol Infect Off Publ Eur Soc Clin Microbiol Infect Dis 2012;18:1117–25. https://doi.org/10.1111/j.1469-0691.2011.03688.x.
- Carbonnelle E, Nassif X. Utilisation en routine du MALDI-TOF-MS pour l’identification des pathogènes en microbiologie médicale. Médecine/Sciences 2011:882–8.
- Body BA, Beard MA, Slechta ES, Hanson KE, Barker AP, Babady NE, et al. Evaluation of the Vitek MS v3.0 Matrix-Assisted Laser Desorption Ionization-Time of Flight Mass Spectrometry System for Identification of Mycobacterium and Nocardia Species. J Clin Microbiol 2018;56. https://doi.org/10.1128/JCM.00237-18.
- Sitterlé E, Giraud S, Leto J, Bouchara JP, Rougeron A, Morio F, et al. Matrix-assisted laser desorption ionization-time of flight mass spectrometry for fast and accurate identification of Pseudallescheria/Scedosporium species. Clin Microbiol Infect Off Publ Eur Soc Clin Microbiol Infect Dis 2014;20:929–35. https://doi.org/10.1111/1469-0691.12574.
- Garrigos T, Neuwirth C, Chapuis A, Bador J, Amoureux L, Collaborators. Development of a database for the rapid and accurate routine identification of Achromobacter species by matrix-assisted laser desorption/ionization-time-of-flight mass spectrometry (MALDI-TOF MS). Clin Microbiol Infect Off Publ Eur Soc Clin Microbiol Infect Dis 2020. https://doi.org/10.1016/j.cmi.2020.03.031.
- Woo PCY, Lau SKP, Teng JLL, Tse H, Yuen K-Y. Then and now: use of 16S rDNA gene sequencing for bacterial identification and discovery of novel bacteria in clinical microbiology laboratories. Clin Microbiol Infect Off Publ Eur Soc Clin Microbiol Infect Dis 2008;14:908–34. https://doi.org/10.1111/j.1469-0691.2008.02070.x.
- Tran A, Alby K, Kerr A, Jones M, Gilligan PH. Cost Savings Realized by Implementation of Routine Microbiological Identification by Matrix-Assisted Laser Desorption Ionization-Time of Flight Mass Spectrometry. J Clin Microbiol 2015;53:2473–9. https://doi.org/10.1128/JCM.00833-15.
- Sanguinetti M, Posteraro B. Identification of Molds by Matrix-Assisted Laser Desorption Ionization-Time of Flight Mass Spectrometry. J Clin Microbiol 2017;55:369–79. https://doi.org/10.1128/JCM.01640-16.
- Schoch CL, Seifert KA, Huhndorf S, Robert V, Spouge JL, Levesque CA, et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi. Proc Natl Acad Sci U S A 2012;109:6241–6. https://doi.org/10.1073/pnas.1117018109.
- Robert MG, Romero C, Dard C, Garnaud C, Cognet O, Girard T, et al. Evaluation of ID-Fungi-PlatesTM media for identification of molds by MALDI-BiotyperTM. J Clin Microbiol 2020. https://doi.org/10.1128/JCM.01687-19.
- Cardot Martin E, Renaux C, Catherinot E, Limousin L, Couderc LJ, Vasse M. Rapid identification of fungi from respiratory samples by Bruker Biotyper matrix-assisted laser desorption/ionisation time-of-flight using ID-FUNGI plates. Eur J Clin Microbiol Infect Dis Off Publ Eur Soc Clin Microbiol 2020. https://doi.org/10.1007/s10096-020-04007-2.
- Ferreira L, Sánchez-Juanes F, González-Avila M, Cembrero-Fuciños D, Herrero-Hernández A, González-Buitrago JM, et al. Direct identification of urinary tract pathogens from urine samples by matrix-assisted laser desorption ionization-time of flight mass spectrometry. J Clin Microbiol 2010;48:2110–5. https://doi.org/10.1128/JCM.02215-09.
- Epperson LE, Timke M, Hasan NA, Godo P, Durbin D, Helstrom NK, et al. Evaluation of a Novel MALDI Biotyper Algorithm to Distinguish Mycobacterium intracellulare From Mycobacterium chimaera. Front Microbiol 2018;9:3140. https://doi.org/10.3389/fmicb.2018.03140.
- Spinali S, van Belkum A, Goering RV, Girard V, Welker M, Van Nuenen M, et al. Microbial typing by matrix-assisted laser desorption ionization-time of flight mass spectrometry: do we need guidance for data interpretation? J Clin Microbiol 2015;53:760–5. https://doi.org/10.1128/JCM.01635-14.
- Sauget M, Valot B, Bertrand X, Hocquet D. Can MALDI-TOF Mass Spectrometry Reasonably Type Bacteria? Trends Microbiol 2017;25:447–55. https://doi.org/10.1016/j.tim.2016.12.006.
- Oviaño M, Bou G. Matrix-Assisted Laser Desorption Ionization-Time of Flight Mass Spectrometry for the Rapid Detection of Antimicrobial Resistance Mechanisms and Beyond. Clin Microbiol Rev 2019;32. https://doi.org/10.1128/CMR.00037-18.
- Florio W, Tavanti A, Ghelardi E, Lupetti A. MALDI-TOF MS Applications to the Detection of Antifungal Resistance: State of the Art and Future Perspectives. Front Microbiol 2018;9. https://doi.org/10.3389/fmicb.2018.02577.
- Dortet L, Tandé D, de Briel D, Bernabeu S, Lasserre C, Gregorowicz G, et al. MALDI-TOF for the rapid detection of carbapenemase-producing Enterobacteriaceae: comparison of the commercialized MBT STAR®-Carba IVD Kit with two in-house MALDI-TOF techniques and the RAPIDEC® CARBA NP. J Antimicrob Chemother 2018;73:2352–9. https://doi.org/10.1093/jac/dky209.