Les ARN messagers possèdent un codon stop, qui déclenche l'arrêt de la traduction. Cependant, la transcription erronée d'un gène peut mener à l'apparition d'un codon stop prématuré, situé bien avant le codon stop naturel. Comment les cellules peuvent-elles différencier ces deux types de codon stop ?
Les gènes codants contiennent l’information nécessaire pour qu’une cellule produise ses propres protéines. L’ARN messager issu de la transcription de l’ADN est lu par les ribosomes, qui traduisent cette séquence de nucléotides en une séquence d’acides aminés qui constitue la protéine. Chaque étape de ce processus est finement contrôlée : l’activation ou la répression de la transcription, la modification, la localisation, la traduction et la dégradation des ARN messagers, et la modification et dégradation des protéines.
L’épissage et les protéines de liaison à l’ARN
L’épissage est un mécanisme important permettant les modifications des ARN prémessagers. En effet, les gènes eucaryotes sont divisés en parties codantes (les exons) et non codantes (les introns). Pendant l’épissage, la machinerie du complexe d’épissage (spliceosome) reconnaît les bornes qui séparent les exons des introns et déclenche une série de réactions pour exciser les introns et relier les exons. Ceci permet d’avoir un ARN messager mature avec une séquence codante continue prête à être lue par le ribosome.
a. Contrairement à la représentation caricaturale qui peut en être faite dans beaucoup d’illustrations, les ARN messagers nus n'existent pas. b. Dans les cellules, les ARNm sont coiffés (point bleu foncé), polyadénylés (AAAA(n)) et associés à de nombreuses protéines (formes colorées)
Dès sa synthèse, l’ARN messager ne diffuse pas aléatoirement dans la cellule sous forme d’une chaîne « nue » de nucléotides. Il est très rapidement couvert de protéines de liaison à l’ARN, l’ensemble constituant alors une particule ribonucléoprotéique . Ces protéines ont un rôle central pendant et après l’épissage, depuis l’exportation du noyau et la traduction des ARN messagers, jusqu’à leur dégradation. Leur importance est mise en évidence par leur taux d’expression, qui peut atteindre environ 20 % de la totalité des gènes exprimés [1].
Le complexe de la jonction exonique, une alarme moléculaire
Dans cette représentation on observe l'ARN messager couvert par des protéines de liaison à l'ARN : le complexe de liaison à la coiffe, à l'extrémité gauche, les protéines de liaison à la queue poly-A, à l'extrémité droite, et le complexe de la jonction exonique sur les jonctions exon-exon.
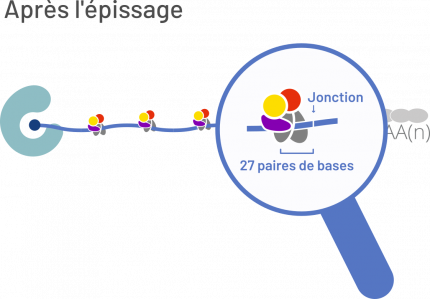
Parmi les protéines de liaison à l’ARN messager, se trouve le complexe de la jonction exonique (EJC en anglais). Il est composé d’un noyau de quatre protéines ainsi que de facteurs périphériques qui ont une présence dynamique pendant la vie de l’ARN [1]. Après l’épissage, ce complexe est déposé sur les ARN messagers à une distance d’environ 27 nucléotides en amont de la jonction entre deux exons. Il accompagnera le transcrit pendant son exportation au cytoplasme et sa localisation dans la cellule. Les composants du complexe seront enlevés lors de la traduction par le ribosome pour ensuite être recyclés et incorporés sur d’autres transcrits.
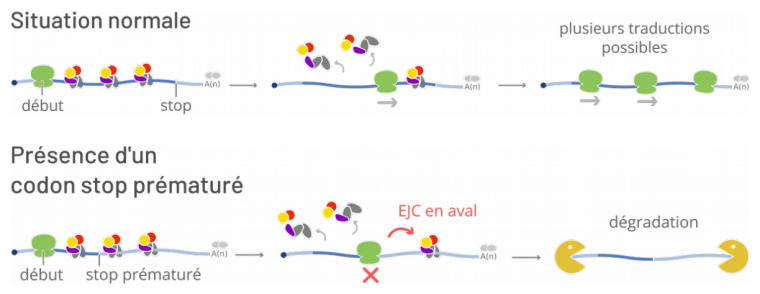
La découverte du complexe de la jonction exonique a été publiée en 2000 suite à des recherches dans le domaine de la régulation post-transcriptionnelle de l’expression génétique [2]. La mise en place du complexe après l’épissage met en évidence que celui-ci a eu lieu entre deux exons qui sont, pour la plupart1, codants. Or, la transcription n’est pas un processus parfait : des erreurs dans le point d’initiation, par exemple, peuvent décaler la phase de lecture du gène, ce qui génère un décalage de la séquence qui va être lue par le ribosome. Ceci produit non seulement une protéine incorrecte, mais peut aussi faire apparaître des codons stop prématurés, qui induisent l’arrêt de la traduction et le blocage du ribosome sur le transcrit.
Comment distinguer ce phénomène d’un arrêt normal de la traduction, et déclencher l’élimination des transcrits incorrects ? Dans une traduction normale, sans codon stop prématuré, la totalité des EJC déposés sur le transcrit sont enlevés par le ribosome. Par contre, dans le cas d’un transcrit possédant un codon stop prématuré, les EJC déposés sur des jonctions en aval de ce codon stop ne seront pas enlevés par le ribosome. La présence d’un EJC après un ribosome bloqué sur un transcrit est le signal qui déclenche le déblocage du ribosome et la dégradation du transcrit. Ce processus s’appelle la dégradation des ARNm non-sens (nonsense-mediated decay, ou NMD), et joue un rôle très important chez les Eucaryotes, mais son déclenchement par l’EJC est spécifique aux Métazoaires.
La présence d’un codon stop prématuré est detectée par les EJC déposés en aval ; ceci permet la dégradation du transcrit erroné par des exonucléases (en jaune).
Depuis sa découverte, de nombreuses publications scientifiques ont caractérisé la composition ainsi que d’autres fonctions de l’EJC, tels que l’exportation du transcrit depuis le noyau, la reconnaissance des exons, et l’augmentation de la traduction. Cependant, des questions restent ouvertes. Comment font les Eucaryotes qui ne possèdent pas d’EJC pour déclencher la dégradation par non-sens ? Quelle est la dose nécessaire d’EJC par transcrit pour garantir le contrôle de son expression ? Est-ce que tous les transcrits ont des EJC, et sur toutes les jonctions ? Est-ce qu’il y a des règles qui déterminent la mise en place (ou non) de l’EJC sur certaines jonctions ? Aujourd’hui, le laboratoire Expression des ARN messagers Eucaryotes, dirigé par le Dr Hervé Le Hir à l’École normale supérieure de Paris, essaye d’y répondre en utilisant des techniques de biochimie, de séquençage d’ADN à haut débit, et d’imagerie microscopique.
Références
- Gerstberger, S., Hafner, M. & Tuschl, T. A census of human RNA-binding proteins. Nat. Rev. Genet. 15, 829–845 (2014).
- Le Hir, H., Saulière, J. & Wang, Z. The exon junction complex as a node of post-transcriptional networks. Nat. Rev. Mol. Cell Biol. 17, 41–54 (2015).