Des complexes naturels de grande taille composés exclusivement d’ARN.
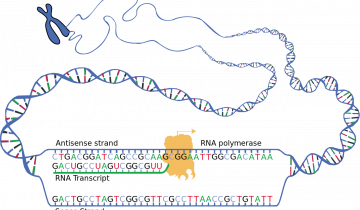
Dans le dogme central de la biologie moléculaire, l’ARN joue un rôle clé en tant que messager entre l’ADN présent dans le noyau et la biosynthèse des protéines dans le cytoplasme. Cependant, chez les êtres humains, seuls 1 à 2 % de tous les ARN transcrits codent des protéines ; la majorité des ARN ne sont donc pas traduits ou sont non codants. Il a été découvert que les ARN non codants jouent un rôle clé dans un large éventail de processus cellulaires fondamentaux, notamment le contrôle de la transcription, l’épissage et la traduction. Ces ARN possèdent souvent des structures secondaires et tertiaires complexes qui sont essentielles à leur fonction. Par exemple, les ARN ribosomiques, ou ARNr, sont de longs ARN non codants qui forment la partie catalytique des ribosomes. Dans de nombreux cas, comme dans les ribosomes, les tâches biologiques sont réalisées par l’association d’ARN à des protéines.
Les structures complexes des ARN
Comment identifier de nouvelles classes d’ARN non codants ayant des fonctions biologiques potentiellement intéressantes ? Les bioinformaticiens ont utilisé des approches de génomique comparative pour découvrir des séquences susceptibles d’avoir une structure secondaire conservée entre les espèces. Les chercheurs du laboratoire de Ronald Breaker, à l’université de Yale, ont utilisé cette stratégie pour découvrir de nouvelles classes d’ARN non codants dans les génomes de bactéries et de bactériophages 1. Parmi les ARN découverts figurait une classe d’ARN inhabituellement grands et avec une structure particulière provenant de bactéries extrémophiles à Gram positif. Ceux-ci ont été nommés ARN OLE (pour Ornate Large Extremophilic en anglais) 2. Une approche similaire a été utilisée pour identifier les ARN ROOL (pour Rumen-Originating, Ornate, Large en anglais) provenant de populations bactériennes vivant dans la panse des bovins et les ARN GOLLD (pour Giant, Ornate, Lake – and Lactobacillales-Derived en anglais) provenant d’échantillons bactériens prélevés dans le lac Gatún au Panama.
Récemment, plusieurs études structurales utilisant des techniques de cryomicroscopie électronique ont montré que ces ARN forment des structures complexes et ordonnées. Les ARN OLE provenant de Clostridium acetobutylicum (entrée de la base de données des protéines (PDB) 9mcw) et de Clostridium botulinum (entrée PDB 9lcr) se replient en dimères symétriques compacts avec une symétrie d’ordre 2 (représentés sur la figure 1 en rose) 3. La majeure partie de la structure est constituée d’une série d’hélices double brin qui apparaissent comme un faisceau serré de tiges.
Il est intéressant de noter que les ARN ROOL et GOLLD peuvent former des architectures creuses en forme de cage. Alors que certains ARN ROOL (représentés en bleu/violet sur la figure 1) forment principalement des cages octamériques (entrée PDB 9mds), d’autres ARN ROOL forment des complexes hexamériques (entrées PDB 9j6y, et 9m78) 1. L’ARN GOLLD (représenté en vert sur la figure 2) a également été observé avec des structures différentes, notamment un assemblage empilé en forme d’étoile constituée de 10 sous-unités d’ARN (entrée PDB 9lee) 2 ou bien des cages creuses constituées de 12 (entrée PDB 9l0r) ou 14 sous-unités d’ARN (entrée PDB 9mee) 3.
Des fonctions hypothétiques
L’importance biologique de ces structures exclusivement composées d’ARN est étayée par deux séries d’arguments. Premièrement, bien que la séquence primaire des ARN OLE, ROOL et GOLLD varie selon les espèces, les chercheurs ont remarqué une conservation des interfaces intermoléculaires et des motifs structurels. Le maintien de ces structures à l’échelle de l’évolution suggère que ces ARN jouent un rôle biologique important. Deuxièmement, des multimères stables d’ARN se forment à des concentrations physiologiques in vitro, ce qui suggère que des structures d’ordre supérieur composées uniquement d’ARN se formeront facilement à l’intérieur des cellules.
Cependant, les rôles précis des grandes structures exclusivement composées d’ARN sont encore largement inconnus. Compte tenu des structures creuses en forme de cage des complexes ROOL et GOLLD, les chercheurs suggèrent qu’ils pourraient agir comme des échafaudages structurels au sein de la cellule, aidant à séquestrer ou à compartimenter les composants cellulaires. On sait que les ARN OLE se lient à plusieurs partenaires protéiques différents et participent à divers processus dans les bactéries extrémophiles, notamment la réponse au stress et la régulation métabolique. Cependant, la manière dont les ARN OLE et leurs partenaires protéiques remplissent ces fonctions reste à découvrir.
Une collection de structures composées uniquement d’ARN
Vous pouvez examiner de plus près les structures complexes composées uniquement d’ARN formées par les ARN OLE (entrée PDB 9lcr), ROOL (entrée PDB 9mds) et GOLLD (entrée PDB 9mee) en vous rendant sur la page de l’article d’origine (en anglais), et en cliquant sur l’onglet JSmol de la section Exploring the structure.
Pour aller plus loin
- Découvrez les ARN auto-épissés dans un ancien article de la molécule du mois.
- Découvrez également d’anciens articles de la molécule du mois sur de grands complexes dont le cœur est constitué d’ARN enzymatiques non codants : les ribosomes et les épissosomes (spliceosomes).
Ce texte correspond à la traduction par Cédric Bordi de l’article Molecule of the Month : Natural RNA-Only Assemblies 1 écrit par Janet Iwasa en janvier 2026 sur le site PDB-101, le portail éducatif de la base de données sur les protéines (PDB).