Le bactériophage phiX174 produit une petite protéine qui tue les bactéries.
Les bactériophages sont des prédateurs naturels des bactéries. Un bactériophage se fixe à la surface d’une bactérie et y injecte son génome, qui est souvent composé d’un seul brin d’ADN. Ce génome dirige ensuite la construction d’un grand nombre de nouvelles copies du bactériophage, qui sortent de la cellule et la tuent. Les bactériophages sont omniprésents dans l’environnement et la plupart des bactéries sont attaquées par de nombreux types de bactériophages. Par exemple, deux bactériophages bien étudiés s’attaquent à la bactérie Escherichia coli : T4 et phiX174.
Franchir la paroi
Une fois que les bactériophages se sont multipliés à l’intérieur d’une cellule, ils sont confrontés à un défi : comment en sortir ? Les cellules bactériennes sont souvent entourées d’une couche de peptidoglycane, un réseau résistant de chaînes de protéines et de sucres. Les bactériophages ont donc besoin de protéines capables d’altérer cette enveloppe protectrice. Le bactériophage T4 utilise le lysozyme, une enzyme qui rompt les chaînes de peptidoglycane. Le bactériophage phiX174, quant à lui, fabrique une petite protéine, appelée protéine E, qui bloque la machinerie qui construit la gaine de peptidoglycane.
La protéine E
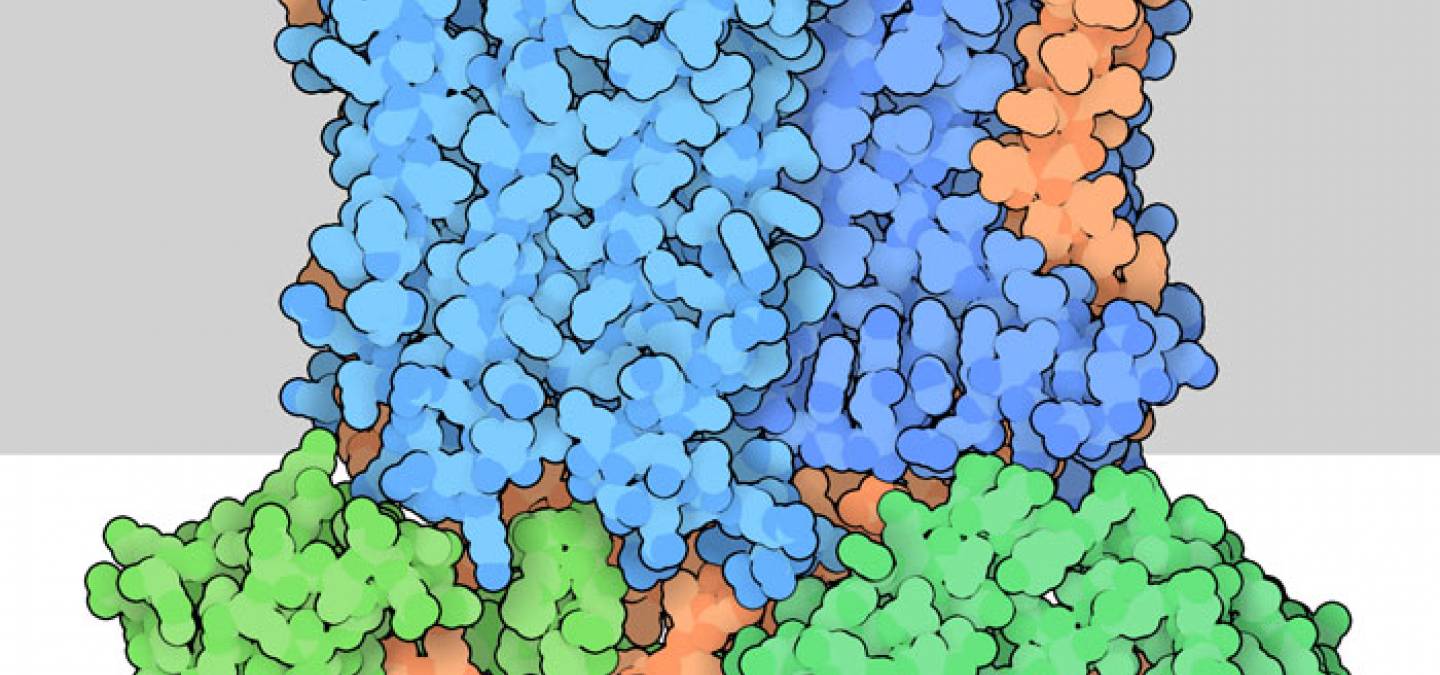
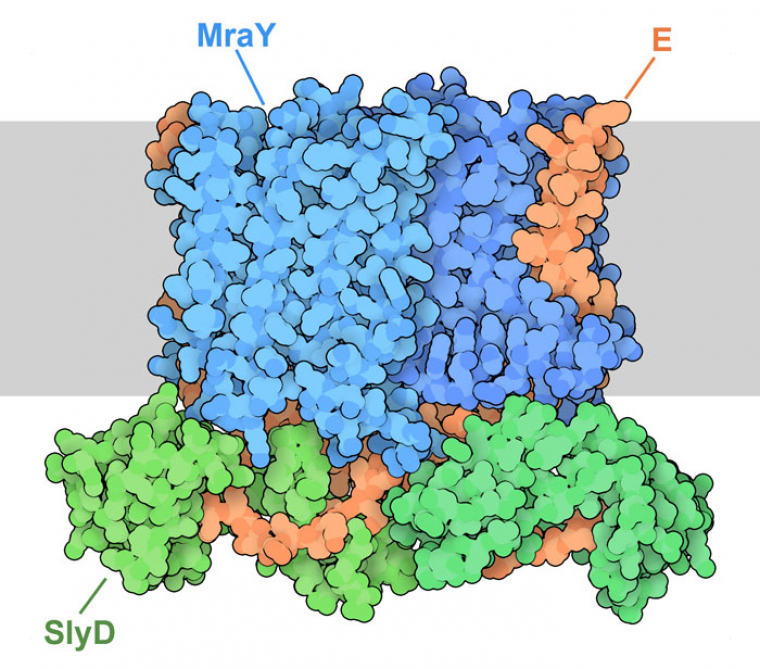
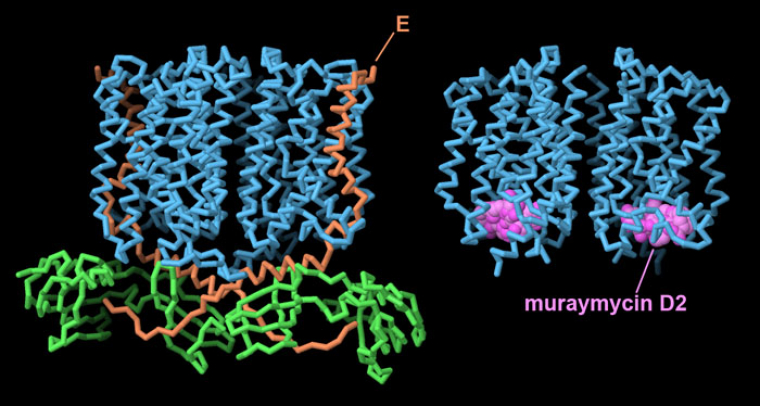
L’entrée 8g02 de la banque de données sur les protéines (PDB) révèle le mode d’action de la protéine E (Figure 1) 1. Elle se lie à l’enzyme bactérienne MraY, qui joue un rôle central dans la synthèse du peptidoglycane. MraY fixe de petits précurseurs du peptidoglycane à un support lipidique. Le lipide permet à d’autres protéines de déplacer facilement le précurseur à l’extérieur de la cellule et de l’attacher aux chaînes de peptidoglycane en cours d’élongation. La protéine E bloque ce processus en se liant à MraY et en recouvrant le site actif. La protéine E recrute également une deuxième protéine, la protéine chaperon SlyD, qui obstrue encore davantage le site actif de MraY. L’ensemble du complexe, comprenant MraY, la protéine E et SlyD, a été appelé le complexe YES.
La thérapie par les phages
Dans la mesure où les phages tuent les bactéries, ils peuvent être utiles pour traiter les infections qu’elles causent 2. La phagothérapie est utilisée depuis plus d’un siècle, avec le traitement précurseur de la dysenterie à l’aide de phages en 1919. Elle est tombée en désuétude dans la médecine occidentale après la découverte de la pénicilline et d’autres antibiotiques. Les antibiotiques présentent des avantages majeurs : ils sont faciles à administrer et ciblent souvent un large éventail de bactéries3. Cependant, l’émergence récente de souches bactériennes résistantes aux antibiotiques a ravivé l’intérêt pour la thérapie par les phages, qui peut constituer une alternative efficace pour les infections pour lesquelles il n’existe pas d’antibiotiques efficaces.
L’injection d’ADN par les bactériophages
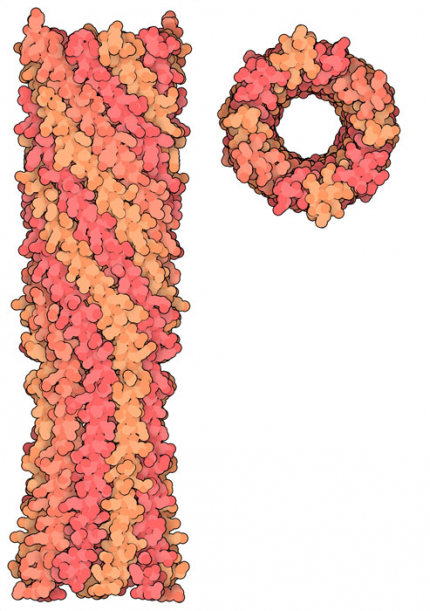
Les parois cellulaires bactériennes posent également des problèmes lors des premières étapes de l’infection par les bactériophages. De nombreux bactériophages, tels que T4, ont des queues élaborées qui se lient à la surface des bactéries et injectent leur ADN à travers la paroi. PhiX174 adopte une approche plus simple. Il code une protéine qui forme un tube à la dernière minute, construisant ainsi, à travers la paroi cellulaire, un chemin juste assez grand pour l’ADN. L’entrée PDB 4jpp montre la partie de la protéine qui forme le tube 1.
Le complexe YES et les antibiotiques
De nombreux antibiotiques s’attaquent aux enzymes bactériennes qui construisent la paroi de peptidoglycane. Par exemple, la pénicilline bloque les enzymes qui relient les précurseurs entre eux pour former le réseau. L’antibiotique naturel muraymycine D2 se lie à MraY (5ckr), bloquant le site actif de manière similaire à la protéine E de phiX174 1.
Pour aller plus loin
Le site PDB-101 propose :
- un court article de synthèse sur la biologie structurale des virus ;
- une sélection de ressources sur la résistance aux antibiotiques ;
- une sélection de ressources sur les virus.
Ce texte correspond à la traduction par Pascal Combemorel de l’article Molecule of the Month: PhiX174 YES Complex 1 écrit par David S. Goodsell et paru en avril 2024 sur le site PDB-101, le portail éducatif de la base de données sur les protéines (PDB). Les notes de bas de page sont du fait du traducteur.