Dans un court article paru dans Cell Genomics, trois chercheurs rappellent que les exons ne sont pas forcément des séquences traduites en protéines et mettent en garde contre l’emploi de l’expression « séquençage de l’exome » pour désigner une technique qui séquence surtout les exons codants.
Alors que certains ouvrages universitaires définissent les exons comme des séquences dont la traduction aboutit à la formation d’une protéine (en particulier « le » Alberts 1 et « le » Stryer 2), Julie Aspden et ses collaborateurs rappellent dans un court article que le terme exon désigne en réalité une séquence d’ARN toujours présente après l’épissage, contrairement aux introns 3. Le terme d’exon désigne aussi bien cette séquence sur l’ARN que son équivalent dans l’ADN.
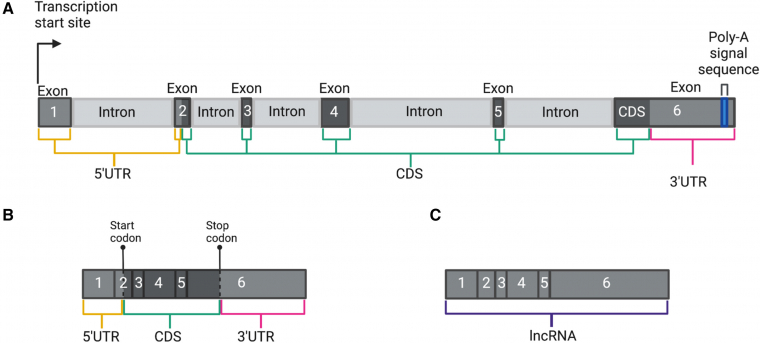
Les exons contenus dans une molécule d’ARN messager correspondent donc aussi bien aux séquences traduites en protéine qu’aux séquences non traduites qui la flanquent en 5’ (5’UTR, pour untranslated regions) et en 3’ (3’UTR). Par ailleurs les ARN non codants (ARNt, ARNr, etc.) sont également formés d’exons (Figure 1). Ainsi, chez l’être humain, seuls 23 % des exons sont codants tandis que 32 % correspondent à des extrémités 5’ et 3’ non traduites et 37 % à des ARN non codants.
A. Organisation d'une portion d'ADN codant un ARN codant lui-même une protéine. Les exons 3, 4 et 5 sont constitués exclusivement de séquences codantes, l'exon 1 est entièrement situé dans la région 5'UTR, les exons 2 et 6 contiennent à la fois une partie codante et une partie non codante.
B. ARNm obtenu après épissage du transcrit produit à partir de la séquence d'ADN représentée en A.
C. Un long ARN non codant, également formé de six exons, tous non codants.
5'UTR : extrémité 5' non traduite ; CDS : séquence codante ; 3'UTR : extrémité 3' non traduite ; lncRNA : long ARN non codant.
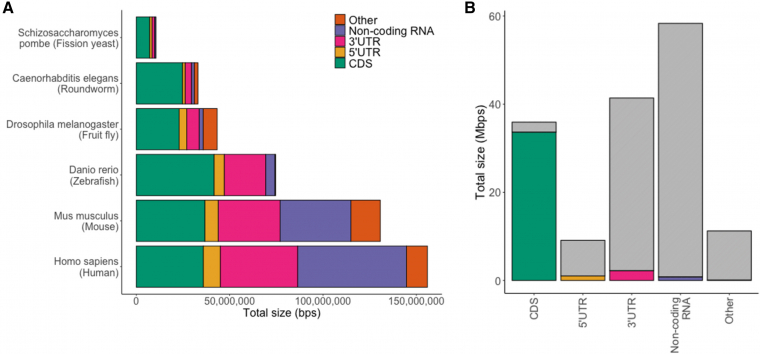
Par ailleurs, les auteurs insistent sur la dénomination trompeuse donnée à la technique de séquençage de l’exome (ou séquençage de l’exome entier, de l’anglais whole-exome sequencing) qui, contrairement à ce que son nom suggère, séquence surtout les exons codants au détriment des exons non codants. C’est ainsi que 89 % des bases séquencées par séquençage de l’exome correspondent à des séquences codantes, ce qui mène à une importante sous-représentation des extrémités non traduites et des ARN non codants dans les bases de données issues des techniques de séquençage de l’exome (Figure 2). Pour désigner la technique consistant à séquencer essentiellement les transcrits codants, et éviter ainsi toute ambiguïté, les auteurs proposent d’employer l’expression de séquençage de l’exome codant (CES, coding exome sequencing).
A. Comparaison, chez six organismes, de la proportion de bases situées dans des exons et annotées en tant que séquence codante (CDS), 5'UTR, 3'UTR, ARN non codant et autre (incluant les éléments transposables et les pseudogènes).
B. Histogramme indiquant, pour l'être humain, le nombre total de bases situées dans des exons et annotées de la même manière qu'en figure A. Les bases séquencées par les techniques de séquençage de l'exome sont en couleur, celles qui ne sont pas séquencées grâce à ces techniques sont en gris.
Sans nier l’intérêt de disposer d’informations sur les exons codants, les auteurs soulignent la nécessité de s’intéresser également aux exons non codants. En effet, les extrémités 5’ et 3’UTR sont indispensables à la stabilité, à la localisation et à la traduction correcte des ARNm. Ces extrémités contribuent également à la régulation de la traduction via l’interaction avec des protéines et des microARN. En outre les ARN non codants jouent également des rôles importants. Par exemple le microARN miR-204 est indispensable au fonctionnement des photorécepteurs de la rétine chez l’être humain, tandis que Xist, un long ARN non codant, est impliqué dans l’inactivation du chromosome X chez les Mammifères.