Parce que voir c’est croire, les scientifiques se sont mis en quête d’observer l’ADN en cours de réplication. Ils y sont parvenus grâce aux progrès de la microscopie et à l’utilisation de marqueurs radioactifs ou fluorescents, décodant peu à peu les mécanismes de la duplication des génomes. Désormais, on peut visualiser la réplication de l’ADN à l’aide d’un séquenceur. Retour sur les grands épisodes d’une histoire en marche.
Observation de molécules d’ADN en cours de réplication par autoradiographie
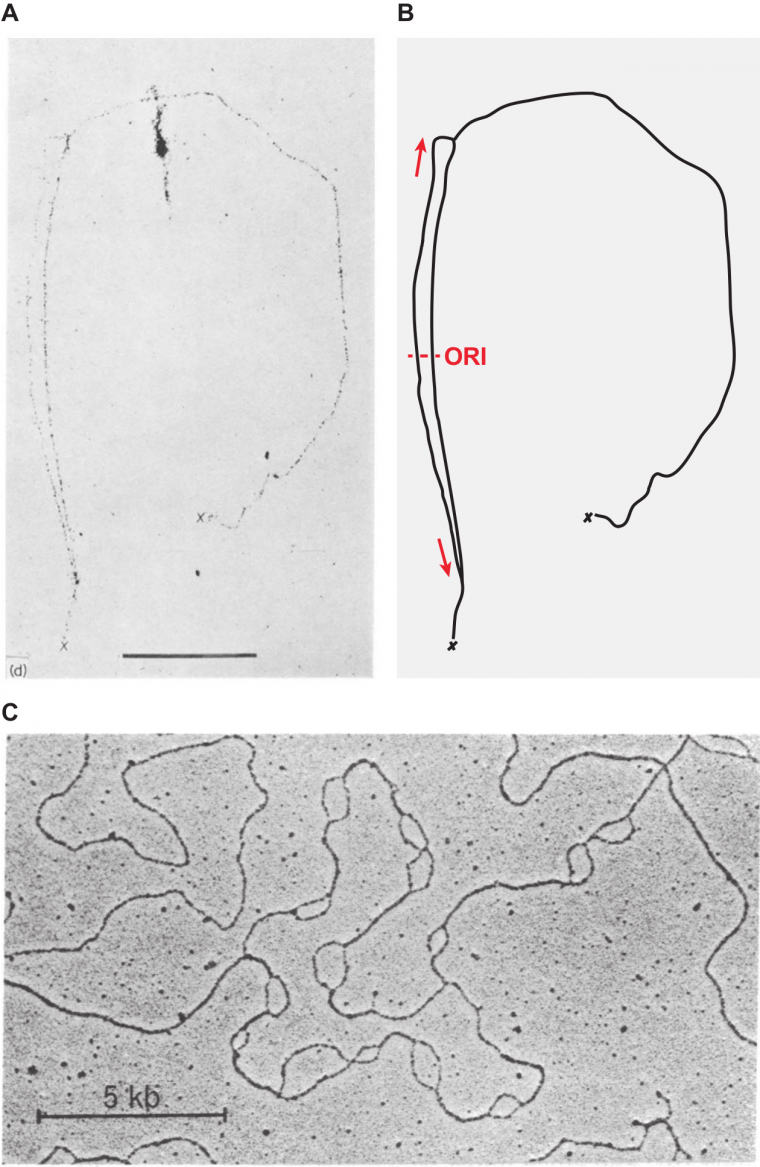
En 1963, le biologiste anglais John Cairns fut le premier à observer un chromosome en train de se dupliquer en utilisant la technique d’autoradiographie (1).Pour cela, Cairns a cultivé des bactéries Escherichia coli dans un milieu contenant de la thymidine marquée au tritium, un isotope radioactif de l’hydrogène, cette thymidine radioactive s’incorporant à l’ADN lors de la réplication. L’ADN extrait des bactéries a ensuite été collecté sur un support puis mis au contact d’un film photographique. Les électrons issus de la désintégration du tritium ont alors transformé les cristaux d’halogénures d’argent du film en argent métallique, opaque, faisant ainsi apparaître les molécules d’ADN en noir. La visualisation des films au microscope a non seulement permis d’observer le chromosome circulaire d’E. coli pour la toute première fois, mais également de le contempler en pleine réplication sous la forme de deux chromosomes entrelacés (Figure 1A). Cairns en a conclu que le chromosome bactérien était dupliqué au niveau d’un point de croissance en forme de « fourche », dont on comprendra plus tard qu’il correspond en fait à deux fourches de réplication divergentes issues de l’origine de réplication du chromosome d’E. coli (Figure 1B). Quelques années après, et toujours grâce à l’autoradiographie, Huberman et Riggs démontreront en effet que la réplication procède de manière bidirectionnelle à partir des origines (2). Ils confirmeront également qu’à la différence des chromosomes circulaires bactériens, les chromosomes linéaires des Eucaryotes sont répliqués à partir de multiples origines de réplication, comme cela avait déjà été observé, par John Cairns évidemment (3).
L’autoradiographie de molécules d’ADN a donc apporté des informations décisives sur la réplication tout en permettant d’en mesurer les paramètres essentiels, en particulier le nombre d’origines de réplication et la distance entre celles-ci. La vitesse de progression des fourches peut elle aussi être estimée par autoradiographie grâce à des protocoles de marquage de l’ADN néosynthétisé adaptés. L’approche développée par Cairns sera ainsi couramment utilisée pendant plusieurs décennies pour étudier les mécanismes cellulaires contrôlant la réplication de l’ADN dans un grand nombre d’organismes. D’autres méthodes ont néanmoins été développées en parallèle, plus confidentielles comme la microscopie électronique (4) (Figure 1C), ou plus indirectes comme l’électrophorèse bidimensionnelle en gel d’agarose (5,6).
(A, B) Visualisation par autoradiographie du chromosome circulaire d’E. coli marqué à la thymidine tritiée.
(A) Autoradiogramme original publié par John Cairns en 1963. La barre d’échelle correspond à 100 µm ; les croix signalent la position d’une cassure du chromosome.
(B) Schéma interprétatif. Les flèches rouges indiquent le sens de progression des fourches de réplication ; ORI marque la position approximative de l’origine de réplication du chromosome.
(C) Visualisation par microscopie électronique d’un chromosome de drosophile en cours de réplication. Les régions répliquées apparaissent sous la forme de « bulles » correspondant chacune à une paire de fourches de réplication divergentes.
Crédits images : (A) J. Cairns, 1963, Journal of Molecular Biology. Image reproduite avec autorisation. (B) B. Le Tallec, CC BY. (C) H.J. Kriegstein et D.S. Hogness, 1974, Proceedings of the National Academy of Sciences
Observation de molécules d’ADN en cours de réplication par immunofluorescence
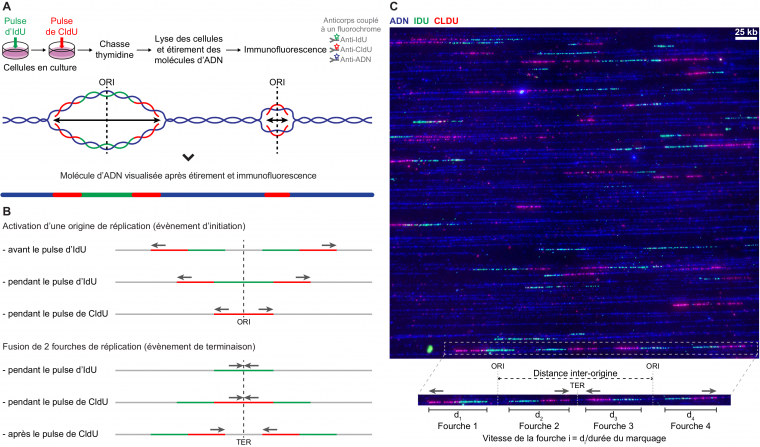
L’autoradiographie de molécules d’ADN a cédé sa place dans les années 90 à une version plus moderne utilisant des analogues de la thymidine en lieu et place de la thymidine tritiée pour marquer l’ADN en cours de synthèse. Une fois incorporés dans l’ADN, ces analogues (bromo, iodo ou chlorodésoxyuridine ; BrdU, IdU, CldU) sont révélés par immunofluorescence, c’est-à-dire qu’ils sont détectés à l’aide d’anticorps spécifiques couplés à des fluorochromes (Figure 2A). Outre le fait d’éviter de manipuler des composés radioactifs, leur usage présente l’avantage considérable de ne prendre que quelques heures pour visualiser l’ADN néosynthétisé, là où l’autoradiographie avec de la thymidine tritiée demande plusieurs mois d’exposition du film photographique pour détecter les signaux réplicatifs. Cette étape de révélation se fait sur des lamelles de verre sur lesquelles les molécules d’ADN, enchevêtrées en solution, ont au préalable été déroulées, rendant ainsi possible une analyse molécule par molécule. L’étirement se fait soit par simple force gravitationnelle (7), soit à l’aide d’un instrument déroulant les molécules d’ADN avec une force constante, ce qui augmente la précision des mesures – on parle dans ce dernier cas de peignage moléculaire de l’ADN (8), technique probablement la plus utilisée au cours des vingt dernières années.
Actuellement, l’analyse de la réplication sur des molécules d’ADN individuelles repose quasi-systématiquement sur un marquage consécutif des cellules avec deux analogues de la thymidine, par exemple l’IdU et le CldU (Figure 2A), ce qui permet d’orienter les fourches de réplication et de mesurer leur vitesse de progression, ainsi que d’identifier les événements d’initiation (progression de deux fourches divergentes issues d’une origine) et de terminaison (fusion de deux fourches convergentes) de la réplication (Figure 2B-C). Il est de plus possible d’étudier la réplication d’une région particulière en associant l’immunodétection de l’IdU et du CldU à l’hybridation de sondes d’ADN spécifiques du locus d’intérêt (9). Difficile à automatiser, ce type d'analyse exige de rechercher au microscope, sur l’ensemble de la lamelle de verre, les rares molécules combinant sondes et signaux réplicatifs, d'où un débit extrêmement faible : des mois de travail sont en général nécessaires pour recueillir les images de quelques centaines de fourches de réplication progressant au sein d’un locus donné.
(A) Schéma du protocole expérimental de double marquage réplicatif de cellules en culture et exemple de signal théorique obtenu. L’ADN néosynthétisé est marqué successivement par un pulse d’IdU (en vert) puis un pulse de CldU (en rouge), dont l’incorporation est stoppée par l’ajout de thymidine (« chasse thymidine »). L’ADN extrait des cellules est ensuite étiré sur une lamelle de verre sur laquelle on procède à l’immunodétection de l’IdU et du CldU à l’aide d’anticorps spécifiques couplés à des fluorochromes. La molécule d’ADN complète (en bleu) est elle aussi révélée à l’aide d’un anticorps spécifique. La lamelle est ensuite observée au microscope. Les molécules d’ADN apparaissent sous la forme de lignes, y compris au niveau des régions répliquées. Les deux molécules filles présentes au sein de la bulle de réplication sont en effet juxtaposées lors de l’étirement.
(B) Signaux réplicatifs obtenus. Le double marquage permet de reconnaître et d’orienter les fourches de réplication, ainsi que d’identifier les évènements d’initiation (progression de deux fourches divergentes suite à l’activation d’une origine) et de terminaison (fusion de deux fourches de réplication convergentes issues d’origines voisines) de la réplication. Le type de signal correspondant diffère selon que l’évènement a eu lieu avant, pendant ou après le pulse d’IdU ou de CldU.
(C) Exemple d’image acquise au microscope après immunodétection. Les molécules d’ADN en cours de réplication ont été extraites de fibroblastes embryonnaires de souris et étirées par peignage moléculaire. Cette technique permet un étirement régulier des molécules d’ADN (2 kb par micromètre) et donc une mesure précise des paramètres de la réplication, en particulier de la distance entre origines et de la vitesse de progression des fourches. ORI, origine de réplication ; TER, terminaison ; les flèches grises indiquent le sens de progression des fourches.
Observation de molécules d’ADN en cours de réplication, nouvelle génération
La principale limitation des méthodes actuelles est que les molécules en cours de réplication examinées restent généralement anonymes : il est possible de détecter une origine de réplication mais sans pouvoir la positionner sur le génome, ce qui limite la portée de l’observation. Localiser les segments d’ADN marqués offre au contraire l’opportunité de réellement cartographier la réplication de l’ADN, et de réaliser par exemple la carte des origines d’un génome donné.
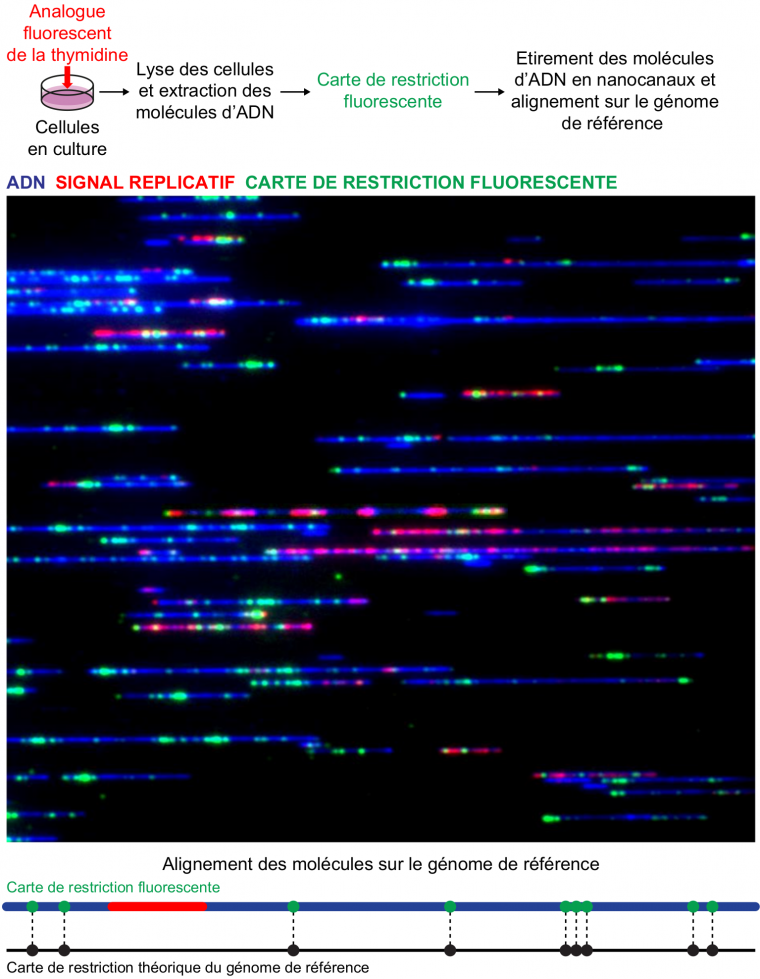
Deux nouvelles méthodes, toutes deux basées sur les nanotechnologies, ont été développées pour visualiser et identifier les molécules d’ADN en cours de réplication. La première, appelée cartographie optique de la réplication (10), est une évolution des techniques classiques. Elle combine (i) le marquage de l’ADN néosynthétisé par un analogue de la thymidine directement fluorescent1, ce qui dispense des étapes d’immunodétection, (ii) le marquage fluorescent des sites de coupure simple brin d’endonucléases spécifiques qui produit une carte de restriction optique pour chaque molécule et permet son alignement sur le génome de référence (iii) l’étirement contrôlé des molécules d’ADN dans des nanocanaux où elles sont imagées et positionnées automatiquement (Figure 3). Cette méthode offre un débit incommensurablement supérieur aux techniques conventionnelles mais, comme celles-ci, sa résolution maximale est de 1 à 2 kb d’ADN étiré en raison de la diffraction de la lumière qui limite la résolution des microscopes optiques à environ 0,2 µm.
L’ADN néosynthétisé est marqué par un analogue de la thymidine couplé à un fluorochrome (AF647-dUTP, en rouge). Après extraction, l’ADN est digéré par une nickase, c’est-à-dire par une enzyme de restriction coupant un seul des deux brins. Puis, des nucléotides fluorescents sont incorporés au niveau des sites de coupure simple brin, créant une carte de restriction fluorescente, ou « code-barre » optique (en vert) propre à chaque molécule. Les fibres d’ADN sont ensuite conduites par électrophorèse dans des nanocanaux où elles sont étirées de façon rectiligne par confinement, imagées automatiquement puis alignées sur le génome de référence grâce au « code-barre » optique, l'alignement étant basé sur la distance séparant les différents points lumineux. L’ADN est marqué au YOYO-1, un agent intercalant fluorescent (en bleu). Les molécules visualisées ici correspondent à de l’ADN de phage lambda.
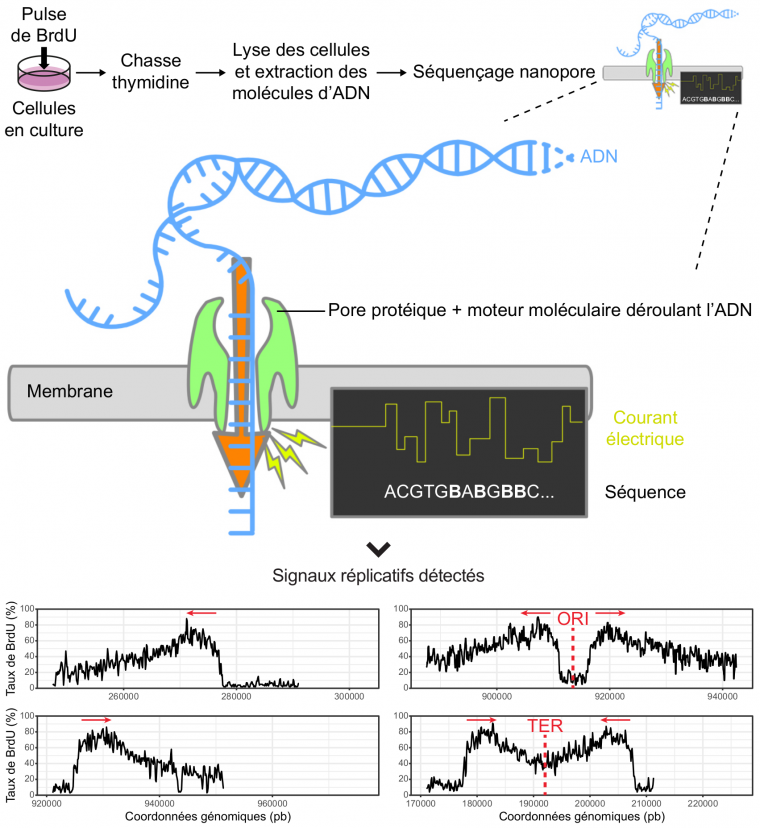
La deuxième approche est basée sur une technologie innovante de séquençage de l’ADN, le séquençage nanopore, qui consiste à faire passer une molécule d’ADN simple brin à travers un pore protéique inséré dans une membrane exposée à un champ électrique ; le passage de l’ADN induit une modification de l’intensité du courant qui dépend des bases présentes à l’intérieur du pore et qui peut donc être traduite en une séquence. Ce dispositif est capable de détecter des changements mineurs dans les bases d’ADN et ainsi d’identifier les analogues de la thymidine, par exemple le BrdU, incorporés pendant la réplication (11,12) (Figure 4). En permettant d’obtenir simultanément le signal réplicatif et la séquence de la molécule, la méthode nanopore simplifie considérablement la cartographie de la réplication par rapport aux méthodes optiques. La précision nucléotidique du séquençage repousse également les limites de résolution spatiale à laquelle la réplication de l’ADN peut être étudiée. Les paramètres mesurés sont les mêmes que pour les autres techniques (localisation des origines de réplication, vitesse de progression des fourches, etc), à ceci près que ceux-ci seront déterminés non plus visuellement mais grâce à des outils informatiques. À l’ère du numérique, observer des molécules d’ADN en cours de réplication ne demande plus un microscope, mais un ordinateur.
L’ADN néosynthétisé est marqué par un pulse de BrdU suivi d’une chasse thymidine. Lors du séquençage nanopore, les molécules sont déroulées en ADN simple brin et transloquées au travers de pores protéiques insérés dans une membrane soumise à un champ électrique. Les modifications du courant lors de la translocation sont enregistrées puis interprétées en séquence par un algorithme capable de détecter le BrdU (noté B) en plus des quatre nucléotides canoniques (A, T, C, G). Le taux de BrdU incorporé dans l’ADN est alors calculé tout au long des molécules séquencées. Lors de l’ajout de BrdU dans la culture, ce taux augmente rapidement, ce qui se traduit par une pente très forte marquant le début du pulse ; l’ajout de thymidine lors de la chasse dilue le BrdU et diminue son incorporation de façon plus progressive. L’asymétrie des pentes permet ainsi d’orienter les fourches de réplication et d’identifier les évènements d’initiation et de terminaison. On peut remarquer que la mesure du taux de BrdU est parasitée par des fluctuations de faible amplitude liées au processus de détection ; on parle de signal bruité. Les signaux réplicatifs présentés ont été détectés sur des molécules d’ADN extraites de la levure de boulanger, Saccharomyces cerevisiae. Les coordonnées génomiques sont indiquées sur l’axe des abscisses. ORI, origine de réplication ; TER, terminaison ; pb, paire de bases ; les flèches rouges indiquent le sens de progression des fourches.
Crédits image : B. Le Tallec et DataBase Center for Life Science, CC BY, Wikimedia (la séquence d’ADN a été modifiée et des annotations en français ont été ajoutées).
Observation des sites de synthèse d’ADN in situ
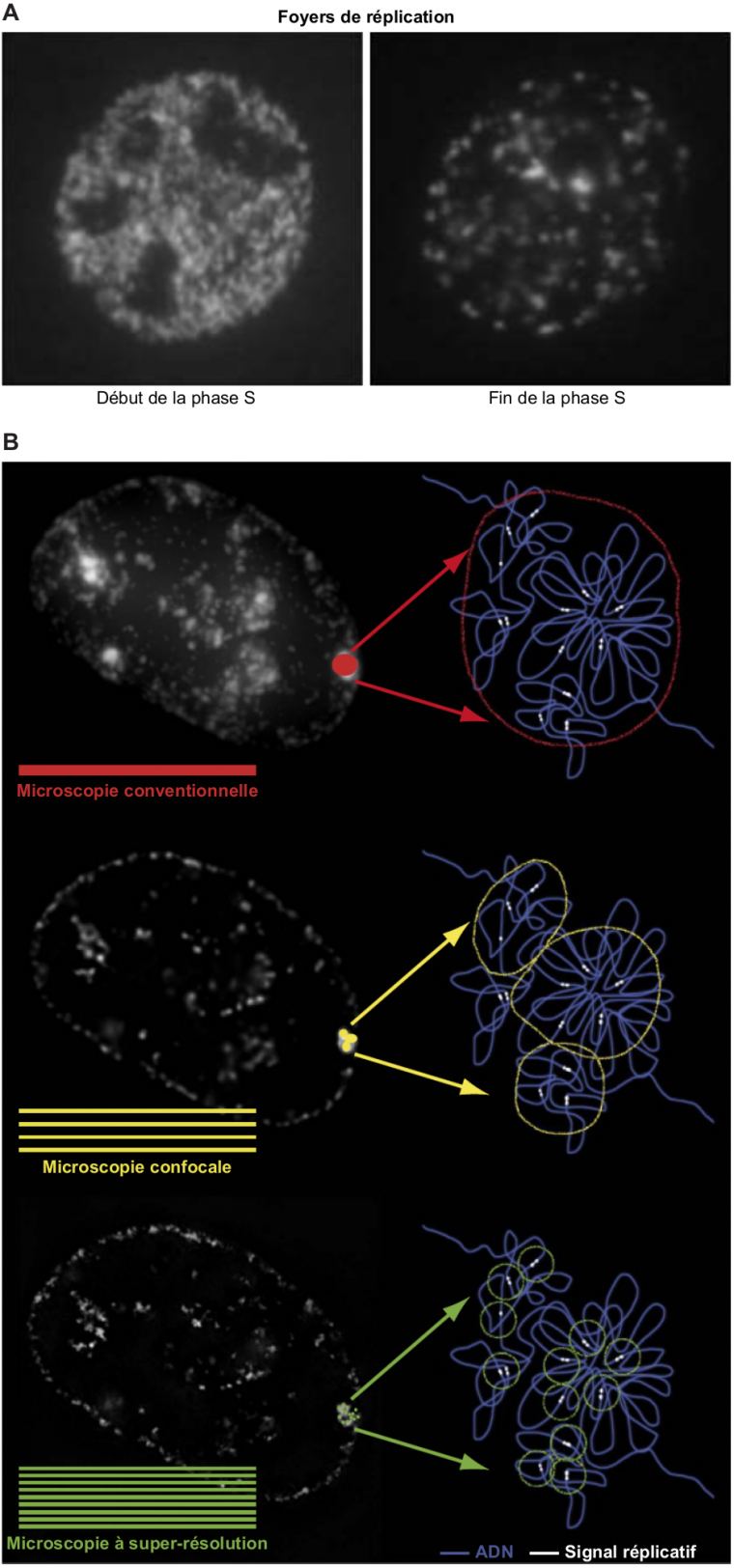
Pour toutes les techniques présentées ci-dessus, les observations sont réalisées sur des molécules d’ADN extraites des cellules. Il est aussi possible de visualiser les régions en cours de réplication au niveau cellulaire en détectant les sites d’incorporation d’analogues de la thymidine ou les composants de la machinerie de réplication par immunofluorescence in situ. L’analyse cytologique en microscopie conventionnelle des sites de synthèse de l’ADN au sein du noyau de cellules eucaryotes a ainsi révélé l’existence de sous-domaines intranucléaires appelés foyers de réplication. Chez les Métazoaires, ces foyers changent de taille, de nombre et de position au cours de la phase S. La réplication se produit dans de nombreux petits foyers répartis dans tout le noyau en début de phase S, alors que la fin de la phase S se caractérise par un nombre limité de grands foyers localisés au niveau de l’enveloppe nucléaire (Figure 5A). Les foyers ont longtemps été considérés comme des « usines de réplication » regroupant plusieurs fourches, mais cette vision est aujourd'hui remise en cause par des études en microscopie à super-résolution montrant qu'un foyer peut correspondre à une unique fourche de réplication (13) (Figure 5B). Cette technologie ouvre la voie au suivi de chaque fourche dans son contexte d’origine, la cellule. Le microscope n’a pas dit son dernier mot.
(A) Foyers de réplication observés en microscopie conventionnelle au début et à la fin de la phase S dans le noyau des cellules de Mammifères. Les sites de synthèse d’ADN ont été marqués par un analogue de la thymidine.
(B) Visualisation des sites de réplication dans le noyau de cellules de Mammifères à différents niveaux de résolution. L’amélioration de la résolution en microscopie a révélé que les foyers observés en microscopie conventionnelle peuvent être décomposés en sites de réplication de plus petite taille par microscopie confocale et microscopie à super-résolution, cette dernière permettant de visualiser des signaux correspondant à une fourche de réplication unique.
Crédits images : (A) S. Takebayashi et coll., 2017, Genes (extrait de l’original ; les annotations originales en anglais ont été traduites en français). (B) V.O. Chagin et coll., 2016, Nature Communications (extrait de l’original ; les annotations originales en anglais ont été traduites en français ; une légende a été ajoutée).
Références
1. Cairns, J. (1963) The bacterial chromosome and its manner of replication as seen by autoradiography. Journal of Molecular Biology, 6, 208-213.
2. Huberman, J.A. and Riggs, A.D. (1968) On the mechanism of DNA replication in mammalian chromosomes. Journal of Molecular Biology, 32, 327-341.
3. Cairns, J. (1966) Autoradiography of HeLa cell DNA. Journal of Molecular Biology, 15, 372-373.
4. Ogawa, T., Tomizawa, J.I. and Fuke, M. (1968) Replication of bacteriophage DNA, II. Structure of replicating DNA of phage lambda. Proceedings of the National Academy of Sciences, 60, 861-865.
5. Brewer, B.J. and Fangman, W.L. (1987) The localization of replication origins on ARS plasmids in S. cerevisiae Cell, 51, 463-471.
6. Huberman, J.A., Spotila, L.D., Nawotka, K.A., El-Assouli, S.M. and Davis, L.R. (1987) The in vivo replication origin of the yeast 2μm plasmid. Cell, 51, 473-481.
7. Parra, I. and Windle, B. (1993) High resolution visual mapping of stretched DNA by fluorescent hybridization. Nature Genetics, 5, 17-21.
8. Michalet, X., Ekong, R., Fougerousse, F., Rousseaux, S., Schurra, C., Hornigold, N., Slegtenhorst, M.v., Wolfe, J., Povey, S., Beckmann, J.S. et al. (1997) Dynamic molecular combing : stretching the whole human genome for high-resolution studies. Science, 277, 1518-1523.
9. Lebofsky, R., Heilig, R., Sonnleitner, M., Weissenbach, J. and Bensimon, A. (2006) DNA replication origin interference increases the spacing between initiation events in human cells. Molecular Biology of the Cell, 17, 5337-5345.
10. De Carli, F., Menezes, N., Berrabah, W., Barbe, V., Genovesio, A. and Hyrien, O. (2018) High-throughput optical mapping of replicating DNA. Small Methods, 2, 1800146.
11. Muller, C.A., Boemo, M.A., Spingardi, P., Kessler, B.M., Kriaucionis, S., Simpson, J.T. and Nieduszynski, C.A. (2019) Capturing the dynamics of genome replication on individual ultra-long nanopore sequence reads. Nature methods, 16, 429-436.
12. Hennion, M., Arbona, J.M., Lacroix, L., Cruaud, C., Theulot, B., Tallec, B.L., Proux, F., Wu, X., Novikova, E., Engelen, S. et al. (2020) FORK-seq : replication landscape of the Saccharomyces cerevisiae genome by nanopore sequencing. Genome biology, 21, 125.
13. Chagin, V.O., Casas-Delucchi, C.S., Reinhart, M., Schermelleh, L., Markaki, Y., Maiser, A., Bolius, J.J., Bensimon, A., Fillies, M., Domaing, P. et al. (2016) 4D Visualization of replication foci in mammalian cells corresponding to individual replicons. Nature Communications, 7, 11231.