Il existe dans la cellule un mécanisme de dégradation des protéines, utilisant de l’ATP, qui a valu à ses découvreurs le prix Nobel de chimie, en 2004. Cette voie est dite « ubiquitine-dépendante », elle permet le catabolisme de protéines ayant subi un marquage spécifique par une petite protéine (76 acides aminés), l’ubiquitine. Cette voie de dégradation est au cœur des processus biologiques, elle ne se cantonne pas à éliminer les protéines anormales ou endommagées, mais intervient dans la régulation fine des taux de protéines clés de la cellule.
Introduction
Avant-propos
La voie de dégradation ubiquitine dépendante fait partie de ces systèmes essentiels de la cellule dont l’existence était totalement ignorée il y a encore quelques dizaines d’années, et dont la découverte a bouleversé une partie de nos connaissances sur le fonctionnement cellulaire (comme a pu le faire la découverte des mécanismes de régulation du cycle cellulaire). L’importance physiologique de ce système a d’ailleurs valu à ses découvreurs le prix Nobel de chimie en 2004.
De nos jours, c’est l’un des sujets de recherche les plus actifs en biologie cellulaire. De ce fait, la situation s’est considérablement complexifiée depuis la première mise en évidence de l’existence d’une dégradation des protéines cellulaires selon un mécanisme nécessitant l’utilisation d’ATP. Afin de ne pas avoir une vision réductrice de cette voie, ce document fait une présentation assez large des connaissances actuelles sur le sujet, y compris des points sur lesquels demeurent des incertitudes ou des zones d’ombre.
Présentation générale
Le système Ubiquitine-Protéasome (UbPr) est au cœur de la plupart des processus biologiques, comme le cycle cellulaire, l’apoptose, la différenciation musculaire ou encore la réponse immune, notamment du fait de son rôle central dans la dégradation contrôlée des protéines régulatrices de ces processus.
Ainsi, le rôle de la protéolyse intracellulaire ne se cantonne pas à éliminer les protéines anormales ou endommagées, mais intervient dans la régulation fine des taux de protéines clés pour la cellule. Au vu de la multitude de substrats et des processus impliqués, il n’est pas surprenant que des dysfonctionnements de ce système soient impliqués dans de nombreuses pathologies, comme les cancers, les maladies neurodégénératives et les maladies auto-immunes.
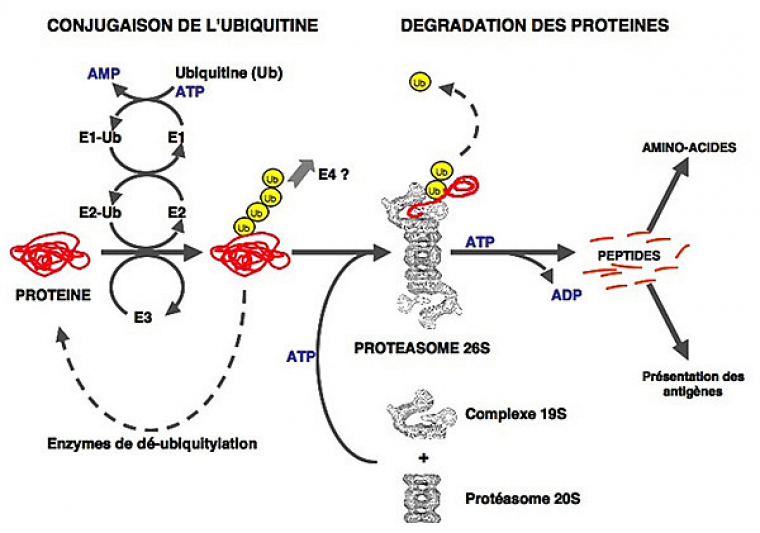
La dégradation d’une protéine via le système UbPr implique généralement deux grandes étapes successives, qui nécessitent toutes deux l’hydrolyse de l’ATP (voir Fig.1).
Dans un premier temps, l’ubiquitylation consiste en la conjugaison covalente d’une ou plusieurs molécules d’ubiquitine (Ub), sur un ou plusieurs résidus lysine de la protéine, via une cascade enzymatique impliquant 3 types de facteurs : l’enzyme d’activation de l’ubiquitine (E1), l’enzyme de conjugaison de l’ubiquitine (E2) et l’ubiquitine-ligase (E3).
Dans un deuxième temps, le substrat ubiquitylé est ensuite adressé au protéasome 26S où il est dégradé. Le protéasome 26S est un complexe protéolytique d’environ 2 500 kDa formé par l’association du protéasome 20S (le cœur protéolytique de l’enzyme) au complexe régulateur 19S (voir Fig.1).
Au cours de cette deuxième étape, le substrat est déubiquitylé, ce qui permet le recyclage de l’ubiquitine, et il est dégradé en peptides qui seront par la suite digérés par des peptidases cytosoliques pour donner des acides-aminés. Ces derniers seront réutilisés soit pour la synthèse de nouvelles protéines, soit pour la production d’énergie pour l’organisme.
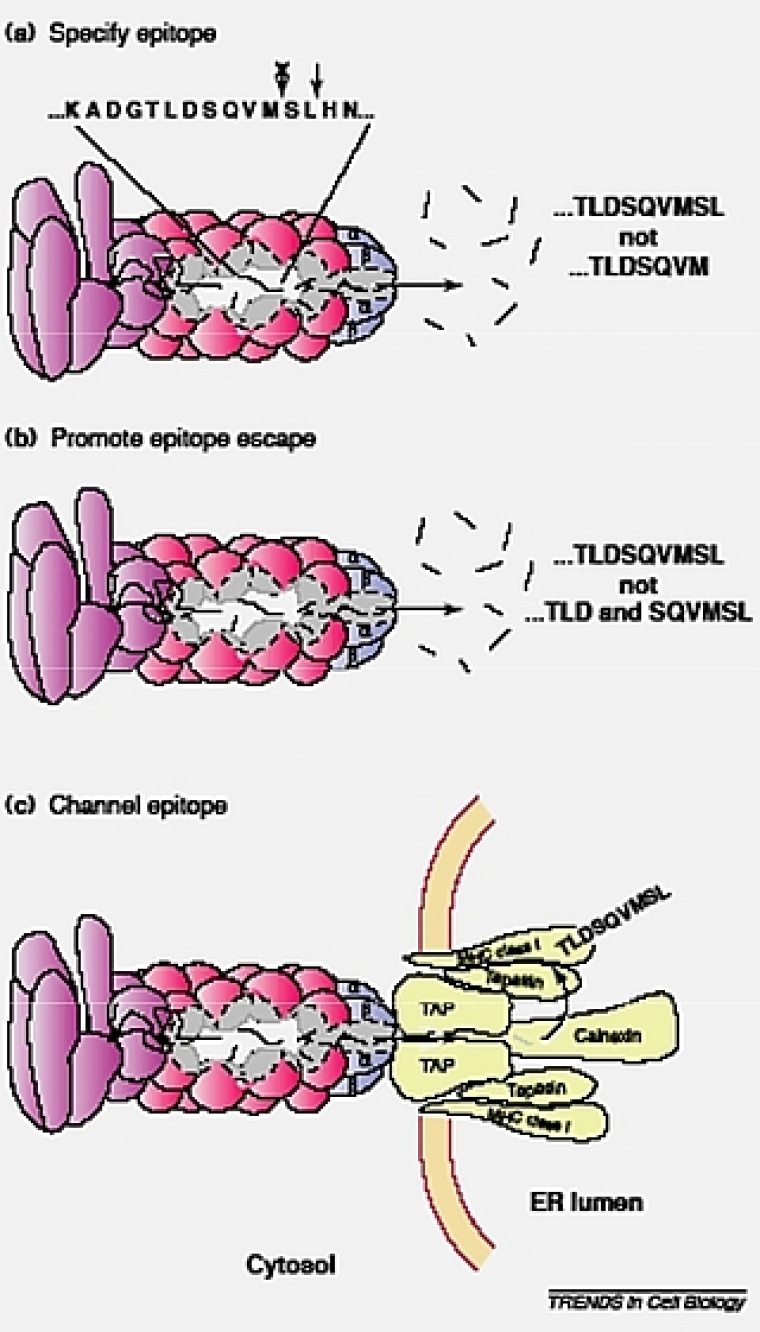
Certains peptides générés par le protéasome sont pris en charge par les molécules du complexe majeur d’histocompatibilité (CMH) de classe I, dans le cadre de la présentation des antigènes. Enfin, certaines protéines ne sont que partiellement dégradées par le protéasome qui est donc impliqué dans la maturation protéolytique de précurseurs protéiques (comme le précurseur p105 de la sous-unité p50 du facteur de transcription NFkB ou Nuclear Factor-kappa B).
Ce schéma en deux grandes étapes du système ubiquitine-protéasome n’est qu’une vue simplifiée de ce système qui est en fait bien plus complexe. De fait, si certains substrats ne nécessitent pas une ubiquitylation préalable avant d’être dégradés, l’ubiquitylation ne mène pas toujours à la dégradation. Elle est en effet impliquée dans des fonctions indépendantes de la protéolyse comme l’endocytose, l’import nucléaire, ou l’activation de kinases par exemple.
En outre, le protéasome 26S et, plus particulièrement, les sous-unités ATPases du complexe régulateur 19S, sont associées à des fonctions non-protéolytiques.
Par ailleurs, le protéasome 20S peut également être associé à d’autres régulateurs (activateurs ou inhibiteurs) que le complexe 19S, ce qui lui confère des fonctions particulières.
Ces variations autour du modèle général présenté dans la Fig.1 illustrent la complexité du fonctionnement et de la spécificité du système UbPr et expliquent qu’il existe encore un grand nombre de « boîtes noires » dans notre connaissance de ce système.
Historique
Jusqu’au début des années 80, l’intérêt scientifique dans le domaine du contrôle de l’expression des gènes a porté essentiellement sur l’étude des acides nucléiques et la traduction des ARN messagers. La dégradation des protéines était peu étudiée, car elle était considérée comme étant non spécifique et non régulée. Bien qu’il fût connu que les protéines devaient être renouvelées, l’ampleur et la spécificité de ce processus, par lequel différentes protéines ont des demi-vies qui vont de quelques minutes à quelques jours, n’étaient pas appréciées. La découverte du lysosome par Christian De Duve en 1953 n’a pas significativement changé cette idée, en effet il était clair que ces protéases n’étaient pas spécifiques des substrats. Aussi, la découverte d’un mécanisme de protéolyse intracellulaire nécessitant de l’énergie a été accueillie avec scepticisme, car les protéases connues jusqu’alors fonctionnaient sans ATP (Simpson, 1953).
Pourtant, en 1977, Etlinger et Goldberg mirent en évidence un système protéolytique non lysosomal dépendant de l’ATP, en travaillant sur des lysats de réticulocytes qui ne contiennent pas de lysosome.
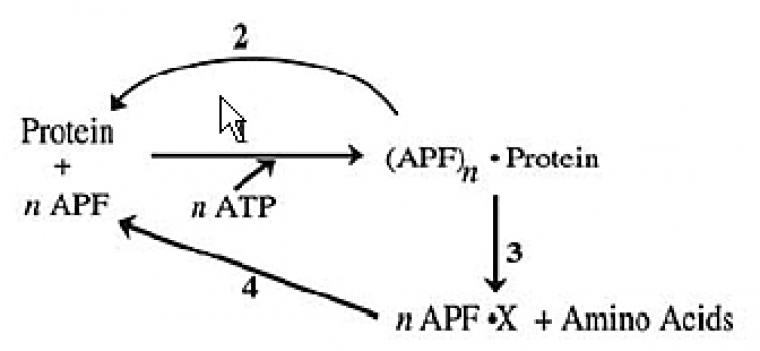
En 1978, Hershko et Ciechanover, en fractionnant ces extraits sur des colonnes de DEAE pour éliminer l’hémoglobine, observèrent que la fraction 1, constituée des protéines non retenues par la colonne, contenait un facteur nécessaire à l’activité protéolytique dépendante de l’ATP. Ils ont purifié ce facteur et l’ont identifié comme étant une petite protéine très stable d’environ 8,5 kDa, qu’ils ont appelée APF-1 pour ATP-dépendent Proteolysis Factor 1. Rapidement, ils se sont aperçus que cette petite protéine, en présence d’ATP, se liait covalemment à des protéines de haut poids moléculaire et que cette réaction était réversible. Ils ont alors imaginé un modèle où la liaison de APF-1 sur des substrats pouvait servir de signal de reconnaissance pour la protéase retenue sur la DEAE (Fig. 2). Ce modèle permettait d’expliquer à la fois la dépendance énergétique et la sélectivité du système. Il est frappant de constater le remarquable parallèle entre ce modèle initial (Fig. 2) et le modèle actuel (Fig. 1).
En 1980, APF-1 a été identifié comme étant l’ubiquitine et les années 1980-1990 ont vu la caractérisation de la cascade enzymatique d’ubiquitylation, avec l’identification des enzymes E1, E2 et E3, ainsi que la compréhension du mécanisme d’activation et de transfert de l’ubiquitine, puis la purification et la caractérisation, par plusieurs laboratoires, du protéasome 26S (responsable de la dégradation des substrats ubiquitylés) et enfin du protéasome 20S (le corps catalytique du 26S). C’est bien la découverte de l’ubiquitylation comme signal d’adressage des protéines pour la dégradation, qui a révolutionné ce domaine.
Par la suite, le début des années 90 a vu naître un intérêt pour la biologie du système UbPr, avec des études de génétique chez la levure en particulier, qui ont montré que l’ubiquitylation et le protéasome étaient essentiels. Dans le même temps, a eu lieu l’identification des premiers substrats du système UbPr chez les mammifères, tels que les cyclines, la protéine p53 et IkBa (IkappaB-alpha), qui est l’inhibiteur de la voie NFkB. Ces exemples sont d’ailleurs souvent cités comme substrats de référence. Depuis, la recherche dans le domaine a connu un essor exponentiel et, aujourd’hui, il est clair que la dégradation des protéines cellulaires est un mécanisme très complexe, contrôlé dans le temps et dans l’espace, d’une grande importance biologique.
Malgré ces très nombreuses avancées, qui ont justifié l’attribution en 2004 du prix Nobel de chimie aux découvreurs de l’ubiquitylation des protéines (Aaron Ciechanover, Avram Hershko et Irwin Rose), de nombreuses « boîtes noires » existent encore dans notre compréhension du fonctionnement du système UbPr. En particulier, des questions centrales comme « comment s’effectue l’allongement de la chaîne de poly-ubiquitine ? », ou « comment fonctionne exactement le protéasome 26S ? », restent encore largement sans réponse précise.
L’ubiquitylation des protéines
L’ubiquitine et la cascade enzymatique d’ubiquitylation
L’ubiquitine est une petite protéine de 76 acides aminés, très stable, présente dans toutes les cellules eucaryotes. Elle est très conservée au cours de l’évolution parmi les différentes espèces eucaryotes : l’ubiquitine humaine et celle de levure partage 96 % d’identité pour leur séquence protéique.
Elle est toujours exprimée sous forme de précurseurs protéiques, soit en fusion N-terminale avec une autre protéine (souvent des protéines ribosomales), soit en une succession de plusieurs molécules d’ubiquitine fusionnées les unes aux autres. Pour rendre disponible l’ubiquitine, ses précurseurs doivent donc être clivés, ce qui est assuré par des enzymes de déubiquitylation ou DUB (pour DeUBiquitinating enzymes).
Dans ce mode d’expression, l’existence de précurseurs de l’ubiquitine et leur clivage subséquent permettent probablement d’établir un « contrôle qualité » afin d’éviter la production d’ubiquitine monomérique mal traduite ou mal conformée. Par ailleurs, les fusions ubiquitine-protéines ribosomales semblent aussi jouer un rôle important dans la biogénèse des ribosomes.
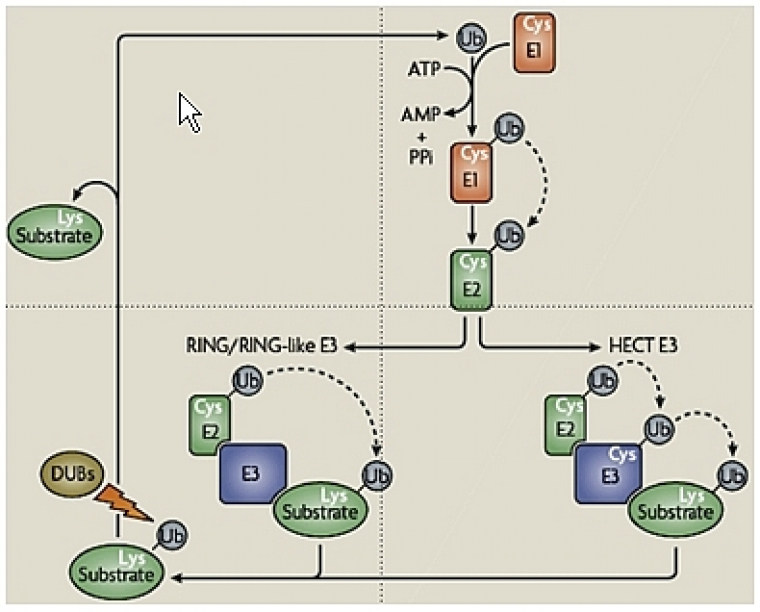
La conjugaison de l’ubiquitine aux substrats se fait par une cascade enzymatique impliquant au moins trois types de facteurs :
-
le premier est l’enzyme d’activation de l’ubiquitine, ou E1, qui effectue l’activation ATP-dépendante de l’ubiquitine en formant une liaison thiol-ester à haute énergie entre la glycine C-terminale de l’ubiquitine (via le COOH porté par son carbone α) et le groupement thiol (SH) d’une cystéine de l’E1 ;
-
le second est une enzyme de conjugaison de l’ubiquitine, ou E2, sur lequel l’ubiquitine activée est transférée au niveau du groupement thiol de sa cystéine active ;
-
enfin le troisième, l’E3, est un facteur habituellement, mais souvent incorrectement, appelé ubiquitine-ligase. Il favorise le transfert de l’ubiquitine de l’E2 vers le substrat par formation d’une liaison amide entre le groupement COOH de la glycine C-terminale de l’ubiquitine et le groupement ε-amine (amine portée sur la chaîne latérale de l’acide aminé et non par le carbone α, voir l’article « Les acides aminés ») d’une lysine du substrat, formant ainsi une liaison isopeptidique (Fig. 3).
Cependant, il a également été décrit que la conjugaison de l’ubiquitine sur le substrat pouvait se faire sur le groupement thiol d’un résidu cystéine ou sur le groupe hydroxyl (OH) d’une sérine ou d’une thréonine, et que, dans certains cas, l’ubiquitine peut être conjuguée non pas à une lysine interne de la protéine, mais au groupement α-amine du résidu N-terminal de la protéine, via une liaison peptidique.
Dans une réaction d’ubiquitylation, l’E3 est un facteur clé puisqu’il est impliqué dans la reconnaissance spécifique du substrat et est donc responsable de l’essentiel de la spécificité de la réaction. Ceci explique qu’il existe une très grande quantité d’E3s différentes, probablement des centaines chez les mammifères, alors que l’on ne compte qu’une trentaine d’E2s et que deux E1s.
Dans certains cas, la poly-ubiquitylation du substrat nécessite l’intervention d’un quatrième facteur, appelé E4, dont le mécanisme et le rôle exact ne sont pas encore bien compris (pour revue, voir Hoppe, 2005). Dans un cas décrit, ce facteur permet d’allonger et de modifier le type de chaîne (de K29 à K48) présent sur le substrat, permettant ainsi sa dégradation efficace par le protéasome.
Par ailleurs, l’ubiquitylation est un processus dynamique et réversible, du fait de l’existence de nombreuses enzymes de déubiquitylation ou DUB (Fig. 3) qui, en plus de permettre la maturation de l’ubiquitine à partir de ses précurseurs, clivent l’ubiquitine de ses substrats (protéines ou peptides associés au protéasome) et désassemblent les chaînes de poly-ubiquitine.
Les différentes familles d’ubiquitine-ligase
Les E3s forment un groupe vaste et varié de protéines qui sont généralement classées en 2 grandes familles : la famille des E3s à domaine HECT (pour Homologous to E6-associated protein C-Terminus), comportant une vingtaine de membres connus chez l’Homme, et celle des E3s à domaine RING (pour Really Interesting New Gene), comportant des centaines de membres (pour revue, voir Ardley & Robinson, 2005). Signalons qu’il existe également quelques E3s décrites dans la littérature qui ne rentrent pas dans ces deux familles.
Dans le cas des E3s à domaine HECT, le terme d’Ubiquitine-ligase est approprié, car elles jouent un rôle direct dans la conjugaison de l’ubiquitine au substrat. De fait, l’ubiquitine activée est transférée de l’E2 sur un résidu cystéine de l’E3, avant d’être conjuguée au substrat (Fig. 3).
Par contre, l’appellation d’Ubiquitine-ligase semble impropre pour les E3s à domaine RING qui, en l’état actuel des connaissances, sont des molécules d’échafaudage, mais pas de véritables enzymes. En effet, ces E3s servent de molécules adaptatrices entre l’E2 et le substrat pour faciliter l’ubiquitylation du substrat, en rapprochant celui-ci de l’E2. D’ailleurs, le motif « RING finger », qui les caractérise, forme des boucles stabilisées par coordination d’ions Zinc et permet le recrutement des E2s.
Les différents types d’ubiquitylation
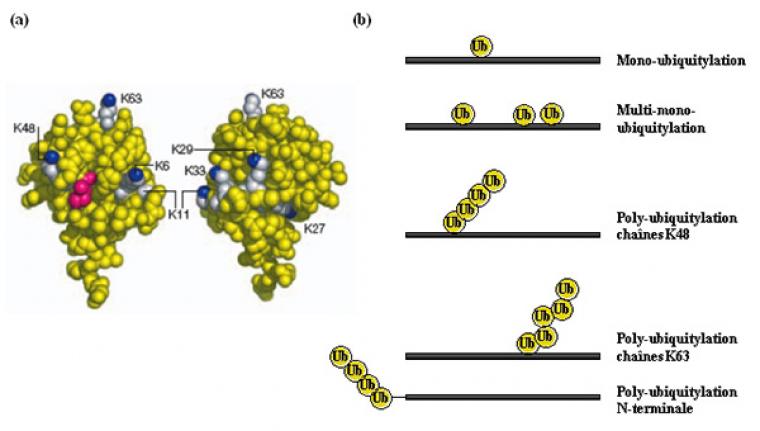
Les protéines peuvent être modifiées soit par une seule ubiquitine (mono-ubiquitylation) soit par des chaînes de poly-ubiquitine (poly-ubiquitylation), sur un ou plusieurs résidus du substrat, au niveau d’une lysine le plus souvent ; on parle alors de multi-mono-ubiquitylation et de multi-poly-ubiquitylation, respectivement (Fig. 4).
Dans le cas de la poly-ubiquitylation, la première molécule d’ubiquitine est conjuguée à une lysine du substrat, puis l’élongation de la chaîne de poly-ubiquitine se fait par la formation d’une liaison isopeptidique entre la glycine C-terminale d’une nouvelle ubiquitine et une lysine de la molécule d’ubiquitine précédemment conjuguée. Comme toutes les lysines de l’ubiquitine sont exposées à sa surface, accessibles et disponibles, il peut se former différentes chaînes, de type et de topologie variés.
Il faut noter, à ce niveau, que la poly-ubiquitylation peut être impliquée dans d’autres voies que la protéolyse cellulaire, comme l’endocytose ou la réponse inflammatoire (pour revue, voir Mukhopadhyay & Riezman, 2007).
En plus de la poly-ubiquitylation, la mono-ubiquitylation joue également un rôle important dans des fonctions très variées, comme l’internalisation des récepteurs membranaires et le trafic des protéines ou encore le remodelage de la chromatine via la modification des histones.
(a) Deux faces opposées de l’ubiquitine sont présentées ; les 7 lysines sont représentées en blanc, avec l’atome d’azote du groupement ε-amine en bleu ; l’isoleucine 44 est représentée en rouge : ce résidu est impliqué dans les surfaces d’interaction entre l’ubiquitine et les UBD. D’après (Hicke et al, 2005).
(b) Représentation schématique de différents types de modification désignés sous le nom générique "ubiquitylation" (notons que toutes les lysines de l’ubiquitine peuvent être utilisées dans la formation de chaînes de poly-ubiquitine).
Comme évoqué précédemment, l’ubiquitine contient 7 lysines (K6, K11, K27, K29, K33, K48, K63 ; Fig. 4) qui peuvent toutes être utilisées dans la formation de chaînes de poly-ubiquitine. Cela a été démontré par des analyses de spectrométrie de masse chez la levure, qui ont permis une quantification de l’abondance relative des différentes chaînes dans cet organisme (K48>K63>K11>K33=K29>K27>K6). Même si cela reste à démontrer, il est fort probable qu’il existe cette même diversité chez les eucaryotes supérieurs.
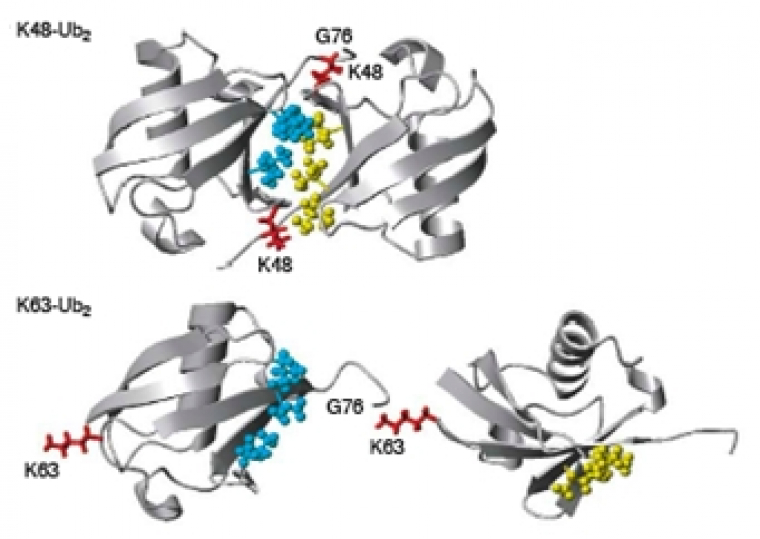
Compte tenu de la distribution des différentes lysines à la surface de l’ubiquitine, les chaînes ont une topologie très différente selon la lysine de l’ubiquitine utilisée pour la conjugaison. Effectivement, une étude de la structure de dimères d’ubiquitine conjugués, via une liaison isopeptidique, sur la lysine 48 ou la lysine 63, montre que le dimère Ub2-K48 a une structure très compacte, alors que le dimère Ub2-K63 a une structure « ouverte », comme représenté en Fig. 5.
Les régions hydrophobes sont représentées en bleu sur l’ubiquitine distale et en jaune sur l’ubiquitine proximale. La lysine impliquée dans la liaison est représentée en rouge.
D'après (Pickart et Fushman, 2004)
Les types de chaîne d’ubiquitines conjugués aux substrats ont été étudiés pendant longtemps en utilisant des mutants de l’ubiquitine ne contenant qu’une seule lysine (les autres étant mutées en arginine), ou au contraire muté sur une seule lysine. Bien que les informations obtenues par ces approches doivent être exploitées avec réserve, car en absence de la lysine « favorite » une autre lysine peut dans certains cas être utilisée, ces travaux ont plaidé pour l’existence de chaînes homogènes dans la plupart des cas, c’est-à-dire n’utilisant qu’une seule lysine de l’ubiquitine.
Le type de chaîne ainsi formé porte le nom de la lysine mobilisée : ainsi on parle de chaînes K48 lorsque l’ubiquitine distale est conjuguée sur la lysine 48 de l’ubiquitine proximale.
Cependant, plus récemment, l’utilisation de la spectrométrie de masse pour l’étude des chaînes d’ubiquitine a montré que des chaînes « mixtes » ou « branchées » pouvaient également exister, au moins in vitro. Bien que des chaînes de ce type avaient été observées, elles étaient jusqu’à récemment considérées comme des exceptions. Mais comme il a été maintenant observé des chaînes mixtes sur un substrat aussi « classique » que la cycline B ubiquitylée par l’APC/C (la cycline B est impliquée dans la régulation du cycle cellulaire, en particulier dans la transition G2/S, voir l’article « La régulation du cycle cellulaire »), cela laisse supposer que la situation pourrait être plus compliquée que ce que suggéraient les modèles initiaux.
Malgré ces difficultés, il ressort des différents travaux que ces différents types de chaîne permettent de cibler les substrats vers des voies de signalisation diverses.
Il y a, par exemple, consensus sur la règle générale énonçant que les chaînes K48 sont les plus efficaces pour l’adressage des substrats au protéasome, alors que les chaînes K63 auraient des rôles de régulation non couplées directement à la protéolyse et qui seraient importantes notamment pour la réparation de l’ADN, le transport des protéines, la réponse inflammatoire ou encore le contrôle de la fonction des ribosomes.
Cependant, cette règle générale rencontre de claires exceptions, comme la régulation par des chaînes K48, et selon un processus indépendant de la protéolyse, de l’activation du facteur de transcription Met4, ou des activités transcriptionnelles de p53.
Plus récemment, des résultats ont montré l’implication de chaînes K29 dans l’adressage des substrats au lysosome où ils seront dégradés. Les chaînes K6 semblent également permettre l’adressage des substrats au protéasome. Enfin, les chaînes de type K11 et K33 n’ont été, pour l’instant, impliquées dans aucune voie de signalisation, bien que les substrats conjugués à chaînes K11 in vitro puissent être dégradés par le protéasome.
Malgré une certaine confusion quant aux rôles spécifiques des différents types de chaîne, il ressort que la variété de leurs topologies, associée à leur reconnaissance par des effecteurs spécifiques, est sûrement responsable de la multitude des signaux transmis par les chaînes d’ubiquitine. Il existe en effet un très grand nombre de protéines possédant des domaines de liaison aux chaînes d’ubiquitine qui interviennent dans « l’aiguillage » des protéines ubiquitylées et donc dans la définition des conséquences de l’ubiquitylation.
Les récepteurs de l’ubiquitine : les protéines à UBD
L’ubiquitylation est une modification qui permet de médier des effets cellulaires aussi variés que la protéolyse, l’endocytose, la réparation de l’ADN, etc. Toutefois, les protéines peuvent être modifiées par l’ubiquitine de différentes façons : la mono-ubiquitylation et la poly-ubiquitylation, et dans ce dernier cas, différents types de chaîne peuvent être conjugués au substrat. Ces protéines ubiquitylées sont reconnues et prises en charge par des « récepteurs de l’ubiquitine » qui vont discriminer quel type d’ubiquitylation est associé au substrat et permettre leur orientation vers différentes voies cellulaires.
Ces récepteurs contiennent généralement plusieurs motifs d’interaction avec l’ubiquitine ou UBD (pour Ubiquitin Binding Domain) qui peuvent interagir de manière autonome avec l’ubiquitine. Ces récepteurs contiennent parfois des motifs d’interaction avec les substrats qui peuvent agir de concert avec les motifs UBD.
Plusieurs types d’UBD ont été caractérisés : les UIM (pourUbiquitin Interacting Motifs), les domaines UBA (pourUbiquitin Associated), les motifs CUE (pour Coupling of Ubiquitin conjugation to ER degradation), UEV, NZF…
Ces motifs sont structurellement différents, par exemple le motif UIM est constitué d’une seule hélice, alors que les domaines UBA sont formés par trois hélices groupées, ce qui peut expliquer en partie qu’ils reconnaissent des types de chaîne différents. Néanmoins, malgré la grande diversité et le nombre élevé de ces UBD, beaucoup de ces domaines vont interagir avec la même région hydrophobe, en surface de la molécule d’ubiquitine, qui comprend les résidus leucine 8, valine 70 et isoleucine 44, représentée en rouge sur la Fig. 4a.
Cependant, il faut également noter que ces domaines de liaison à l’ubiquitine reconnaissent des surfaces variées de l’ubiquitine, et pas uniquement la poche hydrophobe entourant l’isoleucine 44 (pour revue, voir Hurley et al, 2006).
Les protéines à UBD regroupent des protéines diverses, telles que des E3s et des DUB, ou encore des sous-unités du protéasome. Certaines protéines à UBD peuvent également contenir un domaine UBL (pour UBiquitin Like), qui peut lui-même interagir avec des domaines UBD, au sein de la même protéine ou entre deux protéines distinctes. Cette structure confère à certaines protéines la propriété d’adresser des substrats ubiquitylés au protéasome, en interagissant avec les substrats ubiquitylés, via leurs domaines UBD, et avec le protéasome, via leurs domaines UBL.
Les protéines « ubiquitin-like »
En plus des domaines UBL que comportent certaines protéines et qui leur confèrent des propriétés similaires à l’ubiquitine, comme la liaison à des domaines UBD, il existe de nombreuses petites protéines homologues à l’ubiquitine, appelées les protéines « ubiquitin-like ».
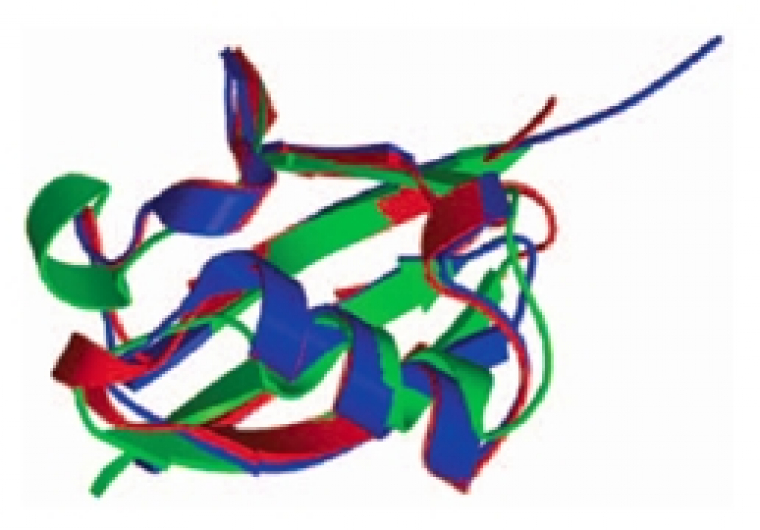
Ces protéines sont caractérisées par une structure très similaire à celle de l’ubiquitine (Fig. 6) bien que certaines ont des séquences primaires très différentes, et sont, comme l’ubiquitine, traduites sous forme de précurseurs, en fusion avec d’autres protéines, puis clivées post-traductionnellement. De plus, ces protéines ont la caractéristique, comme l’ubiquitine, de pouvoir être conjuguées sur des résidus lysines de protéines substrats.
Comme l’ubiquitylation, la conjugaison de ces molécules « ubiquitin-like » dépend d’une cascade enzymatique impliquant une enzyme E1, qui active la protéine « ubiquitin-like » de manière ATP-dépendante, et une E2, qui transfère cette protéine activée sur le substrat. Dans la plupart des cas, une enzyme E3 peut intervenir pour faciliter la conjugaison de ces « ubiquitin-like ».
Enfin, comme pour l’ubiquitylation, la conjugaison de ces « ubiquitin-like » sur des substrats est réversible, et il existe des hydrolases spécifiques de chaque « ubiquitin-like ».
Superposition des structures de l’ubiquitine (bleu), Nedd8 (rouge) et SUMO-1 (vert).
D'après (Welchman et al, 2005).
Il existe donc une grande variété d’étiquettes qui peuvent être conjuguées aux substrats avec la même spécificité que l’ubiquitine, et ces différentes modifications peuvent s’influencer entre elles, ce qui accroît la diversité des signaux menant des substrats spécifiques vers les différentes voies de signalisation. De plus la voie de l’ubiquitylation est conservée au niveau mécanistique pour les « ubiquitin-like ».
Parmi ces « ubiquitin-like », les plus répandues sont les protéines NEDD8 (pour Neural-precursor-cell Expressed Developmentally Down-regulated) et SUMO (pour Small Ubiquitin-like MOdifier). La neddylation, qui consiste en la conjugaison d’une protéine NEDD8 sur un substrat, se fait grâce à une E2 spécifique, l’enzyme Ubc12. Parmi les substrats qui peuvent être neddylés, citons p53 ou Mdm2, ce qui, dans le cas de p53, inhibe son activité transcriptionnelle.
De façon similaire, la sumoylation consiste en la conjugaison d’une protéine de la famille des protéines SUMO, qui comporte 3 membres, SUMO-1, SUMO-2 et SUMO-3 (les deux derniers étant très homologues), sur une lysine d’un substrat. L’E2 impliquée dans la sumoylation est Ubc9, et plusieurs E3 ont été décrites pour activer la sumoylation. Dans la majorité des cas, la lysine sumoylée est située au niveau d’un site consensus yKxE, où « y » représente un acide aminé hydrophobe, ou au niveau d’un site consensus étendu, le PDSM (pour Phospho-Dependent Sumo Motif) yKxExxSP, où la sérine peut être phosphorylée, ce qui a pour effet, le plus souvent, d’augmenter la sumoylation au niveau du site consensus.
De manière remarquable, les protéines SUMO-2 et SUMO-3 comportent un site consensus yKxE, et peuvent donc elles-mêmes être sumoylées ; les substrats sont alors poly-sumoylés. Toutefois, les chaînes de sumoylation semblent plus courtes que celles d’ubiquitylation.
Dans la majorité des cas, les protéines sumoylées sont reconnues par des effecteurs qui reconnaissent les molécules de SUMO via des motifs SIM (pour SUMO Interaction Motif).
La sumoylation joue souvent un rôle dans la localisation des protéines, ainsi que dans l’activation ou la répression de facteurs de transcription.
De plus, comme pour l’ubiquitine, il semble que la mono – et la poly-sumoylation puisse diriger les substrats vers des voies biologiques distinctes. La poly-sumoylation est notamment impliquée dans la dégradation des PML induite par l’arsenic, grâce à la reconnaissance spécifique des chaînes de poly-SUMO par le motif SIM de l’Ubiquitine-ligase à domaine RING RFN4, qui va l’ubiquityler et permettre sa dégradation subséquente par le protéasome.
Enfin, les désumoylases, qui font partie de la famille SENP (pour SENtrin-specific Protease), permettent la réversibilité de la réaction et donc sa régulation.
La déubiquitylation
La déubiquitylation est un processus aussi important dans la régulation du système ubiquitine que les régulations de la réaction d’ubiquitylation elle-même.
De fait, que ce soit pour ses rôles liés à la protéolyse ou ceux liés à la régulation post-traductionnelle, de la localisation ou de la fonction de certaines protéines, l’ubiquitylation doit être extrêmement bien contrôlée pour éviter des effets délétères. Aussi, de la même façon que la phosphorylation des protéines est en équilibre dynamique avec leur déphosphorylation, l’ubiquitylation des protéines est en permanence contrecarrée par leur déubiquitylation.
Par ailleurs, l’ubiquitine est une protéine très stable alors que les protéines auxquelles elle est conjuguée ont des durées de vie beaucoup plus courtes, ce qui implique qu’elle est normalement clivée de ses conjugués par les enzymes de déubiquitylation (DUB) avant ou pendant la dégradation du substrat.
Puisqu’elles sont capables de cliver l’ubiquitine, les DUB sont des protéases. Il en existe plusieurs familles qui jouent un rôle déterminant dans l’efficacité de la machinerie d’ubiquitylation, d’autant qu’elles permettent de recycler l’ubiquitine afin de maintenir un pool d’ubiquitine libre intracellulaire, nécessaire au maintien de la protéolyse. Elles sont donc essentielles dans le contrôle et le devenir des substrats ubiquitylés (pour plus de détails, voir les revues d’Amerik & Hochstrasser, 2004 et de Nijman et al, 2005).
Les DUB sont regroupées en 5 familles, basées sur les similarités de leurs séquences et sur leurs mécanismes d’action : 4 familles de protéases à cystéine (UBP, UCH, OTU/Cezanne et Ataxin-3/Josephine), et une famille de métalloprotéases dépendantes du zinc (MPN+/JAMM).
Les 2 familles principales sont la famille UBP (pour UBiquitin-specific processing Protease group, famille de cystéine-protéases qui contiennent 2 petits motifs très conservés, les boîtes Cys et His) et la famille UCH (pour Ubiquitin Carboxy-terminal Hydrolase, famille de petites protéines qui ont été identifiées au départ pour leur capacité à hydrolyser l’ubiquitine à son extrémité C-terminale).
Au niveau structural, il semble que la spécificité des différentes classes de DUB pour l’ubiquitine soit due :
-
d’une part à la configuration relativement étroite du site catalytique, qui permet de loger les deux résidus glycine situés en position C-terminale de l’ubiquitine, alors que des résidus ayant des chaînes latérales plus complexes ne pourraient vraisemblablement pas y accéder,
-
et d’autre part à une stabilisation du site catalytique entraîné par un réarrangement conformationnel suite à l’association de l’enzyme avec l’ubiquitine.
Au niveau mécanistique, les DUB peuvent être amenées à cliver des liaisons isopeptidiques, des liaisons peptidiques ou des thiol-esters. In vitro, de nombreuses DUB sont capables d’effectuer le clivage de ces trois types de liaisons.
Par ailleurs, on a vu que l’ubiquitine est toujours produite sous forme de précurseurs et qu’un rôle essentiel des DUB est donc de cliver ces précurseurs afin que l’ubiquitine soit disponible pour être conjuguée à des substrats. Il semble que ce rôle puisse être effectué par plusieurs DUB, puisque aucune DUB (à part Rpn11) n’est essentielle chez la levure.
La sous-unité Rpn11 du protéasome, le représentant de la famille des métalloprotéases MPN+/JAMM, présente une activité de déubiquitylation, mais uniquement quand elle est associée au complexe régulateur 19S du protéasome 26S. Par ailleurs, plusieurs activités de déubiquitylation ont été identifiées comme associées au protéasome.
De manière intéressante, beaucoup de DUB sont localisées au niveau de structures cellulaires particulières ou au sein de complexes moléculaires, comme la sous-unité Rpn11. Ces localisations semblent déterminer leur spécificité et influer sur leurs activités catalytiques, de même qu’elles permettent de réguler la fonction des DUB en permettant un rapprochement physique entre l’enzyme et son substrat. D’ailleurs, certaines DUB ont été caractérisées pour avoir une spécificité de substrat. C’est le cas notamment de l’enzyme HAUSP/USP7, qui appartient à la famille des UBP. Cette isopeptidase interagit avec la protéine p53 et est capable de la déubiquityler in vitro et in vivo, ce qui influe sur le niveau d’expression de la protéine.
Enfin, il existe une DUB, appelée isopeptidase T (UBP14 chez la levure), qui clive préférentiellement les ubiquitines de chaînes libres, à partir de l’ubiquitine proximale, ce qui suggère qu’elle travaille probablement en aval du protéasome, digérant les chaînes d’ubiquitine libres libérées par le protéasome, ou par d’autres isopeptidases, en molécules d’ubiquitine monomériques. Cette activité a le double rôle de recycler l’ubiquitine et de débarrasser la cellule des chaînes libres d’ubiquitine qui peuvent, lorsqu’elles s’accumulent, inhiber le protéasome.
Conclusions
Les notions introduites dans cette partie montrent que l’ubiquitylation est une réaction contrôlée à de nombreux niveaux :
-
au niveau du substrat qui peut subir un certain nombre de modifications pour être reconnu par l’E3,
-
au niveau de l’E3 dont l’activité peut être régulée par différents mécanismes,
-
et au niveau des étapes post-ubiquitylation.
Au vu de la diversité des signaux générés par les différents types d’ubiquitylation, ajoutés à ceux dus aux « ubiquitin-like », on peut envisager de découvrir encore de nombreux rôles de ces modifications dans le ciblage de substrats au niveau des différentes voies de signalisation.
Le protéasome
Le protéasome 20S
Le protéasome 20S est le cœur protéolytique des différentes formes de protéasome. Cependant, bien que le protéasome 20S seul peut hydrolyser de petits peptides et certaines protéines dépliées, il ne peut pas dégrader de véritables substrats poly-ubiquitylés, pour lesquels il a besoin d’être associé à son régulateur 19S.
Composition et structure
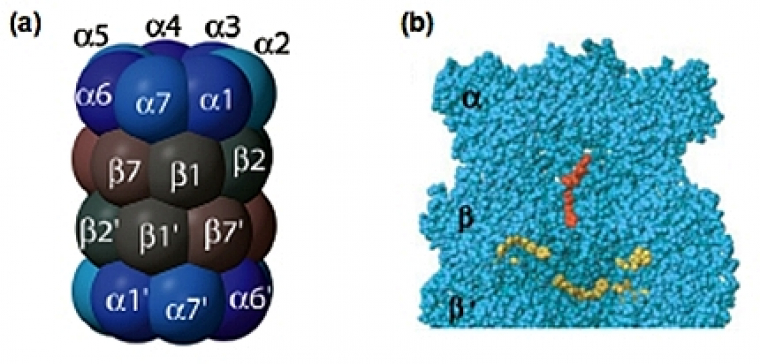
Le protéasome 20S est un complexe multi-protéique d’environ 700 kDa. Sa structure a été déterminée par microscopie électronique puis par cristallographie, ce qui a révélé un cylindre creux, de 15 nm de long et de 11 nm de diamètre, composé de 28 sous-unités assemblées en 4 anneaux heptamériques distincts, identiques 2 à 2, empilés dans une symétrie C2. Les 2 anneaux externes sont formés de 7 sous-unités α et les 2 anneaux internes de 7 sous-unités β, qui portent les activités catalytiques (Fig. 7).
Chez l’archaebactérie Thermophila acidophilum, le protéasome 20S se compose de 14 copies de chacune des 2 sous-unités α et β (α7β7β7α7), alors que chez les eucaryotes, il est construit à partir de 2 copies de 7 sous-unités α et 7 sous-unités β différentes, mais présentant de fortes homologies de séquences (α1-7β1-7β1-7α1-7).
Les 2 anneaux bêta délimitent la cavité interne, tandis que les cavités externes (appelées « antichambres ») sont délimitées par un anneau alpha et un anneau bêta. Cette structure favoriserait la rétention des substrats à l’intérieur du cylindre et jouerait de ce fait un rôle déterminant dans la processivité du protéasome.
Même si la composition du protéasome 20S des eucaryotes est plus complexe que celui des archaebactéries, en raison de l’augmentation du nombre des différentes sous-unités au cours de l’évolution, la structure quaternaire de ces complexes est très similaire.
(a) Vue schématique latérale (Rechsteiner & Hill, 2005).
(b) Vue en coupe, montrant les sites actifs β1, β2 et β5 en jaune et le potentiel site actif β7 en rouge (Unno et al, 2002). Concernant ce dernier, une analyse structurale aux rayons X du protéasome bovin suggère qu’il présente une nouvelle activité nucléophile hydrolase N-terminale. Ce site catalytique pourrait avoir une activité SNAAP (pour Small Neutral Amino Acid-Preferring). Contrairement aux autres sites catalytiques, il n’est pas dans la cavité interne formée par les anneaux bêta, mais au niveau de l’antichambre formée par les anneaux alpha et bêta.
Chez les archaebactéries, toutes les sous-unités β sont identiques, ce qui fait 14 sites catalytiques actifs au total. Chez les eucaryotes, seules 3 des 7 sous-unités β (β1, β2 et β5) possèdent une activité protéolytique, ce qui signifie que chaque protéasome possède 6 sites catalytiques (Fig. 7b, représentés en jaune).
Le site actif est toujours caractérisé par un résidu thréonine dont le groupement amine sert de nucléophile dans le cycle catalytique. Cependant, les sites catalytiques présentent des spécificités distinctes, définies par la nature physico-chimique de l’environnement des sites actifs. Ainsi, les sous-unités β2 qui présentent un environnement acide où vont se loger des résidus basiques, possèdent une activité trypsin-like qui coupe après les résidus basiques. Les sous-unités β5 possèdent une activité chymotrypsin-like qui coupe après les résidus hydrophobes. Les sous-unités β1 possèdent une activité caspase-like qui coupe après les résidus acides.
Chez les mammifères, un traitement à l’interféron γ stimule l’expression de 3 sous-unités β actives supplémentaires, appelées les sous-unités β induites, βi, ou LMP. Chacune d’elles va remplacer une sous-unité β constitutive spécifique, qui lui est génétiquement homologue, et être incorporée à la position correspondante à l’intérieur de l’anneau β du nouveau protéasome assemblé. La sous-unité β1i/LMP2 remplace son homologue exprimé constitutivement β1, β5i/LMP7 remplace β5 et β2i/LMP10 (MECL1) remplace β2. Le protéasome ainsi formé, suite à un signal de l’interféron γ, est appelé « immuno-protéasome »: il génère des peptides dont la taille est mieux appropriée pour la présentation des antigènes (en moyenne 8 à 11 acides aminés au lieu de 6 à 8 en moyenne) par les molécules du CMH de classe I.
Les sous-unités α de T. acidophium, contrairement aux sous-unités β, sont capables de s’associer par elles-mêmes, grâce à leurs extrémités N-terminales, pour former les anneaux et l’assemblage de ces anneaux alpha est nécessaire pour la formation des anneaux bêta. Chez les eucaryotes cependant, plusieurs protéines chaperones sont importantes pour l’assemblage des anneaux alpha et bêta, et pour la formation du protéasome 20S. Les sous-unités β possèdent une pro-séquence N-terminale, qui est clivée pendant la formation du complexe. Chez les eucaryotes, contrairement aux archaebactéries, cette pro-séquence est requise pour l’incorporation des sous-unités et pour l’assemblage normal du protéasome 20S. Le clivage de cette pro-séquence est néanmoins nécessaire à leur activité protéolytique, car il permet d’exposer le résidu thréonine engagé dans les activités catalytiques.
Ouverture du pore
Chez les eucaryotes, les extrémités N-terminales des sous-unités α forment un réseau d’interactions entre elles et constituent ainsi une barrière physique qui obstrue l’entrée du canal et limite l’accès des protéines cytosoliques à l’intérieur de la chambre protéolytique, en imposant une conformation fermée au protéasome. La sous-unité α3 semble être particulièrement impliquée dans la fermeture du pore. En effet, son extrémité N-terminale forme une sorte de pont qui traverse le pore, et elle est en contact avec toutes les autres extrémités N-terminales. De plus, des analyses cristallographiques du protéasome 20S de S. cerevisiae ont montré que la délétion de l’extrémité N-terminale de la sous-unité α3 ouvre un canal vers la cavité catalytique interne du protéasome 20S, levant ainsi la répression de l’hydrolyse peptidique.
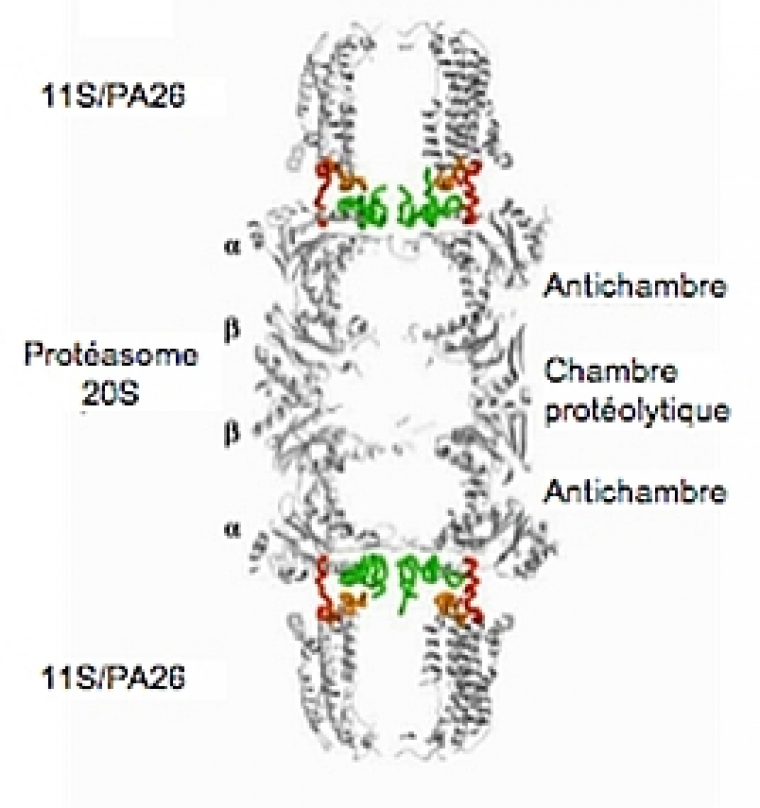
Complexe chimérique constitué du 20S de levure et du 11S de Trypanosoma brucei. Les extrémités N-terminales des sous-unités alpha sont représentées en vert et les extrémités C-terminales du 11S sont représentées en rouge. D’après (Groll & Clausen, 2003).
L’activation du protéasome 20S nécessite donc un réarrangement de ces extrémités.
Effectivement, in vitro, il est possible d’activer artificiellement le 20S par différents traitements, comme une incubation prolongée à 37 °C, un chauffage à 55 °C, une incubation avec des faibles concentrations de SDS (Sodium Dodecyl Sulfate, un détergent) ou avec des acides gras. Ces traitements induisent probablement des changements de conformation des protéines qui altèrent les anneaux alpha et facilitent l’entrée des substrats. Par ailleurs, il a également été montré que certaines protéines non repliées étaient capables d’ouvrir l’accès à la chambre protéolytique en absence d’activateurs, probablement via des interactions non-spécifiques avec des motifs hydrophobes de leurs séquences.
Les extrémités N-terminales des sous-unités α correspondent également aux sites de liaison avec les complexes régulateurs qui modulent l’activité protéolytique du protéasome, en rendant accessible l’intérieur du complexe et en levant ainsi l’inhibition due à ces extrémités N-terminales. Il existe en effet plusieurs régulateurs du protéasome 20S capables de s’associer à ce complexe et de modifier ses activités catalytiques, notamment les régulateurs PA28/11S et le complexe 19S (voir ci-dessous).
La structure cristallographique du complexe 20S-11S a permis de montrer que l’association du corps protéolytique avec son activateur engendre un réarrangement des extrémités N-terminales des sous-unités α, qui, ainsi, n’obstruent plus le pore, mais sont dressées à l’intérieur de l’activateur, ce qui ménage un accès au canal du 20S (Fig. 8).
Les différents régulateurs
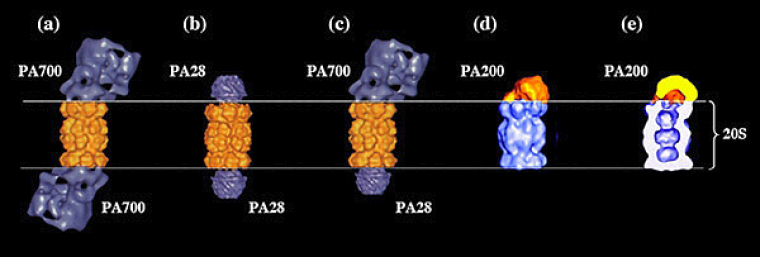
Comme évoqué précédemment, il existe des activateurs du protéasome 20S en absence desquels les extrémités du 20S sont fermées et l’activité du protéasome réprimée. Ces régulateurs positifs sont les complexes 19S (ou PA700), 11S (ou PA28), et PA200 (où PA signifie Proteasome Activator) qui, en s’associant aux extrémités du 20S, au niveau de l’anneau des sous-unités alpha du 20S, vont permettre l’ouverture des canaux et donc l’activation du 20S (Fig.9).
Reconstitutions tridimensionnelles des structures obtenues en microscopie électronique
(a, b et c, d’après Baumeister et al, 1998 ; d et e d’après Ortega et al, 2005).
Les données structurales obtenues pour ces régulateurs du protéasome montrent qu’ils sont traversés par un pore ou un canal qui va se positionner dans le prolongement du canal du 20S. Pour PA28 (ou 11S), les données cristallographiques le montrent très clairement (Fig. 8 et 9b). En ce qui concerne PA200, des données de microscopie électronique prédisent un agencement en solénoïde dont le pore pourrait se positionner dans le prolongement du canal du 20S (Fig. 9e). Pour le 19S (ou PA700), cet agencement semble concerner au moins le sous complexe appelé « base ».
Ces données de structure sont en faveur d’un rôle de ces activateurs dans la régulation du 20S. Néanmoins, seul le complexe 19S, associé au 20S, formant ainsi le protéasome 26S, est considéré comme étant impliqué dans la dégradation de substrats ubiquitylés ou non. Les autres régulateurs, PA28 et PA200, semblent jouer d’autres rôles puisqu’ils sont décrits comme étant uniquement capables d’activer la dégradation de peptides par le protéasome 20S, mais pas celle de protéines. Mais certains résultats laissent entrevoir d’autres rôles possibles.
Des données récentes concernant le complexe PA28 γ, un des 3 membres de la famille PA28, ont montré qu’il était impliqué dans la dégradation de protéines importantes pour le contrôle du cycle cellulaire (voir plus loin, III-C) ce qui remet en question le paradigme selon lequel seul le complexe 19S est le seul régulateur impliqué dans la dégradation des substrats. Par ailleurs, des formes hybrides du protéasome ont été identifiées où le 20S est associé à une extrémité à un complexe 19S et à l’extrémité opposée soit à PA28, soit à PA200. Dans ce contexte, PA28 et PA200 pourraient moduler l’activité du 26S, notamment en facilitant la sortie des peptides produits, suggérant que la stimulation concertée des activités protéolytiques et peptidolytiques pourrait optimiser la dégradation des protéines substrats. De fait, PA28 αβ stimule préférentiellement les 3 activités chymotryptique-, tryptique – et caspase-like du protéasome, PA28 γ l’activité tryptique-like et PA200 l’activité caspase-like.
PA200 est le plus récent régulateur du protéasome à avoir été découvert. Contrairement à PA28 αβ, qui est un complexe multimérique, PA200 est une protéine de 200 kDa qui agit sous forme de monomères. De part son homologie avec la protéine de levure Blm3p (maintenant connue comme Blm10p), dont les défauts sont connus pour conférer une sensibilité à la bléomycine, un agent provoquant des dommages à l’ADN, et l’interaction de cette protéine avec la protéine Sir4p, une protéine de levure qui répare les cassures d’ADN double-brin, un rôle de PA200 dans la réparation de l’ADN a été proposé. Cette hypothèse est appuyée par le fait que, à la fois les ARNm et les protéines PA200 sont très abondants dans les testicules, où des cassures d’ADN double-brin se produisent fréquemment durant la recombinaison méiotique et par le fait que PA200 est présent à l’intérieur du noyau et, comme pour les composants de la réparation de l’ADN dans les cellules de mammifères, forme des foyers après une irradiation-γ. Cependant, d’autres données ont montré que :
une délétion de Blm10p ou de PA200 ne résulte pas en une augmentation de la sensibilité à la bléomycine ;
des souris qui n’ont plus de PA200 présentent un développement lymphocytaire normal, alors que ce processus nécessite des protéines de réparation de l’ADN pour réarranger les segments de gènes des récepteurs aux antigènes ;
PA200 n’est pas essentiel pour la réparation des cassures double-brin de l’ADN, mais qu’il pourrait avoir une importante fonction durant la spermatogenèse. Néanmoins, une étude récente a montré que, suite à une irradiation-γ, PA200 est recruté au niveau de la chromatine où il forme des protéasomes hybrides PA200-20S-19S qui s’y accumulent, aboutissant à une augmentation de l’activité protéolytique du protéasome. Le rôle de PA200 au niveau de la stabilité génomique serait donc dépendant de sa capacité à stimuler l’activité caspase-like du protéasome et agirait plutôt en aval d’une réparation de l’ADN complète.
Il existe également des régulateurs négatifs du protéasome 20S dont un certain nombre d’inhibiteurs naturels. La protéine chaperonne Hsp90 diminue in vitro l’activité du protéasome 20S d’environ 2 fois. Il a par ailleurs été montré que Hsp90 inhibe la forme latente du protéasome 20S, mais pas sa forme active, ce qui peut expliquer que Hsp90 n’affecte pas le 20S associé à PA28 αβ. En outre, Hsp90 n’affecte pas non plus l’immuno-protéasome. Ces données sont cohérentes avec le potentiel rôle de Hsp90 et de PA28 dans la production protéasomale des peptides antigéniques du CMH de classe I.
La protéine PI31 (où PI signifie Proteasome Inhibitor), une protéine de 31 kDa qui contient des séquences riches en prolines, est présente chez les plantes et les eucaryotes supérieurs, mais pas chez la levure. Elle inhibe plus fortement les activités chymotrypsin-like et caspase-like du protéasome 20S que ses activités trypsin-like. De façon surprenante, elle n’inhibe que d’un facteur 2 le protéasome 26S et les complexes 20S-PA28 αβ, même à de très fortes concentrations, alors qu’elle présente une affinité pour le 20S qui est environ 50 fois supérieure à celle de PA28 αβ. La surexpression de PI31 dans des cellules de mammifères inhibe la formation de l’immuno-protéasome et la présentation des antigènes de classe I, sans réellement inhiber les fonctions générales du protéasome. Toutefois, ses fonctions biologiques sont mal comprises, d’autant qu’elle est présente chez les plantes qui n’ont ni immuno-protéasome ni réponse antigénique de classe I.
Un autre inhibiteur riche en prolines et en arginines est la protéine PR39, un poly-peptide de 39 résidus sécrété par les macrophages qui est capable de traverser les membranes cellulaires. De même que PI31, il inhibe in vitro l’activité des protéasomes 26S et des complexes 20S-PA28 αβ, en inhibant majoritairement les activités chymotrypsin-like et caspase-like sans affecter les activités trypsin-like.
Deux autres inhibiteurs d’origine virale ont été identifiés : la protéine Tat du virus VIH et la protéine HBx du virus de l’hépatite B. In vitro Tat inhibe l’activité peptidasique du protéasome 20S et empêche l’activation du 20S par PA28 αβ. In vivo des données montrent une altération de la réponse immune de classe I dans des cellules surexprimant Tat. Ceci-dit, in vivo, un rôle important de Tat serait de favoriser la dissociation du protéasome 20S et du régulateur 19S. En ce qui concerne la protéine HBx, elle est capable d’inhiber le protéasome, tout en en étant une cible. Elle pourrait également inhiber l’activation du protéasome 20S par PA28 αβ. Son action est encore mal comprise, mais un modèle qui se dégage serait en faveur d’une inhibition de l’immuno-protéasome par ces protéines virales. Il existe par ailleurs d’autres inhibiteurs du protéasome, mais dont la fonction et le mécanisme d’action n’ont pas été documentés.
Le protéasome 26S
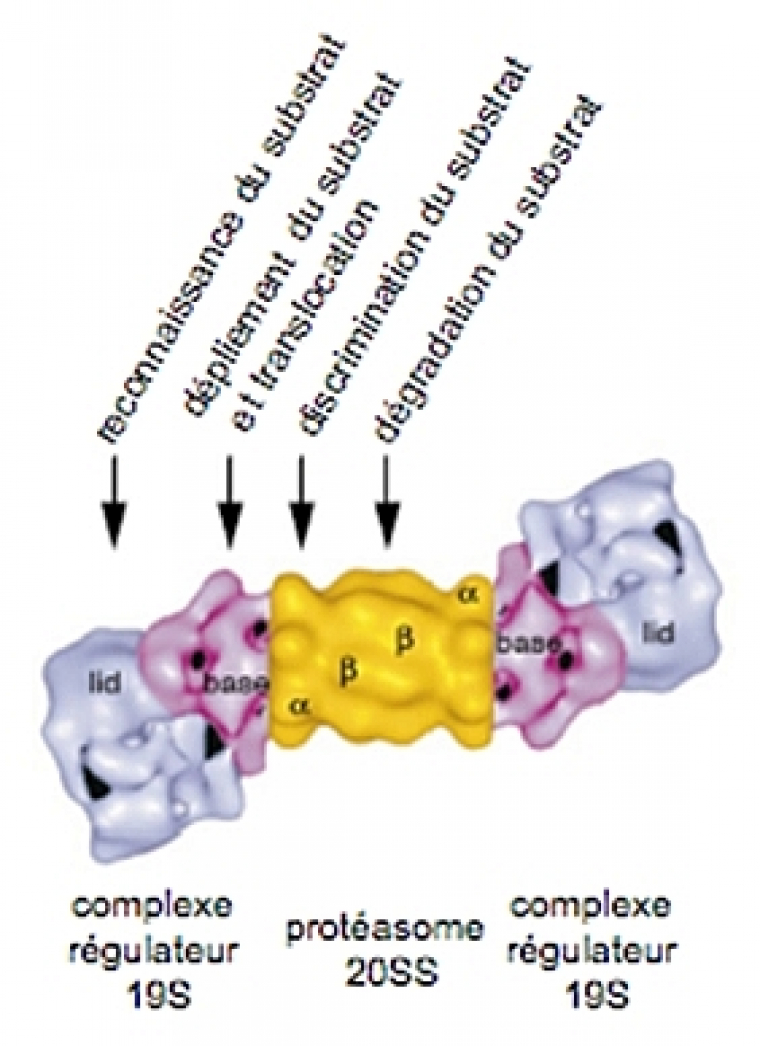
Le protéasome 26S, formé par l’association du corps catalytique 20S avec un ou deux complexes régulateurs 19S, est impliqué dans la dégradation des substrats, ubiquitylés ou non, dépendante de l’ATP. Le complexe régulateur 19S, un complexe macromoléculaire de 900 kDa, prend en charge plusieurs rôles dans la régulation de l’activité du protéasome :
-
la reconnaissance des substrats (ubiquitylés ou non) ;
-
la déubiquitylation des substrats ubiquitylés pour recycler l’ubiquitine ;
-
la dénaturation des substrats ;
-
l’ouverture du canal ;
-
la translocation des substrats dans le 20S.
Il est possible que le 19S joue également un rôle dans la sortie des peptides digérés. De plus, le 19S, tout comme les autres régulateurs du 20S, pourrait jouer un rôle dans la localisation du protéasome, mais ces deux dernières pistes ont été très peu étudiées.
Composition et structure du 19S
Le complexe 19S n’a pas pu être cristallisé, ni seul, ni en association avec le 20S. Sa structure exacte n’est donc pas connue. De plus, le nombre exact des sous-unités du 19S dans les différents organismes est inconnu, car il n’est pas exclu que certaines sous-unités se lient transitoirement et/ou puissent être perdues en fonction des procédures de purification utilisées, qu’elles soient en quantité substoechiométrique ou encore qu’elles soient spécifiques des espèces. Néanmoins, à quelques exceptions près, la composition des sous unités du 19S entre différentes espèces est remarquablement similaire. Pour désigner ces sous-unités, deux nomenclatures sont couramment utilisées, la nomenclature « S » des mammifères (où « S » signifie Subunit) et la « Rp » de S. cerevisiae (où « Rp » signifie Regulatory Particle) où l’on distingue les « Rpt » pour les ATPases et les « Rpn » pour les sous-unités non ATPases. Par ailleurs, la structure analysée par microscopie électronique, combinée à des études de mutagénèse chez la levure ont montré que ce complexe se compose de 2 sous-complexes : la « base », qui contient 6 ATPases (Rpt1 à Rpt6) et 3 sous-unités non-ATPasiques, Rpn1, Rpn2 et Rpn10, et le « couvercle » qui contient 9 sous-unités, toutes non ATPasiques (Rpn3, 5, 6, 7, 8, 9, 11, 12 et Rpn15) (Fig.10). Une autre sous-unité, Rpn13, est considérée comme une sous-unité intrinsèque du 19S, jouant un rôle important dans la reconnaissance des substrats ubiquitylés, mais son appartenance à la base ou au couvercle n’a pas encore été déterminée. Enfin, une sous-unité additionnelle, la sous-unité S5b, qui interagit in vitro avec les sous-unités Rpn1/S2, Rpt2/S4 et Rpt1/S7, est présente dans le complexe de mammifère.
Reconstruction de la structure du protéasome 26S à partir d’images de microscopie électronique montrant les différents sous-complexes et leur fonction. D’après (Zwickl & Baumeister, 1999).
La base
Le protéasome 20S associé à la base du 19S est suffisant pour la dégradation dépendante de l’ATP des substrats non ubiquitylés, mais ce complexe est incapable de dégrader des substrats ubiquitylés, ce qui suggère un rôle de la base dans le dépliement des substrats et dans leur translocation au travers du canal du 20S et un rôle du couvercle dans la reconnaissance des substrats ubiquitylés et dans la déubiquitylation de ces substrats.
Les 6 ATPases de la base contiennent une séquence AAA (ATPases Associated with a variety of cellular Activities) ou CAD (Conserved ATPase Domain) longue de 230 acides aminés, caractéristique de la famille des AAA ATPases. Les données connues sur les ATPases semblent indiquer qu’elles s’associent sous forme d’un anneau hexamérique qui assure l’interface entre le 19S et le 20S et permet l’ouverture du pore. Le fait que l’association du 20S et du 19S nécessite de l’ATP et le parallèle avec le complexe PAN (Proteasome-Activating Nucleotidase) des archaebactéries, un complexe composé de 6 ATPases identiques qui s’organisent en anneau hexamérique pour s’associer à l’extrémité du cylindre du 20S et l’activer, suggère que l’association du 20S et du 19S se fait par une interaction directe entre le 20S et les ATPases. Cette hypothèse est appuyée par les cartes d’interactions entre les différentes sous-unités qui montrent notamment des interactions entre les sous-unités Rpt et les sous-unités alpha du 20S. Néanmoins, des travaux récents indiquent un rôle très important de la sous-unité Rpn2 dans l’interaction 20S/19S.
La sous-unité Rpn10 semble jouer un rôle dans la cohésion entre la base et le couvercle. En effet, l’absence de cette sous-unité entraîne dans certaines conditions la dissociation des deux sous-complexes, le couvercle se détachant donc des protéasomes purifiés à partir de ces mutants de délétion. En outre, même si la sous-unité Rpn10 est étroitement liée à la base, via sa partie centrale, elle peut également se lier au couvercle, via son extrémité N-terminale, ou être trouvée, en quantité élevée, non associée au protéasome. D’ailleurs, alors que la plupart des sous-unités du 20S et du 19S possèdent un même élément PACE (une séquence nonamérique 5'-GGTGGCAAA-3') au niveau de leur promoteur qui indique qu’elles subissent un contrôle de leur transcription coordonné, la sous-unité Rpn10 en est dépourvue, ce qui explique qu’elle soit la seule sous-unité dont le niveau non associé au protéasome est élevé, au moins dans certaines espèces. Il a été suggéré que les sous-unités Rpn10 libres pouvaient séquestrer les substrats poly-ubiquitylés pour les empêcher d’atteindre le protéasome. Cependant, la sous-unité Rpn10 ne semble pas être suffisante pour l’association entre la base et le couvercle ; elle aurait plutôt un rôle stabilisateur de l’association base/couvercle. Aussi, il a été suggéré que Rpn1 et Rpn2 pouvaient également intervenir dans cette association. En effet, ces sous-unités interagissent avec plusieurs Rpt (voir plus loin) et également avec deux sous-unités du couvercle, Rpn11 pour Rpn1 et Rpn12 pour Rpn2.
Ces sous-unités Rpn1 et Rpn2 contiennent des séquences riches en leucine (LRR), également trouvées dans BimE, la plus large sous-unité de l’APC (Anaphase Promoting Complex)/cyclosome. Deux modèles ont été prédits sur la base de ces domaines particuliers. Le modèle de Lupas prédit une structure en demi-tore tandis que le modèle de Kajava prédit une structure en toroïde (en anneau). Cette dernière structure semble particulièrement intéressante parce qu’elle permet d’imaginer un modèle où les sous-unités Rpn1 et Rpn2 sont positionnées l’une au dessus de l’autre, avec leur deux pores qui s’alignent avec le pore potentiel de l’anneau des ATPases, et la position des charges comprises à l’intérieur de ces pores pourrait fournir une force capable de favoriser le dépliement des substrats et leur insertion dans le 20S. D’ailleurs, des données récentes suggèrent que Rpn2 contacte directement l’anneau alpha du protéasome 20S et que Rpn1 se situe au dessus de Rpn2, et sont ainsi en faveur du modèle de Kajava. En outre, des données concernant leur interaction avec les ATPases et les sous-unités de la base montrent qu’il existe des interactions directes de la sous-unité Rpn1 avec les ATPase Rpt1, Rpt2, Rpt6 et Rpn2 et que la sous-unité Rpn2 interagit avec les ATPases Rpt4, Rpt6 et avec la sous-unité Rpn13, que nous considérons ici comme une sous-unité de la base, bien que cette donnée n’a pas encore été démontrée (voir plus haut).
Enfin, il existe un sous-complexe supplémentaire appelé “modulateur”, composé de 2 ATPases (Rpt4 et Rpt5) et d’une protéine appelée p27, qui serait un activateur ou un facilitateur de la formation du 26S ; cependant, la protéine p27 n’est pas considérée comme une sous-unité du protéasome à proprement parler.
Le couvercle
Le couvercle est nécessaire pour la dégradation des substrats ubiquitylés, en assurant la reconnaissance des substrats et/ou leur déubiquitylation, par clivage des chaînes d’ubiquitine via la(es) isopeptidase(s) du couvercle (voir plus loin). En outre, il joue également un rôle au niveau de la structure puisqu’il ménage une cavité à l’intérieur du 19S, probablement pour permettre un positionnement du substrat favorable à sa dénaturation et à sa translocation dans le 20S par la base.
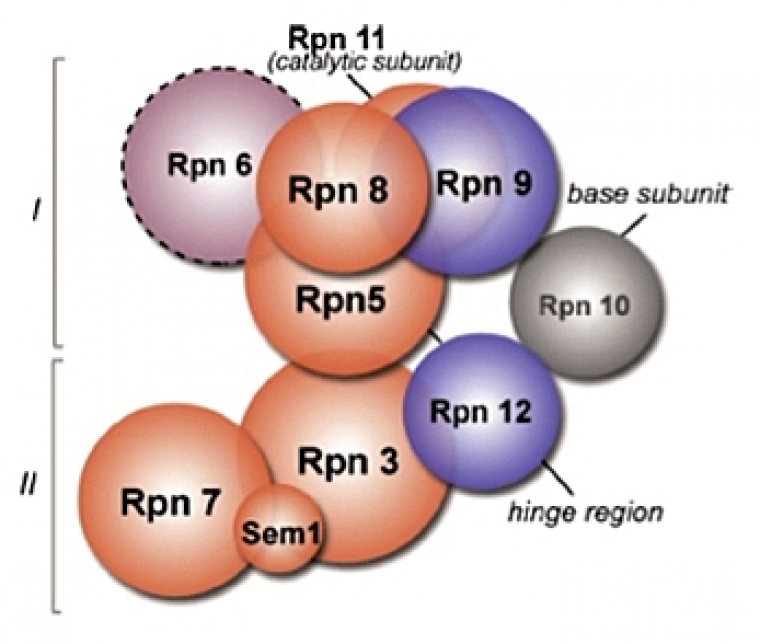
Si la structure du couvercle n’a pas encore été déterminée, la combinaison de plusieurs études (approches double-hybride, expériences de pontage chimique couplées à la spectrométrie de masse, données génétiques et biochimiques) ont permis de proposer un modèle détaillé de l’organisation du couvercle (Fig.11).
Organisation supposée du couvercle du 19S, en 2 sous-groupes : (I) composé de Rpn5, Rpn6, Rpn8, Rpn9 et Rpn11, et (II) composé de Rpn3, Rpn7, Rpn12 et sem1/Rpn15. L’interaction Rpn3-Rpn5 semble assurer le lien entre ces 2 sous-groupes. La sous-unité Rpn10 qui stabilise l’association base/couvercle semble interagir avec Rpn9 et Rpn12.
D’après (Sharon et al, 2006).
Les sous-unités du couvercle sont toutes, à l’exception de Rpn15, des sous-unités non ATPases qui contiennent soit un motif PCI (Proteasome, COP9, eIF3) (Rpn3, 5, 6, 7, 9 et 12), soit un motif MPN (Mpr1, Pad1 NH2-terminal) (Rpn8 et 11), que l’on retrouve dans d’autres complexes protéiques, tel que le complexe régulateur de la traduction eIF3 (eucaryotic Initiation Factor 3) et le complexe CSN (COP9 signalosome), qui semblent avoir des connexions à plusieurs niveaux avec le système UbPr. La composition du couvercle, du CSN et de eIF3 sont étonnamment similaires (6 sous-unités contiennent un motif PCI et 2 un motif MPN). Ces motifs, qui favorisent les interactions protéines-protéines, ont été identifiés, par des études de mutations, comme étant des motifs critiques pour l’assemblage du couvercle, bien que d’autres régions protéiques peuvent également être impliquées.
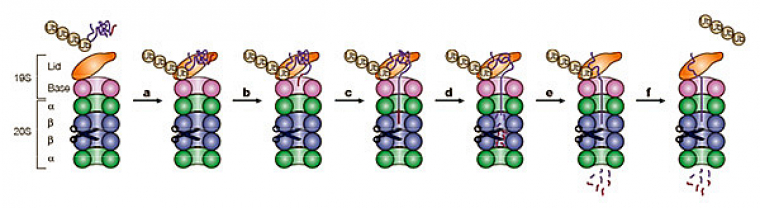
Reconnaissance des substrats
La majorité des substrats du protéasome sont étiquetés par une chaîne de poly-ubiquitine, qui joue probablement un rôle important dans la reconnaissance des substrats par le protéasome (Fig.12a). La sous-unité Rpn10 de la base du 19S est capable d’interagir avec les chaînes d’ubiquitine impliquant les lysines K48 et K63 de l’ubiquitine, avec la même affinité. L’affinité est augmentée lorsque la taille de la chaîne d’ubiquitine augmente, avec un minimum de 4 molécules d’ubiquitine, ce qui peut s’expliquer par l’organisation des chaînes de poly-ubiquitine en modules de tétra-ubiquitine qui pourraient servir d’unités de reconnaissance par le protéasome. Néanmoins, si la spécificité de Rpn10 pour les chaînes longues de préférence est similaire à celle du 19S, en absence de Rpn10, le protéasome est toujours capable de lier les conjugués d’ubiquitine, ce qui est en accord avec l’existence de récepteurs d’ubiquitine additionnels intrinsèques au protéasome. D’ailleurs, il semblerait qu’une ATPase, la sous-unité Rpt5, soit capable de lier la chaîne d’ubiquitine, même si le motif d’interaction de Rpt5 avec l’ubiquitine reste à identifier. Dans ce cas, vu son appartenance à la base, Rpt5 ne fonctionnerait pas en parallèle de Rpn10 ou d’autres récepteurs de l’ubiquitine, mais plutôt en aval. En outre, la sous-unité Rpn13 du 19S a également été caractérisée comme un récepteur à l’ubiquitine.
Rpn10 contient dans sa partie C-terminale un domaine UIM (pour Ubiquitine Interacting Motif) qui permet son interaction avec l’ubiquitine et dans sa partie N-terminale un domaine VWA (un motif structural identifié dans le facteur de Von WillebrAnd) impliqué dans l’interaction avec les autres sous-unités du 19S. Comme évoqué précédemment, la sous-unité Rpn10 est la seule sous-unité trouvée en grande proportion sous forme monomérique non associée au protéasome, au moins chez certaines espèces et on peut penser que sa surexpression entraîne la séquestration de substrats poly-ubiquitylés les empêchant ainsi d’atteindre le protéasome et entraîner ainsi une accumulation de ces substrats ubiquitylés. Cependant, la délétion du domaine UIM de Rpn10 ne développe pas de phénotype de défaut de protéolyse ni d’accumulation de substrats ubiquitylés dans des conditions normales, alors que la délétion de son extrémité N-terminale provoque un phénotype similaire à la délétion de la protéine entière, à savoir un défaut de la dégradation et une sensibilité accrue aux analogues des acides aminés. Ceci suggère que les fonctions de Rpn10 au niveau du protéasome sont indépendantes de sa capacité à lier les chaînes d’ubiquitine et semble indiquer que d’autres sous-unités du protéasome peuvent interagir avec les chaînes d’ubiquitine. Toutefois une étude plus récente a montré que la délétion de la sous-unité Rpn10 affecte la capacité du complexe 20S-19S de lier des conjugués d’ubiquitine ainsi que des chaînes libres, ce qui suggère au contraire que la sous-unité Rpn10 contribue à la reconnaissance des chaînes d’ubiquitine par le protéasome et que la capacité de liaison des chaînes d’ubiquitine par Rpn10 est importante et a probablement une signification physiologique.
Dans sa partie N-terminale, Rpn13 possède un domaine PRU (pour Pleckstrin-like Receptor for Ubiquitine), capable de lier avec une très forte affinité les tétramères d’ubiquitine, mais également des monomères ou des dimères d’ubiquitine, ainsi que la sous-unité Rpn2. Certains substrats mono-ubiquitylés peuvent également être ciblés au protéasome et leur reconnaissance pourrait dépendre tout particulièrement de Rpn13, même si Rpn10 peut également lier une protéine mono-ubiquitylée. Il faut noter que les interactions de Rpn13 avec Rpn2 et l’ubiquitine ne sont pas exclusives, ce qui confirme que Rpn13 est un récepteur intrinsèque du 19S à l’ubiquitine. Néanmoins, il semblerait que l’affinité de Rpn13 soit dirigée contre un dimère d’ubiquitine au sein d’un tétramère et que cette affinité soit 15 et 30 fois inférieure à l’affinité de Rad23 et Rpn10, respectivement, pour un tétramère d’ubiquitine, ce qui suggèrerait que le rôle de Rpn13 soit principalement l’aide au positionnement des chaînes d’ubiquitine en vue de leur désassemblage.
Enfin, il existe des protéines à domaines UBL/UBA, comme Rad23 ou KPC2, qui ont donc une organisation similaire à celle de la sous-unité Rpn10 avec ses motifs VWA et UIM, mais qui ne font pas partie du protéasome à proprement parler. Ces protéines pourraient jouer un rôle dans l’adressage des substrats au protéasome. On peut noter que les fonctions de Rad23 et Rpn10 semblent se chevaucher, au regard de l’aggravation des phénotypes chez les double-mutants de délétion. Une certaine redondance fonctionnelle a également été suggérée entre Rpn13 et Rpn10. Par ailleurs, des études chez la levure montrent que si la délétion du couvercle n’affecte pas la dégradation indépendante de l’ubiquitine, le couvercle est nécessaire à la dégradation des substrats ubiquitylés, ce qui laisse penser que le couvercle contient des sous-unités importantes pour la reconnaissance des chaînes de poly-ubiquitine et/ou pour la déubiquitylation.
Par ailleurs, certaines sous-unités doivent être capable d’interagir avec des substrats indépendamment de l’ubiquitine puisque la dégradation de certains substrats du protéasome ne nécessitent pas leur ubiquitylation préalable, comme c’est le cas pour ODC, p21 ou encore c-fos. La base pourrait servir de plateforme d’interaction avec les substrats du protéasome et permettrait leur positionnement adéquat pour les étapes suivantes de dénaturation et de translocation. En effet, des études sur les protéasomes eucaryotes ont montré que la base peut interagir avec des substrats non-ubiquitylés dénaturés et le complexe archaebactérien PAN interagit avec des protéines natives/repliées et est capable de les dénaturer.
En conclusion, le protéasome 26S peut reconnaître ses substrats à la fois via des récepteurs intrinsèques (Rpn10, Rpn13 et Rpt5) mais aussi des récepteurs extrinsèques qui s’associent de façon réversible et transitoire avec le protéasome, et il n’est pas impossible qu’un substrat soit reconnu de manière coordonnée par plusieurs récepteurs.
Dénaturation des substrats
Les substrats doivent être dénaturés pour être transférés à l’intérieur du 20S (Fig.12b et c), car, même une fois ouvert, le canal du 20S est encore trop étroit pour laisser entrer des protéines sous leur forme native. Outre le besoin d’ATP pour l’association du 19S et du 20S, la protéolyse des protéines par le protéasome nécessite l’hydrolyse d’ATP. Par homologie avec les protéines chaperones, on peut penser que cette énergie permet des cycles de haute et de basse affinité du 19S pour les substrats, permettant ainsi de les maintenir dans une conformation dénaturée pour qu’ils puissent être transférés à l’intérieur du 20S. Ces fonctions (dépliement et translocation des substrats) sont probablement assurées par la base, du fait de sa localisation et de la présence des 6 ATPases à son niveau. Cette hypothèse est soutenue par le fait que le 20S associé à la base est aussi efficace que le 26S pour la dégradation des substrats non-ubiquitylés et par homologie avec le complexe PAN qui est capable de dénaturer des substrats non ubiquitylés et de les transloquer dans le 20S de manière ATP-dépendante. Il faut noter que pour des substrats non ubiquitylés et non structurés, tels que p21 et la caséine, il a été montré que leur dégradation ne nécessitait pas l’hydrolyse de l’ATP, suggérant que le transit de ces protéines aux sites catalytiques se fait par un processus passif et que la translocation par elle-même ne nécessite pas d’être couplée à l’hydrolyse de l’ATP. De même, chez les archaebactéries, l’association du 20S et du complexe PAN ainsi que la dégradation de protéines non structurées ne nécessite pas l’hydrolyse de l’ATP.
D’autre part, des études faites in vitro montrent que la base du protéasome eucaryote peut lier des protéines dénaturées et les relarguer de manière ATP-dépendante, mais peut aussi les renaturer, ce qui dénote des propriétés chaperones, et laisse présager de leur capacité à dénaturer les protéines natives. Toutefois, malgré les activités chaperone-like intrinsèques du protéasome, de nombreux résultats montrent que des protéines chaperones agissent de concert avec le protéasome pour son bon fonctionnement, comme c’est le cas pour la protéine Hsp90 qui est souvent copurifiée avec le protéasome. Ces chaperones peuvent promouvoir l’ubiquitylation de substrats mal repliés et aider à l’adressage et la présentation des substrats au protéasome.
Déubiquitylation des substrats
Une fois reconnu par le protéasome 26S, le substrat poly-ubiquitylé est déplié et transloqué au travers du canal du 20S vers la cavité protéolytique. De façon concomitante, le substrat est déubiquitylé (Fig.12f). Comme évoqué précédemment, la déubiquitylation des substrats au niveau du protéasome est importante pour plusieurs raisons, non seulement pour le recyclage de l’ubiquitine, mais également du point de vue du fonctionnement du protéasome. De fait, la chaîne formée de 4 ubiquitines qui constitue, dans la majorité des cas, le signal minimal de dégradation, a un diamètre d’environ 28 Å, alors que le diamètre du pore axial du protéasome 20S n’excède pas 20 Å : sans la déubiquitylation, cette chaîne bloquerait la translocation du substrat à l’intérieur du 20S. De plus, les chaînes libres d’ubiquitine doivent être éliminées : sans cela, elles pourraient inhiber le protéasome 26S en entrant en compétition avec les substrats ubiquitylés. Ainsi, la déubiquitylation doit être étroitement couplée à la dégradation et plusieurs activités de déubiquitylation ont été identifiées comme associées au protéasome.
L’association 19S-20S permet d’ouvrir le pore axial du protéasome 20S. Le complexe régulateur 19S, associé à des protéines accessoires du protéasome (non représentées) assure la reconnaissance du substrat lié à une chaîne de poly-ubiquitine (a). Le 19S permet alors le dépliement du substrat (b) et sa translocation (c) vers la chambre protéolytique du 20S (d) où il est hydrolysé en petits peptides (e). Le substrat est déubiquitylé au cours du processus de dégradation avant l’entrée dans la chambre catalytique.
D’après (Pickart & Cohen, 2004).
La sous-unité du couvercle Rpn11, le représentant majeur de la famille des métalloprotéases MPN+/JAMM, présente une activité de déubiquitylation, mais uniquement quand elle est associée au complexe régulateur 19S. L’étude d’un mutant au niveau de son site catalytique a montré que, chez la levure, cette sous-unité est essentielle et que, in vitro, elle est nécessaire à la déubiquitylation et à la dégradation des substrats ubiquitylés par le protéasome 26S. Ceci associé au fait que son activité est ATP-dépendante suggère que son activité est couplée à l’activité ATPasique de la base, appuyant le fait que la déubiquitylation des substrats soit couplée à leur dégradation.
Une autre isopeptidase serait également une sous-unité intrinsèque du complexe régulateur 19S. Elle se distingue de la sous-unité Rpn11 par sa sensibilité à l’ubiquitine aldéhyde et par le fait qu’elle ne soit pas affectée par l’ATP. Cette DUB serait capable de cliver les molécules d’ubiquitine en attaquant la chaîne par l’extrémité distale. Cette activité aurait un rôle de contrôle, permettant d’empêcher la protéolyse de protéines conjuguées par des chaînes courtes, qui ont été adressées au protéasome de façon inappropriée et qui ne sont à priori pas destinées à être dégradées, en les relâchant avant que le processus de dégradation ne commence. Mais cette enzyme est un membre de la famille UCH, alors qu’il est clair que beaucoup des activités de déubiquitylation du protéasome sont portées par des protéines de la famille UBP, ce qui suggère que les activités de déubiquitylation du protéasome soient assurées par plus d’une protéine.
Par ailleurs, associée au protéasome dans des proportions quasiment stoechiométriques dans les cellules, on trouve l’enzyme UBP6, qui s’associe probablement au niveau de la sous-unité Rpn1 du protéasome via un domaine UBL N-terminal. Son activité isopeptidasique est stimulée d’un facteur 300 environ quand elle est associée au complexe régulateur 19S du protéasome 26S. Des études de mutants de délétion de UBP6 chez la levure ont montré que l’ubiquitine est déstabilisée en absence d’UBP6, et ce de manière protéasome-dépendante, suggérant que l’ubiquitine serait ainsi dégradée en même temps que le substrat. Alors que Rpn11 déubiquityle « en bloc » les substrats, UCH37 et UBP6 déubiquitylent graduellement les substrats (une ubiquitine à la fois pour UCH37, une ou plusieurs pour UBP6), et on peut envisager que ces DUB servent de « contrôle qualité » qui permet de n’adresser au protéasome que les substrats dont les chaînes sont suffisamment longues.
D’autre part, il a été suggéré que l’enzyme Doa4 soit associée au protéasome, via son extrémité N-terminale, et que cette association joue un rôle fonctionnel au niveau de son activité isopeptidasique qui permet le clivage de l’ubiquitine directement liée au substrat. Toutefois, on la retrouve en proportion sub-stœchiométrique par rapport au protéasome, ce qui suggère qu’elle est impliquée dans la déubiquitylation d’autres substrats que ceux liés au protéasome. On la retrouve en effet impliquée dans la déubiquitylation des substrats adressés à la vacuole après leur internalisation.
Enfin, l’isopeptidase T travaille probablement en aval de toutes ces activités, digérant les chaînes d’ubiquitine libres libérées par le protéasome ou par d’autres isopeptidases en molécules d’ubiquitine monomériques. Cette activité a le double rôle de recycler l’ubiquitine et de débarrasser la cellule des chaînes libres d’ubiquitine qui peuvent, lorsqu’elles s’accumulent, inhiber le protéasome.
Ouverture du pore
La délétion de la queue N-terminale de la sous-unité α3 décrite précédemment, si elle active fortement le protéasome 20S, n’a pas d’effet notoire sur le 26S, ce qui suggère que le 19S joue bien un rôle dans l’ouverture du 20S. Cet effet a été imparti aux ATPases du 19S, Rpt2 et Rpt5, via leur motif conservé HbYX (pour Hydrophobic-Tyrosine-X) au niveau de leurs extrémités C-terminales, qui permet l’ouverture du canal du 20S via un mécanisme différent que celui opéré par le régulateur 11S.
L’implication de la sous-unité Rpt2 dans l’ouverture du pore du 20S avait déjà été suggérée car une mutation ponctuelle de cette sous-unité affecte sévèrement l’activation du protéasome 20S par le 19S en raison d’un défaut d’ouverture. Ce mécanisme a été confirmé par l’analyse de la structure tridimensionnelle du complexe 20S-19S qui a montré que la liaison du complexe régulateur 19S lui-même provoque un déplacement radial des sous-unités α, adjacentes au 19S, qui peut également être transmise à l’intérieur de la structure du 20S, avec un déplacement des sous-unités β, permettant un accès plus large aux sites catalytiques.
Par ailleurs, si la régulation du 20S par ces régulateurs positifs (activateurs) est admise, il semblerait que les régulateurs eux-mêmes puissent être régulés par le 20S dans un mécanisme de contrôle réciproque. De fait, il a été montré que des inhibiteurs du protéasome sont capables de stabiliser l’interaction du 20S et du 19S au sein du protéasome 26S par engagements des sites protéolytiques du 20S, suggérant que les sites actifs du 20S puissent communiquer avec le régulateur 19S, probablement par un changement de conformation des sous-unités β qui mènerait à la modification de l’interface de liaison avec le 19S au niveau de la surface distale de l’anneau des sous-unités α. De plus, ce processus de régulation mutuelle entre les ATPases et les protéases par des changements allostériques semble être conservé entre les eucaryotes et les bactéries. En effet, chez la bactérie, un tel mécanisme a été observé pour les complexes HslVU, ClpXP et ClpAP, qui présentent de grandes similarités avec le protéasome 26S, à savoir une protéase (HslV ou ClpP) associée à une ATPase hexamérique (HslU ou ClpX et ClpA). Dans le cas de HslVU, il a été montré que la liaison de l’inhibiteur au niveau des sites catalytiques de HslV induit également une forte interaction entre HslV et HslU, mais aussi une activation des ATPases HslU, suggérant, ici aussi, que HslV « lié à un substrat » peut réguler de façon allostérique la fonction de HslU. De même, pour la protéase ClpP, une forme modifiée de ClpP qui mime sa liaison à un substrat présente une affinité plus forte pour ses régulateurs ClpX et ClpA.
Autres rôles du 19S et/ou du 26S
Un autre rôle intéressant du protéasome est son activité de « processing ». En effet, dans certains cas, le protéasome ne dégrade pas entièrement le substrat, mais permet la production d’une forme tronquée du précurseur. Cette activité est un mécanisme de régulation qui permet de faire passer une protéine d’une forme à une autre, et modifier ainsi ses activités cellulaires.
L’exemple le plus documenté est celui du précurseur p105 de la protéine p50, un constituant du facteur de transcription NF-kB. Dans un premier temps, la protéine p105 est ubiquitylée, au niveau de son extrémité C-terminale, puis dans un second temps, la moitié C-terminale est dégradée par le protéasome, et enfin, la région N-terminale de 50 kDa est relâchée comme une sous-unité stable et active du facteur de transcription NF-kB. La région riche en glycines (GRR pour Glycin-Rich Region) située au milieu de la protéine p105 pourrait servir de “signal” pour la dégradation partielle de la protéine. Cette séquence et les interactions spécifiques de certains acides aminés au niveau du domaine p50, qui stabilisent la structure tridimensionnelle et empêchent sa dénaturation, permettent de déterminer quelle région doit être dégradée. Il existe d’autres exemples de maturation protéolytique, comme celui du précurseur p100 de la protéine NF-kB2 ou celui du précurseur p120 du facteur de transcription Spt23, ce qui montre que ce mécanisme occupe une place importante dans les fonctionnalités du protéasome.
Par ailleurs, d’autres fonctions non protéolytiques de type chaperon ou d’ancrage qui pourraient participer aux régulations de l’organisation de la chromatine ou au recrutement de facteurs de transcription ont été attribuées au protéasome 26S et plus particulièrement aux sous-unités ATPases du complexe régulateur 19S. On trouve par exemple le protéasome impliqué dans le désassemblage et le réarrangement du complexe de réparation d’excision nucléaire, indépendamment de la protéolyse et les sous-unités du 19S qui colocalisent in vivo avec les protéines chaperones aux sites des agrégats de récepteurs aux androgènes mal repliés.
Les complexes 20S-11S
Composition et structure du 11S
L’activateur du protéasome 11S a initialement été découvert dans des lysats de cellules sanguines humaines et bovines comme un facteur capable d’activer fortement l’hydrolyse de petits peptides fluorogéniques, mais incapable de stimuler la dégradation de protéines natives, ubiquitylées ou non. Toutefois, pour certains peptides, la stimulation du protéasome 20S par l’activateur 11S peut avoisiner les 50 fois, ce qui suggère un rôle important de cet activateur sur l’activité du protéasome. Cet activateur 11S regroupe en fait une famille de protéines de 28 kDa dénommés PA28 ou 11S REG qui comprend trois membres homologues : PA28 α, PA28 β et PA28 γ (Fig. 13).
|
Sous-unités PA28 α et β |
Sous-unité PA28 γ |
|
|
Ces protéines forment des anneaux multimériques qui peuvent s’associer à l’anneau des sous-unités α du protéasome 20S. Tandis que PA28 γ forme des homo-heptamères in vitro et in vivo, les sous-unités α sont capables de former des homo-heptamères in vitro, mais elles sont le plus souvent retrouvées en complexes avec les sous-unités β. Les sous-unités PA28 α et β peuvent s’assembler en hétéro-hexamères (α3β3) ou en hétéro-heptamères (α3β4). Néanmoins de nombreux arguments physiques et biochimiques sont en faveur d’une association des sous-unités α et β en heptamères, ce qui est en accord avec son association à l’anneau alpha du protéasome 20S qui est constitué de 7 sous-unités.
La liaison de PA28 aux anneaux externes du protéasome 20S provoque une ouverture du pore d’entrée et de sortie du protéasome, avec un alignement du canal central formé à travers le centre de PA28 avec celui du protéasome. Ces données structurales suggèrent que l’ouverture du pore permet aux peptides substrats de diffuser au travers du canal central de PA28 et à l’intérieur du protéasome. Bien que ceci puisse expliquer l’activation, il est plus probable de penser que l’hydrolyse des peptides soit modulée par des changements de conformation induits aux sites catalytiques. D’ailleurs, PA28 αβ etPA28 γ ne stimulent pas les activités catalytiques du protéasome de la même façon. PA28 αβ stimule les 3 activités chymotryptiques-, tryptiques – et caspases-like du protéasome, alors que PA28 γ stimule uniquement l’activité tryptique-like. De plus, les complexes PA28 γ, contrairement à PA28 αβ, ont une structure plus rigide, entraînant des changements de conformation du protéasome plus importants et une inhibition des activités chymotryptiques – et caspases-like.
Bien que les propriétés biochimiques de PA28 soient connues, ses rôles précis sont peu clairs et controversés. L’absence de PA28 chez la levure suggère que PA28 ne participe pas à un processus cellulaire fondamental, mais qu’il contribue probablement à une fonction biologique propre aux eucaryotes supérieurs. La localisation de PA28 αβ au niveau des tissus immuns, son induction par l’interféron gamma et sa capacité à stimuler l’hydrolyse des peptides par le protéasome semblent indiquer un rôle de PA28 αβ dans la réponse immune et notamment dans la génération et la présentation des peptides antigéniques, ce qui semble beaucoup moins clair pour PA28 γ.
PA28 αβ : rôle dans la réponse immune
L’immunité chez les vertébrés implique deux voies distinctes : la réponse humorale, caractérisée par des anticorps circulants dirigés contre des épitopes générés dans les endosomes et présentés par les molécules du CMH de classe II, et la réponse cellulaire, médiée par les lymphocytes T cytotoxiques (CTL), qui détruisent les cellules infectées, après reconnaissance de celles-ci grâce aux épitopes étrangers générés dans leur cytosol et présentés à leur surface par les CMH de classe I. Cette classe de peptides, d’une taille optimale de 8 à 11 résidus, est essentiellement générée par le protéasome. Après leur production par le protéasome, ils sont ensuite transportés dans la lumière du réticulum endoplasmique où ils sont pris en charge par les molécules du CMH de classe I pour être exposés à la surface de la cellule. Comme évoqué précédemment, les corrélations entre induction à l’interféron gamma et localisation dans les tissus immuns des complexes PA28 αβ, suggèrent un rôle de ces complexes dans la présentation des antigènes.
Trois théories quant au rôle que PA28 αβ pourrait jouer dans la réponse immune ont été développées.
La première porte sur un changement de la taille des peptides générés : le protéasome est capable de générer des peptides d’une taille allant de 3-4 à plus de 30 acides aminés, avec néanmoins une prédominance de peptides de 6-8 résidus, ce qui est trop court pour une liaison efficace sur les molécules du CMH de classe I. Etant donné que la structure du protéasome 20S-11S montre que l’association avec PA28 αβ augmente le diamètre de l’ouverture de l’anneau alpha du protéasome, ceci pourrait faciliter la sortie de peptides plus longs (Fig.14b). Cependant, une étude a montré que PA28 αβ n’augmente pas la taille des peptides générés par le protéasome 26S.
La deuxième porte sur une altération des sites de clivage : l’association avec PA28 αβ, en imposant des changements de conformation importants et en modifiant de manière allostérique la spécificité des sites catalytiques, pourrait modifier les sites de clivage par le protéasome au sein d’un polypeptide, générant ainsi des épitopes uniques (Fig. 14a).
Enfin, la troisième porte sur l’assemblage de l’immuno-protéasome : PA28 αβ favoriserait l’assemblage de l’immuno-protéasome, mais ce dernier point est assez controversé, au vu des résultats obtenus par deux études d’inactivation des gènes PA28 α et β chez la souris. Chez un groupe, il a été observé une déficience importante de la réponse immune médiée par les CTL, associée à une très forte diminution de la proportion d’immuno-protéasome, suggérant un rôle de PA28 αβ dans le recrutement des sous-unités βi pour l’assemblage de l’immuno-protéasome. Chez l’autre groupe, la réponse immune n’est quasiment pas affectée et la composition en immuno-protéasome correcte, avec toutefois, une déficience dans la production de certains épitopes. Enfin, le fait que la surexpression de PA28 αβ dans des fibroblastes ait un effet positif sur la présentation des antigènes, indépendamment de la quantité d’immuno-protéasome, semble infirmer le rôle de PA28 αβ dans l’assemblage de l’immuno-protéasome. Par contre, des travaux ont montré, par l’étude du rôle de Hsp90 dans la production d’un épitope particulier, dans des cellules PA28 αβ-/- et PA28 αβ+/+, que la protéine Hsp90 et le complexe PA28 αβ pouvaient tous deux servir de chaperones et assurer la prise en charge et le transfert des peptides issus du protéasome aux molécules du CMH de classe I au niveau de la membrane du réticulum endoplasmique (Fig. 14c).
(a) Modification du site de coupure.
(b) Facilitation de la sortie des peptides de 8 à 11 acides aminés.
(c) Prise en charge et transfert des peptides au réticulum endoplasmique.
D’après (Rechsteiner & Hill, 2005).
Si plusieurs corrélations sont en faveur d’un rôle du complexe PA28 αβ dans la présentation des antigènes de classe I, les nombreuses études menées à ce sujet n’ont pas permis de clarifier ni son rôle exact dans cette voie, ni des propriétés spécifiques des complexes 20S-PA28 αβ par rapport au 26S. Par ailleurs, les complexes PA28 n’étant pas considérés comme capables de dégrader des protéines, leurs fonctions sont élaborées sur le modèle de protéasomes hybrides, où un complexe 19S à une extrémité contrôle l’accès des substrats protéiques aux sites catalytiques, et un complexe PA28 αβ à l’autre extrémité augmente l’efficacité de la protéolyse et contrôle le devenir des produits de dégradation et/ou la localisation du complexe. Cependant dans la cellule, il existe des complexes 11S-20S-11S, dans lesquels les deux extrémités du 20S sont associés à un 11S dont on imagine mal la fonction du fait qu’ils sont incapables de dégrader des protéines ubiquitylées in vitro. Ces conclusions sont sans compter que PA28 αβ pourrait avoir des fonctions indépendantes de la réponse immune, étant donné que les taux de PA28 αβ peuvent être affectés dans de nombreuses conditions physiologiques et pathologiques qui ne semblent pas être liées à la réponse immunitaire.
PA28 γ
Contrairement aux nombreuses données relatives aux fonctions de PA28 αβ dans la réponse immune, les propriétés de PA28 γ ne semblent pas le prédisposer à une fonction particulière, même si un rôle dans la progression du cycle cellulaire et dans l’apoptose est envisagé.
Des études de knock-out du gène PA28 γ chez la souris et d’ARN interférence chez la Drosophile suggèrent un rôle dans la progression du cycle cellulaire et dans l’apoptose. En effet, les souris résultantes sont de petite taille, et les fibroblastes issus de leurs embryons présentent une accumulation en phase G1. La petite taille des souris résulte en partie d’une augmentation de l’apoptose, ce qui suggère un rôle anti-apoptotique de PA28 γ. Ce rôle hypothétique est renforcé par des études de double-hybride qui ont permis de mettre en évidence l’interaction de PA28 γ avec plusieurs protéines impliquées dans l’apoptose (MEKK3, FLASH, Daxx, RanBPM et PIAS1). De même, les cellules de Drosophile traitées par les ARNs interférants dirigés contre PA28 γ présentent un défaut de prolifération et s’accumulent en phase G1. De plus, PA28 γ a récemment été identifié comme un acteur principal de la dégradation de l’oncogène SRC-3 et de la protéine p21 CKI et il a également montré comme un nouveau régulateur de l’intégrité des corps de Cajal suite à un stress génotoxique. Enfin, il a été montré que PA28 γ est présent dans les speckles nucléaires (NS, pour Nuclear Speckles), où il forme un complexe actif avec le protéasome 20S. Cette étude a montré que les cellules où l’expression de PA28 γ est inhibée présentent une altération dans la morphologie des NS et semble impliquer PA28 γ dans l’organisation des NS et dans le trafic intranucléaire des facteurs d’épissage.
En ce qui concerne le mode d’action de PA28 γ, toutes les hypothèses sont ouvertes : facilitation de la dégradation de certains substrats spécifiques (pro-apoptotiques, par exemple ?), rôle transcriptionnel (localisation strictement nucléaire de PA28 γ ?), inhibition par interaction directe avec des facteurs régulant l’apoptose et/ou le cycle cellulaire ?
Il est clair que si le complexe 19S se compose de sous-unités essentielles à la dégradation des protéines, les complexes PA28 en sont dépourvus et il est difficile d’envisager comment ils pourraient activer les fonctions protéolytiques du 20S contre des protéines de manière autonome, même s’ils activent l’ouverture du pore. Ainsi, si certaines voies dans lesquelles les complexes PA28 interviennent ont pu être identifiées, les mécanismes d’action de ces complexes sont loin d’être compris.
Localisation des protéasomes
Le rôle majeur de la dégradation dépendante du système UbPr nécessite que ce système soit très finement régulé. Cette régulation se fait en partie par l’étiquetage des protéines avec des chaînes particulières d’ubiquitine et par l’association du protéasome avec ses différents régulateurs, mais la localisation cellulaire du protéasome semble aussi être un point majeur de sa régulation. Aussi, si le protéasome est ubiquitaire et abondant, de nombreuses études ont montré qu’il pouvait avoir des localisations particulières, importantes pour ses fonctions. Ces études ont montré que le protéasome a une localisation nucléo-cytoplasmique, essentiellement diffuse, avec exclusion vacuolaire et nucléolaire, à certaines exceptions près.
Cette localisation a été confirmée par l’étude de lignées cellulaires exprimant une sous-unité inductible du protéasome fusionnée à une protéine fluorescente, LMP2-GFP, qui montre en plus que le protéasome est transporté de manière unidirectionnelle du cytoplasme vers le noyau de la cellule puis redistribué uniformément dans toute la cellule lors de la rupture de l’enveloppe nucléaire au cours de la prophase. La présence de séquences de localisation nucléaire (NLS) chez certaines sous-unités du protéasome et la phosphorylation de ces mêmes sous-unités peuvent servir à réguler ce transport. Chez la levure, il semble que l’import nucléaire du protéasome soit couplé à son assemblage. Toutefois, il est peu probable que ce transport unidirectionnel du cytoplasme vers le noyau soit conservé dans des cellules post-mitotiques, telles que les neurones.
Distribution nucléaire
Il existe des structures subnucléaires, appelés clastosomes, où on trouve du protéasome 20S, son complexe régulateur 19S, de l’ubiquitine et un certain nombre de substrats du système UbPr. Le nombre de ces structures, faible en conditions normales, est augmenté lorsque la protéolyse dépendante du protéasome est activée. Par contre, ces structures disparaissent suite au traitement des cellules avec des inhibiteurs du protéasome. Par ailleurs, des complexes 20S-PA28 γ sont présents au niveau des speckles nucléaires (structures subnucléaires enrichies en facteurs d’épissage des ARN), où ils semblent impliqués dans l’organisation de ces structures nucléaires.
Il existe d’autres foyers dans lesquels le protéasome peut être concentré, parfois uniquement dans certaines pathologies. Par exemple, une fraction des protéasomes, ainsi que son régulateur PA28 αβ, sont associés avec les corps PML (pour ProMyelocytic Leukemia) trouvés dans les cellules nerveuses de malades touchés par l’ataxie spinocérébelleuse de type 7, une maladie neurodégénératives caractérisée par l’accumulation de protéines abérentes portant de nombreuses glutamines. De plus, les concentrations de PA28 αβ et de l’immuno-protéasome sont augmentées à ce niveau suite à un traitement des cellules à l’interféron γ.
Distribution cytoplasmique
Au niveau du cytoplasme, on trouve du protéasome et des substrats du protéasome concentrés dans une région entourant le centrosome. Cette région a même été proposée comme étant un lieu préférentiel de la dégradation protéasomale et, de ce fait, a été appelée « centre protéolytique ». Par la suite, il a été montré que des agrégats de protéasomes et de substrats ubiquitylés du protéasome, appelés « aggrésomes », se formaient à ce niveau, suite à une inhibition du protéasome ou à une surexpression de substrats du protéasome. Cet aggrésome permettrait de regrouper les protéines délétères afin de limiter leurs effets toxiques dans la cellule. Toutefois, l’activité du protéasome au niveau de cette structure semble inhibée et la concentration de protéasome à ce niveau ne serait que la conséquence du fait que ces protéines sont des substrats du système UbPr. Chez la levure, il a été montré que, lorsque les cellules entrent en quiescence, les sous-unités du protéasome se relocalisent massivement du noyau dans des structures cytoplasmiques mobiles, qui servent de réservoirs cytoplasmiques de protéasomes, qui peuvent être rapidement mobilisés lorsque les cellules sortent de quiescence.
Le protéasome semble s’accumuler dans des régions particulières : une fraction du protéasome cytoplasmique est étroitement associée à différents éléments du cytosquelette, en particulier aux filaments intermédiaires. Une fraction du protéasome est également associée au niveau de la surface externe du réticulum endoplasmique, où il est probablement impliqué dans le système ERAD (Endoplasmic Reticulum Associated Degradation), qui consiste en un processus d’export rétrograde de protéines du réticulum endoplasmique mal conformées ou anormales vers le cytosol, couplé à leur ubiquitylation et leur dégradation par le protéasome. De manière intéressante, la fraction de protéasome associée à ce niveau est enrichie en immuno-protéasome, ce qui est cohérent avec le modèle de prise en charge directe des peptides délivrés par l’immuno-protéasome par le transporteur TAP, qui transporte les peptides dans la lumière du réticulum endoplasmique, où ils s’associent avec les molécules du CMH de classe I.
Le cas particulier de la mitose
Au cours de la mitose, on a vu que le protéasome se redistribue suite à la rupture de l’enveloppe nucléaire, mais il occupe également des localisations particulières : au début de la prophase, il s’accumule autour des chromosomes condensés, avec exclusion de la chromatine tout au long de la mitose, puis il se localise au niveau du fuseau mitotique. En effet, il s’associe aux pôles du fuseau au cours de la métaphase et de l’anaphase, puis au « mid-body » au cours de la télophase. Cette localisation semble corrélée avec la dégradation de la cycline B au cours du cycle.
Localisation du protéasome et type cellulaire
Enfin, il a été rapporté un certain nombre de localisations remarquables du protéasome selon le type cellulaire et son état de différenciation (exemple des myoblastes au cours de la différenciation musculaire, ou des différents types de cellules du système nerveux central), ce qui suggère que la distribution intracellulaire du protéasome refléterait le besoin d’une protéolyse augmentée et/ou diminuée de substrats spécifiques au niveau des différentes régions intracellulaires, même si on ne peut exclure une fonction non-protéolytique de ces protéasomes.
Bibliographie
- Amerik AY, Hochstrasser M (2004) Mechanism and function of deubiquitinating enzymes. Biochim Biophys Acta 1695(1-3) : 189-207
- Ardley HC, Robinson PA (2005) E3 ubiquitin ligases. Essays Biochem 41 : 15-30
- Baumeister W, Walz J, Zuhl F, Seemuller E (1998) The proteasome : paradigm of a self-compartmentalizing protease. Cell 92(3) : 367-380
- Coux O, Tanaka K, Goldberg AL (1996) Structure and Functions of the 20S and 26S Proteasomes. Annual Review of Biochemistry 65 : 801-847
- Groll M, Clausen T (2003) Molecular shredders : how proteasomes fulfill their role. Curr Opin Struct Biol 13(6) : 665-673
- Hershko A, Ciechanover A, Heller H, Haas AL, Rose IA (1980) Proposed role of ATP in protein breakdown : conjugation of protein with multiple chains of the polypeptide of ATP-dependent proteolysis. Proc Natl Acad Sci U S A 77(4) : 1783-1786
- Hicke L, Schubert HL, Hill CP (2005) Ubiquitin-binding domains. Nat Rev Mol Cell Biol 6(8) : 610-621
- Hoppe T (2005) Multiubiquitylation by E4 enzymes : 'one size' doesn't fit all. Trends in biochemical sciences 30(4) : 183-187
- Hurley JH, Lee S, Prag G (2006) Ubiquitin-binding domains. Biochem J 399(3) : 361-372
- Mukhopadhyay D, Riezman H (2007) Proteasome-independent functions of ubiquitin in endocytosis and signaling. Science 315(5809) : 201-205
- Nijman SM, Luna-Vargas MP, Velds A, Brummelkamp TR, Dirac AM, Sixma TK, Bernards R (2005) A genomic and functional inventory of deubiquitinating enzymes. Cell 123(5) : 773-786
- Ortega J, Bernard Heymann J, Kajava AV, Ustrell V, Rechsteiner M, Steven AC (2005) The Axial Channel of the 20S Proteasome Opens Upon Binding of the PA200 Activator. J Mol Biol 346(5) : 1221-1227
- Pickart CM, Cohen RE (2004) Proteasomes and their kin : proteases in the machine age. Nat Rev Mol Cell Biol 5(3) : 177-187
- Pickart CM, Fushman D (2004) Polyubiquitin chains : polymeric protein signals. Current opinion in chemical biology 8(6) : 610-616
- Ravid T, Hochstrasser M (2008) Diversity of degradation signals in the ubiquitin-proteasome system. Nat Rev Mol Cell Biol 9(9) : 679-690
- Rechsteiner M, Hill CP (2005) Mobilizing the proteolytic machine : cell biological roles of proteasome activators and inhibitors. Trends in cell biology 15(1) : 27-33
- Sharon M, Taverner T, Ambroggio XI, Deshaies RJ, Robinson CV (2006a) Structural Organization of the 19S Proteasome Lid : Insights from MS of Intact Complexes. PLoS Biol 4(8) : e267
- Simpson MV (1953) The release of labeled amino acids from the proteins of rat liver slices. The Journal of biological chemistry 201(1) : 143-154
- Unno M, Mizushima T, Morimoto Y, Tomisugi Y, Tanaka K, Yasuoka N, Tsukihara T (2002) The Structure of the Mammalian 20S Proteasome at 2.75 A Resolution. Structure (Camb) 10(5) : 609-618.
- Welchman RL, Gordon C, Mayer RJ (2005) Ubiquitin and ubiquitin-like proteins as multifunctional signals. Nat Rev Mol Cell Biol 6(8) : 599-609
- Zwickl P, Baumeister W (1999) AAA-ATPases at the crossroads of protein life and death. Nat Cell Biolog 1(4) : E97-E98