Présentation de la technique d'invalidation d'un gène (knock-out), de sa mise en œuvre et de son intérêt.
Exemple de l'invalidation des gènes HOX8.
Pourquoi invalider un gène ?
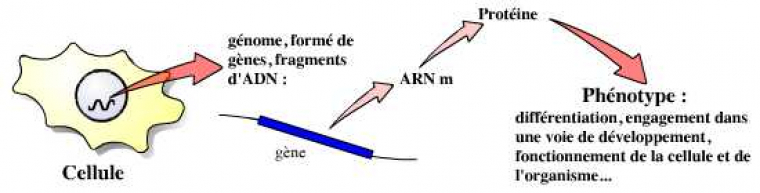
Les gènes sont à la base de la vie de la cellule, et par là même de celle de l’organisme pluricellulaire. Les cellules de différents organes expriment ainsi des gènes distincts, qui leur permettent d’exercer leurs fonctions. Dans l’organisme en développement, l’expression de gènes spécifiques par les cellules leur permet ainsi de s’engager dans une voie de développement donnée, puis de se différencier.
Afin de comprendre comment et par quels gènes sont contrôlés les mécanismes de la différenciation cellulaire, les chercheurs ont développé une stratégie : regarder ce qui se passe lorsqu’un gène « fonctionne mal », est absent, etc. Ces études ont d’abord été réalisées grâce à l’étude de mutants, naturels ou provoqués par l’utilisation de produits chimiques ou ionisants. C’est ainsi que, par une stratégie de mutagenèse particulièrement importante, Nüsslein-Volhardt, Lewis et Wieshaus ont démontré l’implication des gènes du complexe Hox dans le développement embryonnaire précoce de la drosophile.
Néanmoins, ces techniques de mutagenèse aléatoire posent un gros problème : les chercheurs n’ont aucun contrôle sur les mutations qui vont avoir lieu… Ceci impose un fastidieux travail de criblage pour rechercher les mutants. De plus, les mutations obtenues ont souvent pour conséquence une diminution de l’expression du gène ou une diminution de l’efficacité de la protéine codée. Il est ainsi difficile de comprendre si l’effet observé est dû à l’absence de la protéine, à une expression incorrecte de la protéine, à la présence d’une protéine modifiée, etc.
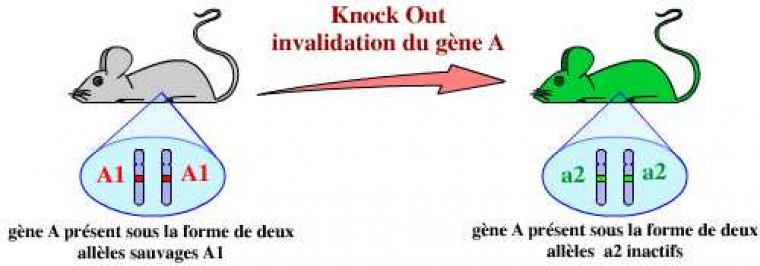
Une approche a priori bien plus intéressante est donc d’éliminer purement et simplement un gène. C’est ce que l’on appelle l’invalidation d’un gène, ou plus communément le KO (knock-out) d’un gène. Cette technique est basée sur la recombinaison homologue. Ceci permet, d’après les conséquences observées, de proposer un rôle pour le gène en question.
Quels sont les prérequis à l’invalidation d’un gène ?
L’invalidation de gènes n’a pu être possible que suite à l’avènement de la biologie moléculaire. La technique de KO nécessite en effet de disposer de nombreux « outils moléculaires », permettant la manipulation de fragments d’ADN, et donc de gènes.
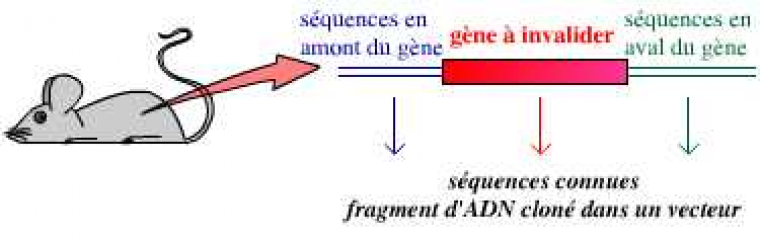
Il n’est pas possible de décider « je vais invalider le gène de tel ou tel autre caractère ». Pour invalider un gène, il est nécessaire de l’avoir identifié au préalable : le gène, sous forme d’un fragment d’ADN, doit avoir été cloné – c’est-à-dire qu’il doit avoir été isolé, conservé, sa séquence nucléotidique déterminée intégralement. Il est souvent aussi nécessaire de disposer de la séquence nucléotidique des fragments d’ADN bordant ce gène dans le génome de l’organisme étudié.
Il est nécessaire de disposer du gène à invalider et des séquences le bordant dans le génome, avant de pouvoir procéder à son invalidation.
Enfin et surtout, il faut avoir une bonne raison d’invalider le gène… Ce processus est long et coûteux, et n’est pas employé « à la légère » par les laboratoires de recherche. Il faut compter d’importants moyens techniques (animalerie, matériels), humains (chercheurs, techniciens), et en moyenne deux années de travail pour réaliser un KO chez la souris…
Comment fait-on pour invalider un gène ?
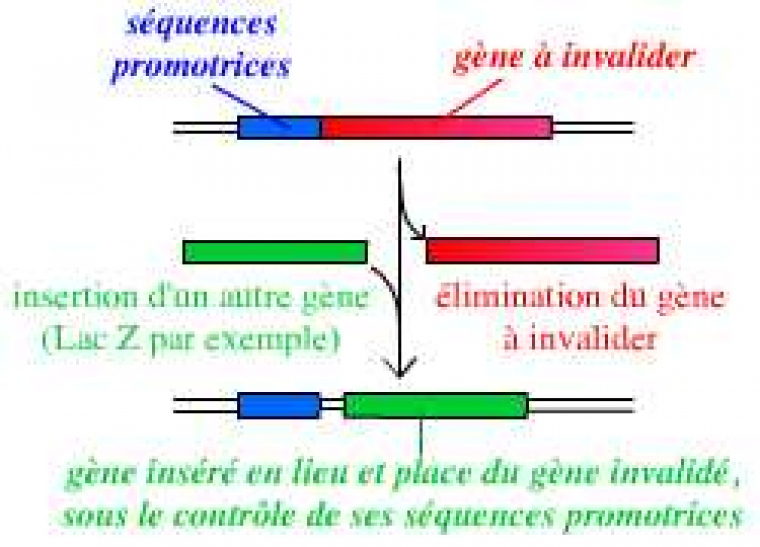
La technique d’invalidation d’un gène se base sur la recombinaison homologue : on peut ainsi remplacer le gène complet, présent dans le génome de l’organisme, par une version suffisamment incomplète du gène pour qu’il ne s’exprime pas, ou par un autre gène (ce qui permet de suivre l’expression du gène invalidé).
Pour invalider un gène, on peut le remplacer par un autre gène, par exemple un gène rapporteur tel que le gène lacZ (qui code la β-galactosidase).
La description suivante est faite chez la souris. Elle est valable pour tout organisme Mammifère.
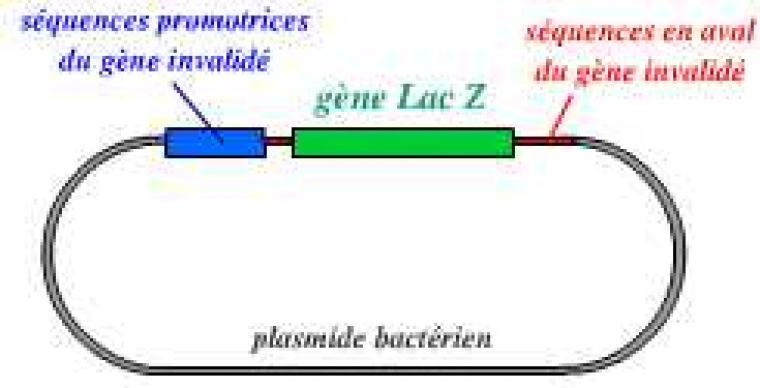
La première étape consiste donc en la création d’un vecteur, portant le gène invalidé :
Ce vecteur est un plasmide bactérien portant le gène lacZ (qui remplace le gène invalidé), avec, en amont et en aval, des séquences qui entourent le gène à invalider dans le génome de l’organisme.
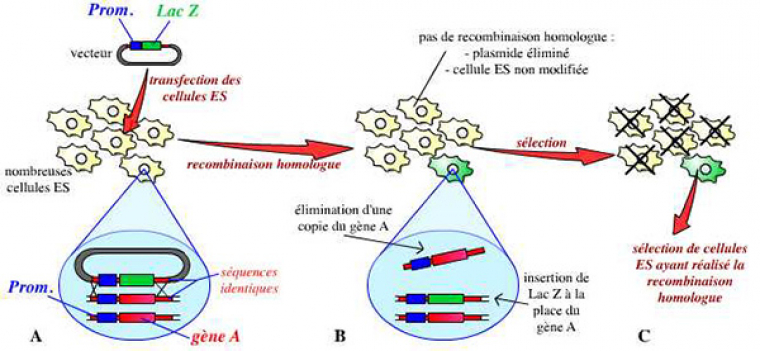
Une fois la construction réalisée, ce vecteur est introduit dans des cellules ES (embryonic stem cells : cellules souches embryonnaires) par électroporation. On utilise des cellules ES car ces cellules sont capables de donner toutes les cellules de l’organisme (elles sont totipotentes). Une étape longue et fastidieuse consiste alors à sélectionner un clone de cellules ES ayant effectivement reçu le vecteur, et ayant procédé à la recombinaison homologue. Une des deux copies du gène est ainsi invalidée dans leur génome :
A. Un vecteur a été réalisé : plasmide portant le gène lacZ (dans notre exemple), entouré des séquences bordant le gène A dans le génome. Ce vecteur est introduit dans des cellules ES.
B. Dans quelques cellules ES, un évènement de recombinaison homologue a lieu : grâce à une reconnaissance des séquences communes entre le vecteur et le génome de la cellule, une copie du gène A est remplacée par la construction portée par le vecteur (ici, le gène lacZ).
C. La majorité des cellules ES n’a en fait pas réalisé cette recombinaison homologue. Les quelques cellules ES qui l’ont réalisée sont sélectionnées, multipliées, et vont permettre la suite du protocole.
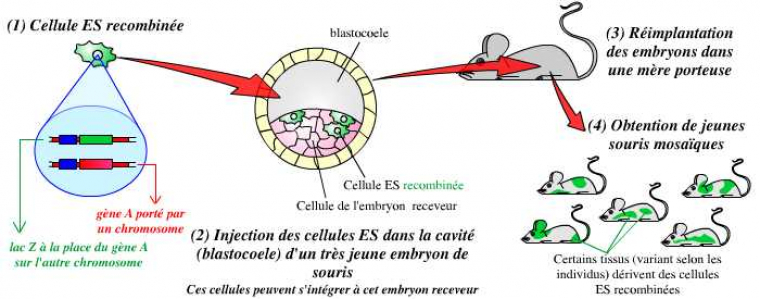
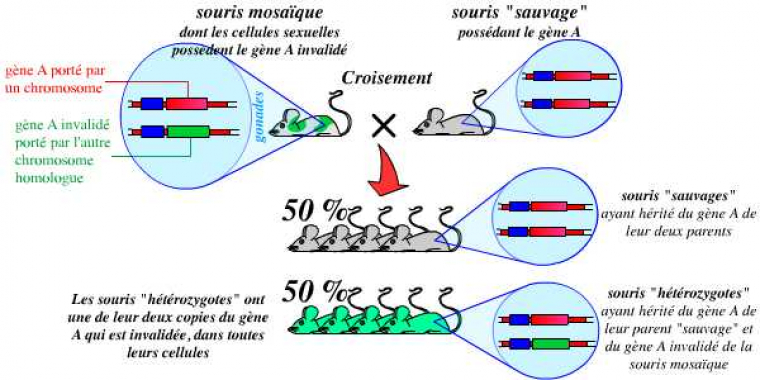
La deuxième étape consiste en l’obtention d’animaux génétiquement modifiés. Pour cela, on procède à l’injection de quelques cellules ES modifiées dans le blastocœle (cavité) de très jeunes embryons de souris. Ces cellules s’intègrent naturellement à la masse cellulaire interne (MCI), ensemble de cellules dont dérivent toutes les cellules du futur organisme. Les embryons ayant reçu ces cellules ES sont alors réimplantés dans des mères porteuses. On obtient ainsi, à la naissance, des souris « mosaïques » dont les tissus dérivent pour partie des cellules de la MCI « normales », et pour partie de cellules ES modifiées :
L’injection de cellules ES recombinées dans de très jeunes embryons de souris (ces cellules s’intègrent à l’embryon) permet l’obtention d’individus mosaïques, dérivant pour partie de cellules non modifiées, et pour partie de cellules recombinées.
Parmi ces individus mosaïques, il est alors nécessaire de sélectionner ceux dont les cellules de la lignée germinale dérivent de cellules ES : eux seuls seront capables de transmettre le gène invalidé à leur descendance.
À partir du moment où l’on dispose de tels individus mosaïques, la troisième étape consiste en un croisement entre ces souris et des souris « sauvages » (c’est-à-dire possédant l’allèle normal, non modifié). Ce croisement permet d’obtenir des souris hétérozygotes possédant un allèle sauvage et un allèle invalidé :
Le croisement d’une souris mosaïque (dont les cellules sexuelles dérivent des cellules ES recombinées) avec une souris sauvage (ne possédant pas cette mutation) permet d’obtenir, pour 50 % de la descendance, des souris hétérozygotes pour cette mutation.
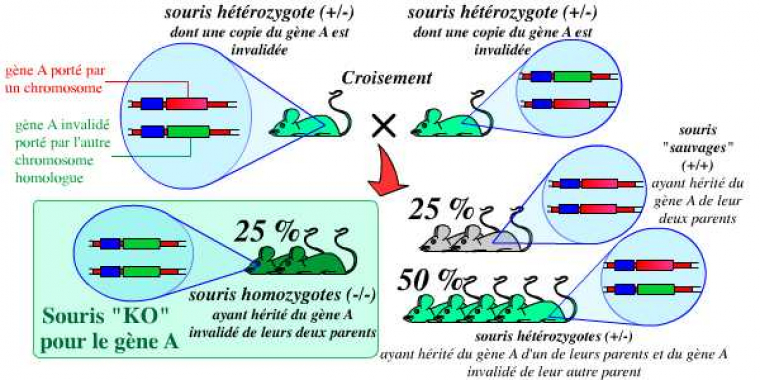
La dernière étape consiste alors à croiser ces souris hétérozygotes entre elles pour obtenir (dans une proportion de 25 %) des souris homozygotes, possédant deux copies du gène invalidé (et donc, logiquement, aucune copie du gène fonctionnel) :
Le croisement des hétérozygotes obtenus précédemment, entre eux, permet l’obtention (dans la proportion de 1/4 de la descendance) d’individus homozygotes : ce sont les individus KO, chez qui le gène a été invalidé.
Quels résultats peut-on obtenir suite à l’invalidation d’un gène ?
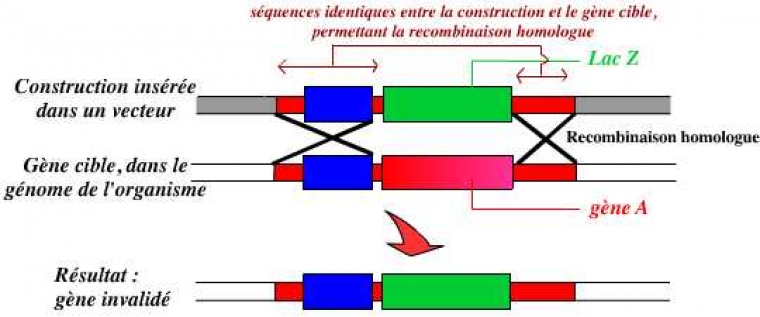
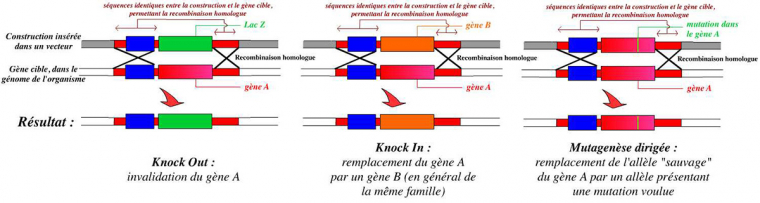
En résumé, l’invalidation d’un gène par KO consiste donc à remplacer dans le génome de l’organisme ce gène par une version modifiée ne permettant pas son expression :
L’invalidation d’un gène est le remplacement de ce gène, dans le génome de l’organisme, par une version modifiée, inactive, de ce gène.
Dans cet exemple, le gène A est remplacé par un autre gène : lacZ. Ce remplacement est permis par la présence de séquences identiques entre la construction et le génome de l’organisme, au niveau desquelles peut se réaliser une recombinaison homologue.
Intérêt des hétérozygotes
Chez les hétérozygotes, le développement n’est en général pas affecté, l’expression de la copie subsistante du gène s’effectuant normalement. Il est de plus souvent possible de suivre alors de manière précise l’expression du gène étudié.
En effet, une technique courante est de remplacer le gène ciblé par un autre gène, qui sera facilement détectable, tel que le gène codant la β-galactosidase (gène lacZ). Ceci permet d’observer l’expression précise du gène étudié, puisque le gène introduit (lacZ) se retrouve sous le contrôle des séquences promotrices du gène invalidé.
Exemple de HOXB8 chez la souris.
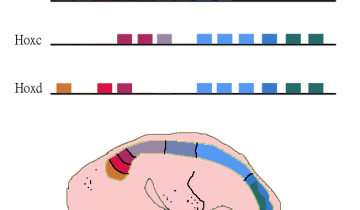
Les Mammifères possèdent 4 complexes de gènes Hox (A, B, C et D) comptant chacun plusieurs gènes (numérotés par un numéro entre 1 et 13). Ces gènes ont une importance particulière lors de la détermination des devenirs cellulaires le long de l’axe antéro-postérieur. Afin d’étudier le rôle du gène 8 du complexe B (HOXB8), des chercheurs ont procédé au remplacement d’une copie de ce gène par le gène lacZ.
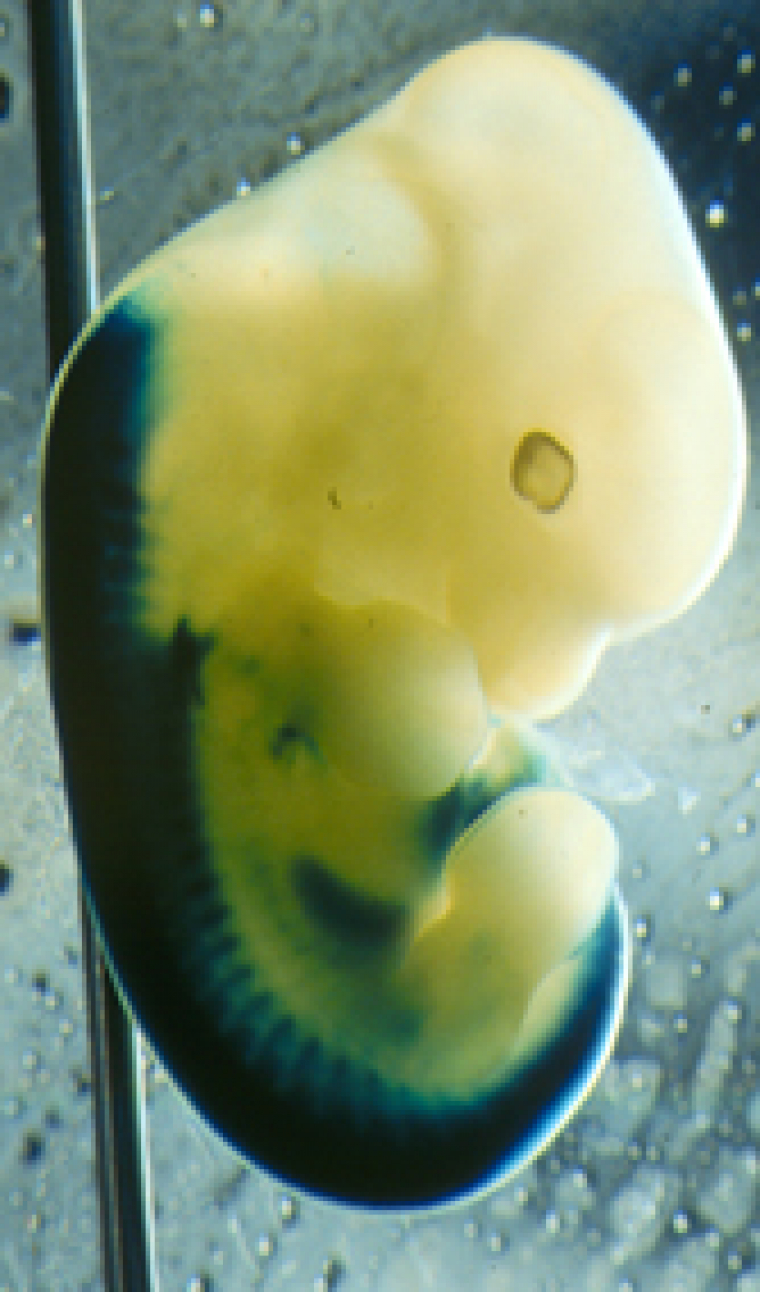
Les souris hétozygotes (+/-) ne présentent pas de phénotype particulier. Chez ces souris, le gène lacZ s’exprime selon le même patron d’expression que le gène HOXB8 puisqu’il est placé sous le contrôle des mêmes séquences promotrices. Or, il est facile de détecter la présence de la β-galactosidase, produit de l’expression du gène lacZ, par une réaction colorée : on peut alors connaître avec précision les lieux où s’exprime normalement le gène HOXB8 (voir fig. 11).
Expression du gène lacZ (en bleu foncé) en lieu et place de HOXB8 chez un jeune embryon de souris. On détecte une expression dans le tube neural et les somites, avec une frontière antérieure au niveau de la 7e pré-vertèbre de l’embryon.
Intérêt des homozygotes
Chez les individus homozygotes pour l’invalidation du gène, il est possible d’étudier les différentes conséquences de l’absence de ce gène sur l’organisme. Ceci peut se manifester par des phénotypes très marqués, facilement identifiables, tels que de graves altérations du développement embryonnaire, des malformations, une stérilité, des dérèglements physiologiques. Toutefois, dans de nombreux cas, les effets peuvent être plus délicats à déceler. Ceci peut avoir de nombreuses causes : il est possible que l’absence du produit du gène soit compensée par l’activation d’autres voies de régulation physiologique. Il est aussi possible que le gène soit redondant avec d’autres gènes.
C’est en particulier le cas des gènes homéotiques. Ces gènes ont une grande importance chez la drosophile. Leur présence et leur similitude d’expression chez les Mammifères suggèrent un rôle important de ces gènes lors du développement de ces organismes. Mais, il s’est avéré que de nombreux KO de gènes homéotiques chez la souris ne conduisent qu’à de très faibles effets phénotypiques. Comment expliquer ces faits apparemment contradictoires ?
Il faut se rappeler que les gènes homéotiques sont présents sous la forme de quatre complexes chez les Vertébrés. Ainsi, si on invalide un gène homéotique au hasard (appelons le HOXAx : gène Hox numéro x du complexe A) il existe souvent deux ou trois autres gènes homologues situés dans les autres complexes (par exemple HOXBx et HOXCx). Ces gènes peuvent très bien s’exprimer d’une manière très proche de celle de HOXAx. Ainsi, en l’absence de HOXAx, les deux autres gènes (HOXBx et HOXCx) peuvent « prendre le relais » et palier à l’absence de HOXAx qui ne se traduit alors que par de très faibles effets phénotypiques.
Pour ces raisons, une stratégie actuelle est de procéder à des KO multiples. Dans notre exemple, on procédera ainsi à l’inactivation simultanée des trois gènes HOXx (des complexes A, B et C). À la suite d’une telle stratégie, on peut alors (enfin…) souvent voir apparaître un effet phénotypique important qui permet alors de spéculer sur le rôle de ces gènes.
On le voit bien, étant donné la complexité des réseaux génétiques dans l’organisme il n’est pas toujours simple d’interpréter les phénotypes observés. L’invalidation d’un gène ne présente donc jamais un résultat final mais permet seulement de dégager les grandes directions qui devront être suivies pour la compréhension parfaite des rôles de ces gènes, ou de la réalisation de certains phénomènes dans l’organisme.
Exemple de l’invalidation du gène HOXB8.
En utilisant les hétérozygotes (+/-) obtenus en invalidant HOXB8 (voir exemple ci-dessus), on peut obtenir des homozygotes (-/-) : le gène HOXB8 est invalidé chez ces souris.
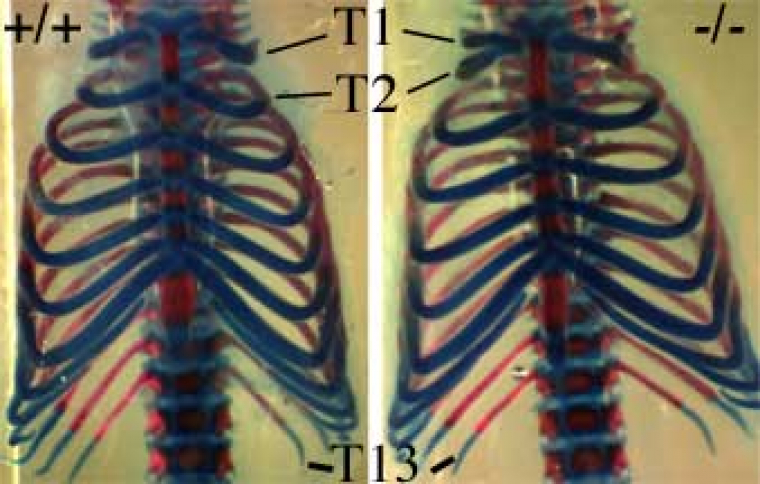
L’invalidation de HOXB8 conduit à plusieurs défauts de développement, et en particulier à une fusion partielle des deux premières côtes. Ceci correspond en fait à un changement d’identité des vertèbres (qui portent les côtes) : la deuxième vertèbre thoracique (T2) « prend l’identité » de la première vertèbre thoracique (T1). C’est ce changement qui conduit à cette fusion partielle de côtes.
Cette modification est présentée sur les figures 12 et 13.
En plus de ce changement d’identité T2 vers T1, on détecte aussi souvent une modification de la première vertèbre thoracique (T1) vers une identité de 7e vertèbre cervicale (C7) adjacente.
Ces modifications sont décelables chez environ 1/4 des homozygotes. Ce relativement faible pourcentage montre bien toute la limite d’une telle stratégie d’invalidation de gène : ici, près de 3/4 des souris ne possédant plus HOXB8 ne présentent pas ces phénotypes !
Vue de face des cages thoraciques d’une jeune souris sauvage (+/+) et d’une souris KO pour HOXB8 (-/-).
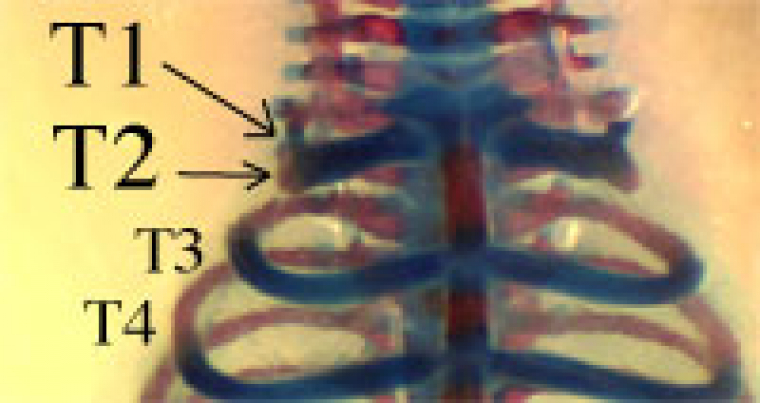
Vue de côté (détail).
En plus de la modification observable au niveau des cotes T1 et T2, noter, chez la souris KO, l’absence d’une excroissance typique de la vertèbre T2 (petit cadre à gauche).
Exemple d’invalidations multiples : ensemble des paralogues HOX8 (HOXB8, HOXC8 et HOXD8)
Au sein des quatre complexes Hox des Mammifères, trois présentent un gène HOX8 : HOXB8, HOXC8 et HOXD8. Ces trois gènes (dits gènes paralogues) dérivent d’un même gène HOX8 ancestral, et sont ainsi de séquences très proches. Leur expression chez les Mammifères est aussi très similaire : tous trois s’expriment le long de l’axe antéro-postérieur au cours du développement embryonnaire. HOXB8 (voir fig. 12) et HOXC8 s’expriment de manière très proche ; HOXD8 s’exprime dans les mêmes tissus, mais son expression la plus intense est légèrement décalée vers l’arrière de l’animal.
L’invalidation de chacun de ces gènes a été réalisée, avec des résultats divers (conventions de notation : C = vertèbre cervicale ; T = vertèbre thoracique ; L = vertèbre lombaire. Chaque vertèbre est suivie de son numéro, la 1 étant la plus antérieure) :
-
HOXB8 -/- : changement d’identité T2 vers T1 et T1 vers C7 (voir fig. 12) chez environ 1/4 des individus -/-
-
HOXC8 -/- : changement d’identité T7 vers T6, T8 vers T7 et L1 vers T13 (voir fig. 13) chez environ 50 à 90 % des individus -/-
-
HOXD8 -/- : très faible phénotype, proche de celui observé pour HOXC8 (voir fig. 14).
Ces trois invalidations montrent un rôle des gènes HOX8 dans la détermination des devenirs le long de l’axe antéropostérieur, plus précisément dans la région thoracique. Leur invalidation conduit à une « antériorisation » : les vertèbres présentent un phénotype décalé vers l’avant, par rapport à une souris sauvage. Il est cependant à noter que ces phénotypes ne touchent en général que entre 5 et 7 individus sur 10… Or, on aurait pu penser que, ces gènes étant importants, tous les individus mutés auraient présenté le phénotype…
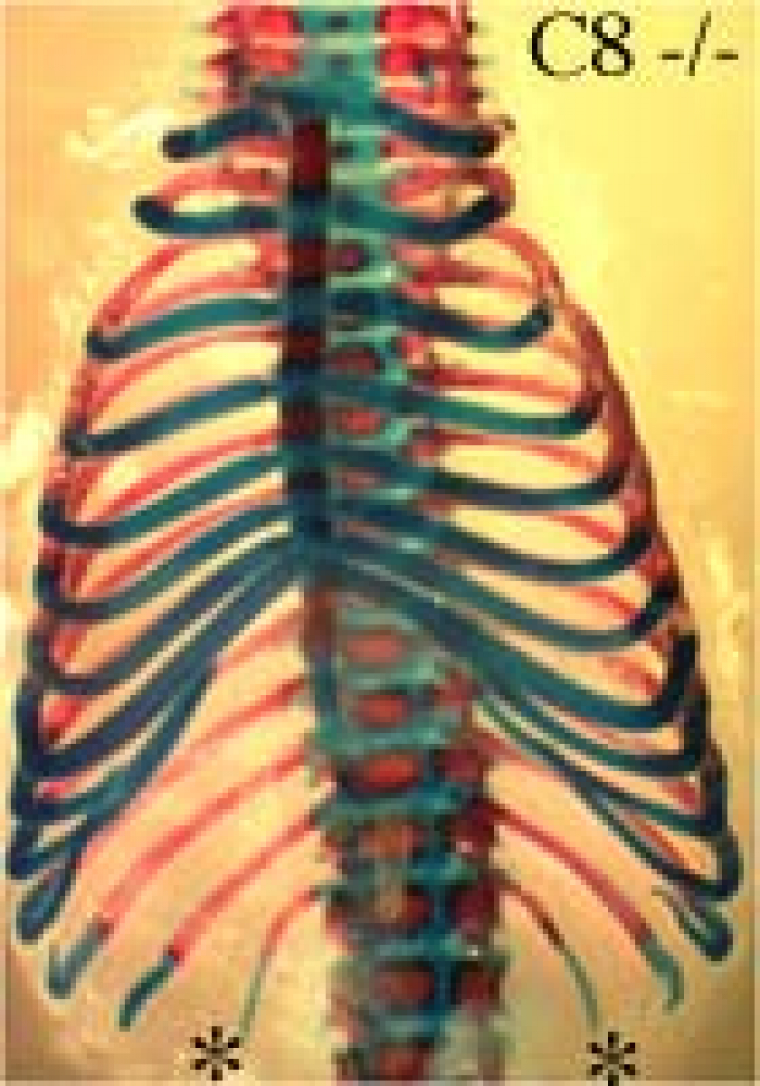
Chez les souris HOXC8 -/- , dans près de 9 cas sur 10 on observe un changement d’identité de la première vertèbre lombaire (L1) vers une identité de vertèbre thoracique (T13).
Ceci se manifeste par une paire de côtes flottantes supplémentaires (« côtes 14 ») notées par une étoile * sur cette photo.
Il a donc été réalisé des doubles invalidations, puis des triples invalidations de ces gènes, conduisant à des individus HOXB8 -/-, HOXC8 -/-, HOXD8 -/- (« triple KO »). Chez tous ces individus, on retrouve les phénotypes caractéristiques des invalidations de HOXC8 et de HOXD8. Le plus intéressant se situe au niveau des phénotypes caractéristiques de HOXB8. En effet, on trouve les proportions suivantes de phénotypes :
| Pourcentage de souris présentant le phénotype parmi celles ayant eu les gènes suivant invalidés | T2 vers T1 | T1 vers C7 |
|---|---|---|
| HOXB8 -/- | 27 | 23 |
| HOXB8 -/-, HOXC8 -/- | 80 | 70 |
| HOXB8 -/-, HOXD8 -/- | 79 | 74 |
| HOXB8 -/-, HOXC8 -/-, HOXD8 -/- | 100 | 91 |
Un exemple de souris « triple KO » obtenue par invalidations des trois gènes HOX8 (B, C, D) : zoom sur le haut du thorax montrant la fusion partielle des deux premières côtes.
Ce phénotype est grandement amplifié chez les triples KO par rapport aux souris chez lesquelles le seul gène HOXB8 a été invalidé.
On peut donc remarquer que le phénotype, présent chez seulement 1/4 des individus en l’absence de HOXB8, est présent chez 4/5 des individus si un deuxième gène HOX8 est invalidé, et chez la quasi-totalité des souris en cas d’invalidation des trois gènes HOX8. Ce résultat montre que, bien que HOXB8 joue un rôle important dans la détermination de l’identité des vertèbres T1 et T2, les autres gènes HOX8 (C8 et D8) peuvent « compenser » son absence, diminuant ainsi le phénotype observé. C’est seulement en leur absence totale que le phénotype apparaît clairement avec une pénétrance quasi-totale (90 à 100 % de souris présentant le phénotype).
Cet exemple illustre bien toute la difficulté des invalidations de gènes : il existe chez les Mammifères de nombreux gènes paralogues appartenant à des familles multigéniques. Lorsque l’on invalide un gène on ne peut ainsi jamais être sûr d’observer le phénotype lié au rôle de ce gène dans l’organisme. Il est souvent possible que d’autres gènes aient « pris le relais », masquant ainsi plus ou moins le phénotype.
De nombreuses expériences d’invalidation de gène se sont ainsi soldées par une absence quasi-totale de phénotype. Ce qui ne veut pas dire que le gène en question ne sert à rien ! La recherche s’oriente donc quand c’est possible (et que cela semble pertinent) vers des stratégies d’invalidations multiples, bien plus complexes, mais fournissant des résultats bien plus « satisfaisants ».
Des techniques variantes : le knock-in, la mutagenèse ciblée
La technique de recombinaison homologue peut permettre de réaliser des expériences plus « fines » que la simple invalidation d’un gène.
En premier lieu, il est possible de remplacer par cette technique un gène par un autre : c’est ce qui est réalisé, par exemple, lors de l’invalidation d’un gène en le remplaçant par un gène « rapporteur » tel que lacZ. Dans ce cas-là, on a remplacé le gène invalidé par un gène étranger (bactérien dans le cas du gène lacZ). Mais il est possible de le remplacer par un autre gène tiré du génome de l’organisme (voir fig. 16).
Par exemple, l’invalidation du gène Engrailed 1 chez la souris conduit à des défauts de développement se manifestant au niveau du cervelet. On peut relier ces défauts à l’expression d’Engrailed 1 au niveau de la future région du cervelet lors du développement embryonnaire. Un autre gène de la même famille multigénique, Engrailed 2, s’exprime sensiblement au même endroit un peu plus tard. Engrailed 1 et Engrailed 2 ont de plus des séquences très proches. Une expérience a donc consisté à remplacer le gène Engrailed 1 dans le génome de la souris par le gène Engrailed 2. Cette expérience est nommée un « knock-in d’Engrailed 2 dans Engrailed 1 ». Dans ce cas on s’aperçoit que, bien que le gène Engrailed 1 ait été invalidé, aucun phénotype n’est visible : ceci montre que les deux protéines issues des gènes Engrailed 1 et Engrailed 2 ont une action potentiellement équivalente. La différence d’action entre ces deux gènes se réduit donc en fait à une différence dans le contrôle (spatial et temporel) de l’expression de ces gènes.
Un knock-in est donc en fait un cas particulier de knock-out. Les expériences de knock-in permettent d’observer ce qui se passe si l’on remplace un gène par un autre gène qui lui est proche. On place ainsi ce deuxième gène sous le contrôle de l’ensemble des séquences régulant l’expression du gène invalidé.
La recombinaison homologue permet de remplacer un gène par un autre. Cependant, il est aussi possible de procéder ainsi à une mutation ponctuelle : on peut très bien remplacer un gène A par une version très légèrement différente de ce gène ne comportant une différence qu’au niveau de quelques nucléotides. On procède alors de la même façon que précédemment, en plaçant juste une version modifiée du gène A (voir fig. 16) dans la construction.
Différents résultats sont obtenus par la recombinaison homologue en fonction de la construction réalisée au départ : knock-out (invalidation du gène), knock-in (remplacement par un autre gène), ou mutagenèse dirigée (remplacement par un allèle muté).
Remerciements
Un grand merci au Dr. Jacqueline Deschamps (Hubrecht Laboratory, The Nederlands Insitute for Developmental Biology, Utrecht, Pays-Bas) pour les photographies de KO des gènes HOX8.