La transcription est le processus de copie du matériel génétique (ADN ou ARN) en ARN.
Chez les procaryotes une seule ARN-polymérase effectue la transcription pour tous les types d'ARN, tandis que chez les eucaryotes trois ARN-polymérases différentes interviennent selon qu'il s'agit de produire un ARN ribosomique, un ARN messager ou un petit ARN (ARN de transfert par exemple).
Cet article présente, chez les eucaryotes, les évènements impliqués dans la transcription des gènes codant les chaînes polypeptidiques, c'est à dire le processus de production des ARN messagers.
Introduction
La transcription est le processus de copie du matériel génétique (ADN ou ARN) en ARN.
Chez les procaryotes une seule ARN polymérase-ADN dépendante effectue la transcription pour tous les types d’ARN, tandis que chez les eucaryotes, trois ARN polymérases (ARNpol) interviennent : l’ARNpol I ou A pour les ARN ribosomiques transcrits dans le nucléole (28S, 18S et 5,8S), l’ARNpol II ou B pour les ARNm, et l’ARNpol III ou C pour les petits ARN (ARNt, ARNr 5S, ARNsn). Pour certains virus à ARN, enfin, l’ARN est transcrit par une ARNpol-ARN dépendante appelée aussi réplicase.
Intéressons-nous plus particulièrement à la transcription des gènes codant pour les chaînes polypeptidiques chez les eucaryotes. Leur transcription se déroule en deux étapes : formation d’un ARN prémessager puis maturation de cet ARN prémessager pour former un ou plusieurs ARN messagers.
La formation d’un ARN prémessager
L’initiation de la transcription
Tout l’ADN n’est pas transcrit, seules les régions correspondant à des gènes le sont. Et encore, cette expression peut être régulée selon le stade de développement, le type cellulaire, l’environnement, etc. Dès lors, un acteur doit intervenir pour déterminer à quel endroit une région d’ADN doit commencer à être transcrite : c’est le rôle du promoteur.
Le promoteur
Le promoteur correspond à une région non transcrite de l’ADN, généralement juste en amont du début de la région transcrite, dont la séquence permet le recrutement de l’ARNpol II. Certaines séquences du promoteur (surnommées « boîte ») ont une importance particulière dans ce processus, essentiellement parce que ces séquences sont reconnues spécifiquement par différentes protéines appartenant au complexe d’initiation (voir paragraphe suivant) :
-
la « boite TATA » riche en thymine et adénine, la plus importante, est située vers -25 à -30 nucléotides du site de démarrage de la transcription (noté +1) ;
-
des éléments proximaux :
-
la « boîte CAAT » (facultative), contenant de la cytosine, est située vers -120 à -80 nucléotides du site de démarrage de la transcription.
-
la « boîte GC » (facultative également), riche en guanine et cytosine, peut être présente entre la boîte CAAT et la boîte TATA.
-
Signalons que si ces séquences sont souvent bien conservées, une variabilité non négligeable peut également être observée. Ainsi, il existe des promoteurs sans « boîte TATA » [référence 1].
Le complexe d’initiation
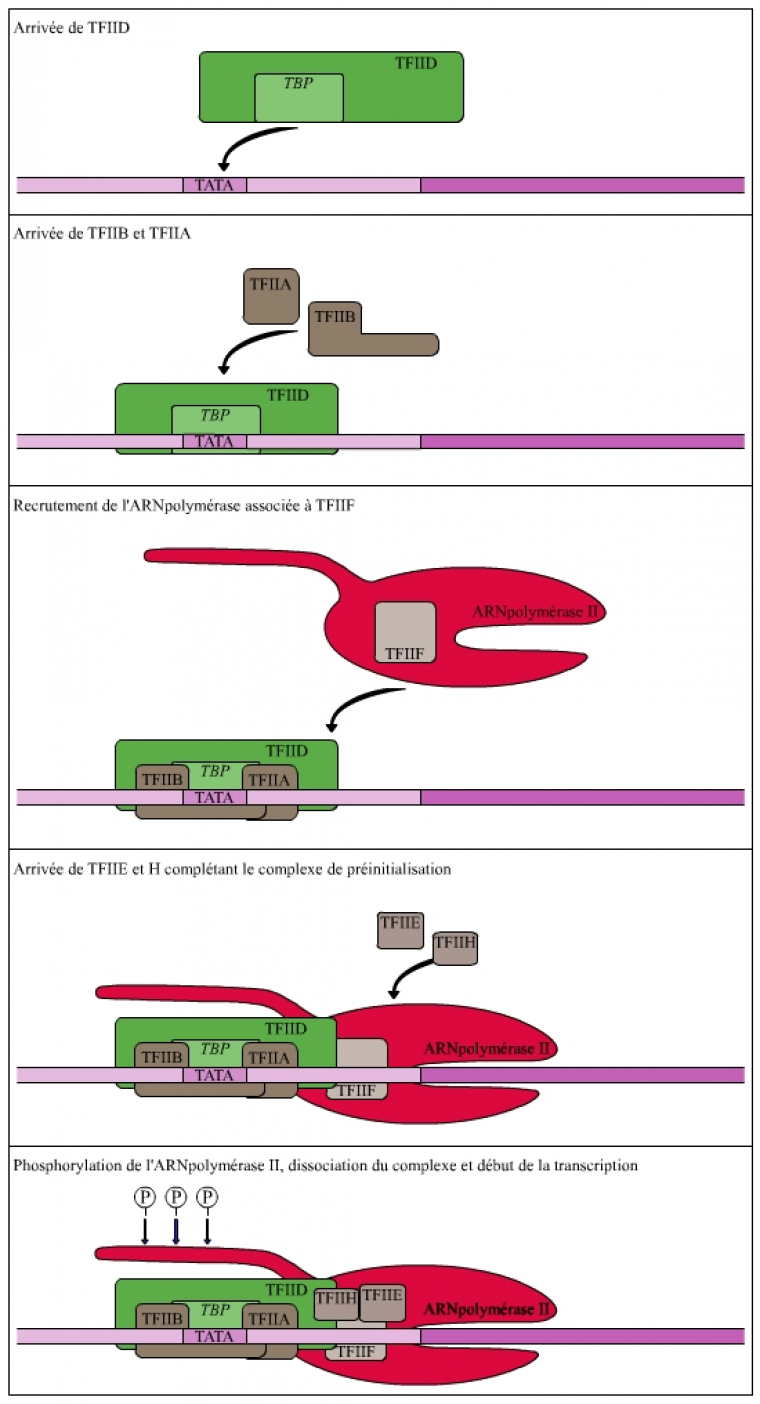
Contrairement à ce qui se passe chez les procaryotes, l’ARNpol II des eucaryotes ne reconnaît pas seule le promoteur proximal. Elle effectue ce travail en compagnie de nombreux co-facteurs protéiques qui se recrutent les uns les autres et qui forment avec elle un complexe d’initiation. Ces facteurs sont notés TFIIA, TFIIB, etc. pour Transcription Factor for RNA polymerase II. Ils correspondent aux facteurs généraux de la transcription, car ils s’assemblent sur tous les promoteurs utilisés par l’ARNpol II. La séquence d’assemblage du complexe d’initiation est décrite sur la figure 1 [référence 2].
La TBP est la première protéine qui reconnaît une séquence spécifique de l'ADN initiatrice de la transcription (la boite TATA). Le facteur TFIIB semble impliqué dans la sélection précise du site d'initiation (nucléotide à partir duquel se déroule la transcription). Le facteur TFIIH comporte plusieurs activités enzymatiques dont une activité hélicase permettant l'ouverture de la double hélice d'ADN au niveau du promoteur, et une activité kinase responsable de la phosphorylation de la queue C-terminale de l'ARN polymérase II. Cette phosphorylation provoque une modification de la structure tridimensionnelle de l'ARN polymérase qui entraîne la dissociation du complexe d'initiation et le début de la transcription.
TFII = Transcription Factor for RNApolymerase II (facteurs généraux de la transcription pour l'ARN polymérase II).
TBP = TATA box-Binding Protein (protéine de liaison à la boite TATA).
La liaison du complexe de transcription au promoteur proximal provoque l’ouverture et le déroulement des deux brins de son ADN, tout en indiquant le brin qui va être transcrit.
Intervention de facteurs spécifiques de la transcription
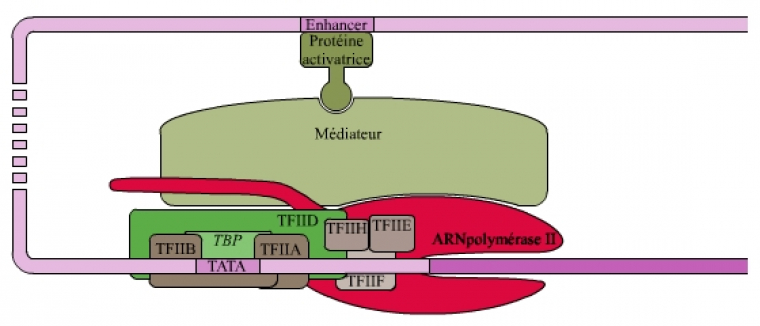
Le complexe d’initiation composé de l’ARNpol II et des différents TFII est suffisant pour obtenir une activité transcriptionnelle in vitro mais à très faible taux. L’augmentation de cette activité basale (ou sa répression) est sous la dépendance de facteurs spécifiques qui vont interagir avec le complexe d’initiation. Ces protéines activatrices ou inhibitrices (éléments trans-régulateurs) se lient à des promoteurs distaux spécifiques (séquences cis-régulatrices) de l’ADN, appelées amplificateurs (enhancers) lorsqu’ils recrutent des cofacteurs activateurs, ou silenceurs (silencers) lorsqu’ils recrutent des cofacteurs inhibiteurs. Ces promoteurs distaux peuvent être situés à des milliers de nucléotides du promoteur proximal. Malgré la distance qui sépare les promoteurs proximaux des promoteurs distaux, ces derniers agissent sur le promoteur proximal par le jeu de courbures de l’ADN, des facteurs de transcription et du médiateur qui maintient liés tous ces acteurs (voir fig. 2).
Les amplificateurs (enhancers) sont des séquences situées parfois à plusieurs milliers de nucléotides d'un promoteur et qui, par un jeu d'interactions protéiques, stabilisent le complexe d'initiation, favorisant ainsi la transcription. Ces interactions sont rendues possibles par la courbure de l'ADN qui rapproche des éléments situés à de grandes distances les uns de autres. Cette activation est spécifique du gène et utilise une multitude de facteurs de transcription spécifiques (les protéines activatrices) qui agissent généralement sous forme dimérique.
Une répression spécifique peut agir selon le même type de modalité.
L’élongation
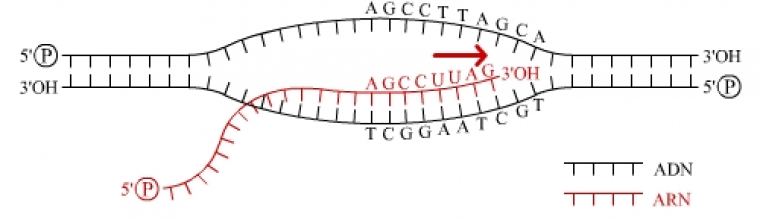
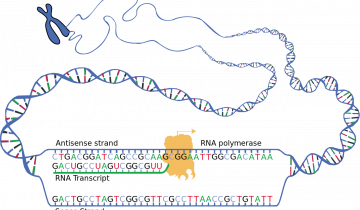
L’ARNpol II est équipée de facteurs protéiques d’élongation qui facilitent sa progression au travers d’une chromatine dont ils relâchent la structure (c’est l’un d’entre eux qui est mis hors d’état d’agir par le poison de l’amanite phalloïde…). Un ARN pré-messager complémentaire du brin matrice de l’ADN (brin antisens), donc identique au brin codant de l’ADN (brin sens), aux riboses et uraciles près, commence à être synthétisé selon la direction 5'-3' (voir fig. 3).
L'ARN polymérase lit le brin patron (ou brin antisens), qui est complémentaire du brin codant (ou brin sens), depuis son extrémité 3' vers son extrémité 5'. Le brin d'ARN néosynthétisé est donc identique au brin codant d'ADN, aux uraciles et riboses près.
La terminaison
L’ARNpol II est également équipée de facteurs protéiques de terminaison. Elle reconnaît ainsi un ou plusieurs signaux de terminaison portés par le brin progressivement parcouru et qui annoncent la fin de la transcription sur le brin d’ADN matrice (TTATTT par exemple, parfois aussi plus en aval ATACAAC…). Elle arrête bientôt son travail de transcription et libère l’ARNpm qu’elle vient d’assembler.
La formation d’un ou plusieurs ARNmessager(s)
Le transcrit primaire n’est pas utilisé tel quel pour la synthèse protéique (la traduction). Il doit subir des modifications qui répondent à plusieurs impératifs (augmentation de la demi-vie, modification de la séquence). Toutes ces modifications sont réalisées au fur et à mesure de la progression de la synthèse du préARNm dans le nucléoplasme. Il existe trois grands types de modification, catalysées chacune par des enzymes de nature protéique ou ribonucléique.
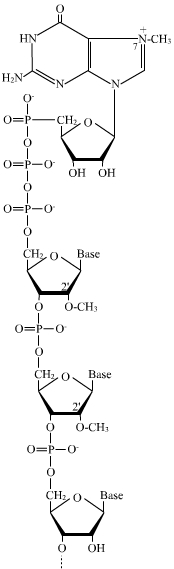
L’addition d’une coiffe en 5'
Elle a lieu dès le début de la transcription avant que la chaîne ne compte plus de 30 nucléotides. Elle consiste en l’ajout d’un nucléotide à guanine sur l’extrémité 5' de l’ARN suivi de sa méthylation sur l’azote 7 de la base, ainsi que de la méthylation en 2' du ribose du premier ou des deux premiers nucléotides du transcrit primaire. La particularité de cet ajout consiste dans le type de liaison mis en jeu : au lieu d'être reliés par une liaison ester-phosphorique entre le groupement OH porté par le carbone 3' du ribose du nucléotide à guanine et l'acide phosphorique alpha du premier nucléotide de l'ARN natif, les deux nucléotides sont reliés par une liaison anhydride d'acide entre les acides phosphoriques des deux nucléotides.
Il résulte de ces modifications que l’extrémité 5' de l’ARNm n’est pas porteuse des trois acides phosphoriques libres habituels, mais d’un GMP, ce qui limite la réactivité de cette extrémité et sa reconnaissance par les exonucléases (protection contre la dégradation). Cet ensemble sert de coiffe protectrice à l’extrémité 5' de l’ARNm. Elle est également nécessaire à l’exportation de l’ARNm vers le cytoplasme et à la liaison de ce dernier avec la petite sous-unité du ribosome, lors de l’étape d’initiation de la traduction.
L’excision-épissage
Chez les eucaryotes, les archéobactéries et les cyanobactéries, les gènes sont morcelés : constitués d’une alternance d’exons (parties codantes du gène) et d’introns (parties non codantes, bornées par des séquences de bases spécifiques : 5'GU et 3'AG), ils sont d’abord intégralement recopiés dans l’ARNpm, puis subissent une opération d’excision des introns (ainsi que celle parfois de petits morceaux d’exons) suivie d’un épissage (splicing), c’est-à-dire la réunion bout à bout des exons restants qui constituent l’ARNm. Ce remaniement se déroule au fur et à mesure de la progression de la transcription.
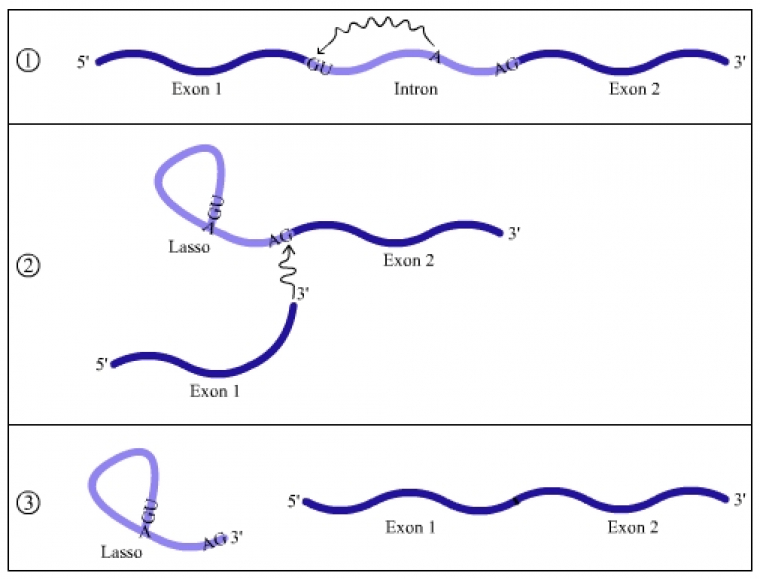
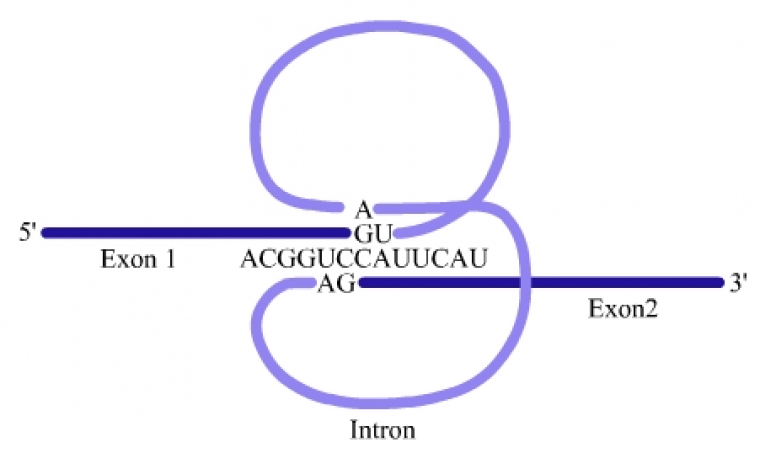
L’excision des introns s’opère par l’entremise d’une formation dite « en lasso » (voir fig. 5 et 6).
L'excision-épissage est réalisée par réaction d'un nucléotide à adénine (A) situé dans l'intron avec un nucléotide à guanine situé en 5' de l'intron. Cela entraîne la séparation de l'intron d'avec l'exon 1 (situé en amont) et la formation d'une structure en lasso interne à l'intron. Ensuite, l'extrémité 3' de l'exon 1 réagit avec l'extrémité 5' de l'exon 2 permettant l'épissage des deux exons et la libération du lasso qui sera dégradé par des ribonucléases.
Le splicéosome fait appel à des facteurs spécifiques : ribonucléoprotéines contenant des petits ARN (snRNP), ribozymes (ARN ayant des propriétés catalytiques), des enzymes spécifiques (maturases)... La structure secondaire du transcrit met en contact trois séquences de l'intron : la séquence du début de l'intron (extrémité 5'-GU ou site donneur), la séquence de la fin de l'intron (extrémité 3'-AG ou site accepteur) et un nucléotide à adénine (A du branchement) situé 40 nucléotides avant le site receveur.
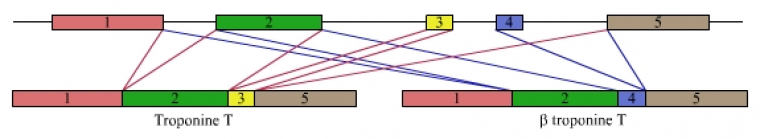
Mais l’épissage n’est pas seulement constitutif : il existe également des épissages alternatifs, dans lesquels l’élimination des introns (ou des portions d’exons) peut faire se lier entre eux des exons différents. C’est ce mécanisme qui démultiplie les capacités codantes d’un gène (voir un exemple d’épissage alternatif dans la fig. 7).
À partir du même préARNm peuvent être obtenus deux ARNm matures différents codant pour deux isoformes de la troponine, une molécule impliquée dans la structure des myofibrilles (voir l'article La contraction musculaire).
L’addition d’une queue polyA en 3'
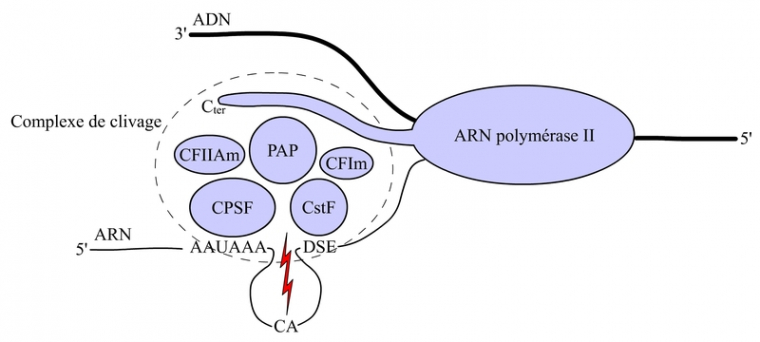
Le site de polyadénylation est codé au niveau du gène. Le site de clivage déterminé par le dinucléotide CA est entouré par une séquence AAUAAA très conservée, située 10 à 30 nucléotides en amont du site de clivage, et par une séquence DSE (DownStream Element) riche en U ou en GU, situé une trentaine de nucléotides en aval du site de clivage. La séquence AAUAAA est reconnue spécifiquement par un complexe protéique appelé CPSF (Cleavage and Polyadenylation Specific Factor), et la séquence DSE par un complexe CstF (Cleavage Stimulation Factor). Ces deux complexes et d’autres composants, comme l’ARN polymérase II et la poly(A) polymérase (PAP), vont interagit en formant le complexe de clivage qui va cliver la molécule de préARN au niveau du site de clivage (voir fig. 8).
CPSF = Cleavage and Polyadenylation Specific Factor
CstF = Cleavage Stimulation Factor
PAP = Poly(A) Polymerase
CF = Cleavage Factor
DSE = Downstream Element
La PAP va alors ajouter environ 200 nucléotides, une poly(A) binding protein II (PABP2) qui interagit avec la PAP se chargeant d’activer l’enzyme et de contrôler la longueur de cette queue poly(A). Il est intéressant de noter que cette polymérase n’utilise pas de matrice ADN pour créer cette séquence poly(A). La queue Poly(A) n’est donc pas codée par le génome. Notons que ce n’est pas un exemple isolé, d’autres mécanismes comme l’éditing ou la modification enzymatique de certaines bases (cytosine en uracile par exemple) étant encore beaucoup plus intrigants. Cette queue poly(A) confère de la stabilité au futur ARNm et se perd au fur et à mesure qu’il est traduit.
Conclusion
La formation de l’ARNpm puis de l’ARNm a été élucidée dans ses grandes lignes au prix d’un formidable travail dont Joseph Goldstein a rendu compte de façon très amusée, en 2003, lors de l’attribution du prix Lasker à l’un de ses contributeurs, Robert Roeder [référence 4].
Son mécanisme dépend à la fois d’une foule de protéines et de ribonucléoprotéines, elles-mêmes codées par une foule de gènes, et des séquences d’ADN avec lesquelles elles interagissent. On devine, à cette présentation générale résumée, à quel point la moindre mutation se produisant à l’un ou l’autre de ces niveaux peut avoir des conséquences majeures sur la transcription.
Bibliographie
Références
- Housset C. et Raisonnier A. Cours (2006-2007) de biologie moléculaire du CHU Pitié Salpêtrière.
- Sabine Caussanel. (2003) Contribution à l’étude du rôle de CKIP-1 dans la différenciation musculaire régulée par PI3-K – Identification des ARN messagers correspondant aux formes de la protéine CKIP-1. Mémoire de l’École Pratique des Hautes Études.
- Gottlieb S. (2003) The splice of life. Nature Horizon.
- Discours de Joseph Goldstein présentant le Prix Lasker de Robert Roeder. (2003) Fondation Lasker.
Quelques livres généraux
- Alberts B. (2004). "Biologie moléculaire de la cellule", 4e édition. Flammarion. Médecine sciences, ISBN 2257161211.
- Campbell N.A. et Rice J.B. (2004). " Biologie ", 2e édition. De Boeck Université