L’émergence désigne l’apparition d’un nouveau phénomène dans un système. En virologie, il s’agit de l’apparition ou réapparition d’un virus au sein d’un hôte donné, créant alors une nouvelle interface hôte-pathogène. La plupart des virus émergents chez l’Homme sont zoonotiques et circulent d’abord naturellement dans un réservoir animal avant de l’infecter. Les interactions hommes-animaux et le passage de la barrière d’espèce sont alors des facteurs essentiels modulant l’émergence virale. De nouveaux virus peuvent également émerger ou ré-émerger à partir d’autres virus humains, ou de virus que l’on pensait avoir éradiqué. La problématique de l’émergence s’inscrit dans la prévention et le contrôle des pandémies, comme en témoignent les récentes émergences de virus influenza (responsables de la grippe), de coronavirus (causant le SRAS, syndrome respiratoire aigu sévère) ou d’Ebola ces dernières années. La compréhension de l’émergence de virus humains repose sur l’étude des différents types de réservoirs viraux et de leurs contacts avec l’Homme, des mécanismes de variabilité des génomes viraux ainsi que de nombreux facteurs écologiques, économiques et sociaux. Dans cette revue, nous présentons les principaux mécanismes impliqués dans l’émergence virale et mettons ainsi en évidence l’aspect multifactoriel des causes conduisant à la dissémination d’un nouveau virus dans la population humaine.
Introduction
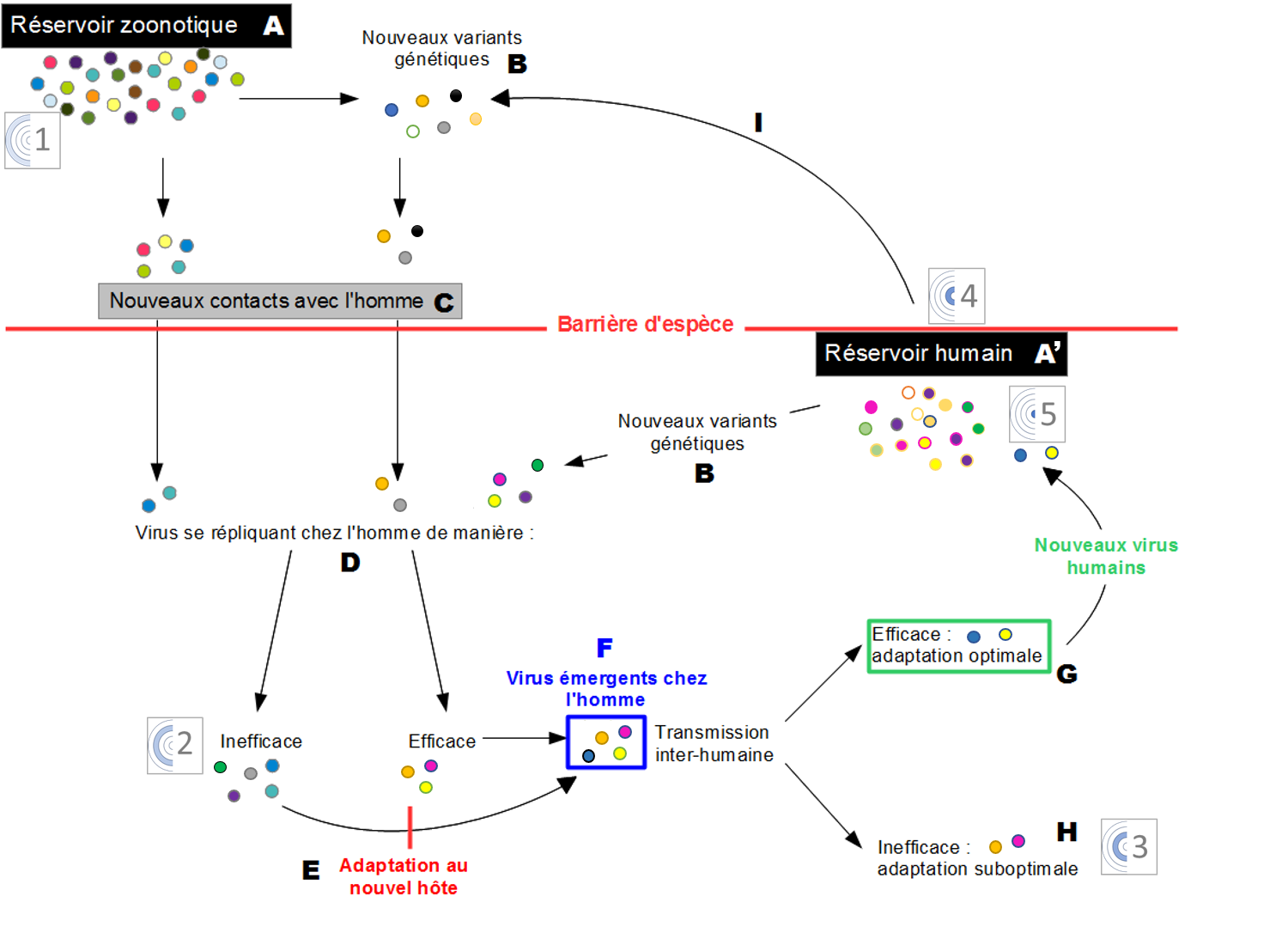
Les virus sont responsables de 25 % des maladies infectieuses émergentes humaines (Jones et al., 2008). Les exemples les plus connus sont l’épidémie de grippe espagnole de 1918 et du VIH (virus de l’immunodéficience humaine) depuis les années 80. L’émergence correspond à l’apparition d’un nouveau pathogène dans un hôte donné (Domingo, 2010), qui peut dans certains cas conduire à une épidémie, voire à une pandémie. Une ré-émergence correspond à la réapparition d’un pathogène après une période d’absence. Pike et al., (2010) ont défini 5 stades dans la transformation d’un virus animal en un virus humain, présentés en Figure 1. Le passage d’un stade à l’autre n’est pas inéluctable et requiert un certain nombre d’adaptations permises par la plasticité des génomes viraux (Domingo, 2010).
Figure adaptée de (Pike et al. 2010)
Réservoirs viraux
Lors de l’émergence d’un nouveau virus chez l’Homme, l’hôte d’origine du virus, qu’il soit le réservoir principal dans la nature, ou bien une espèce dite intermédiaire, est dans la grande majorité des cas de nature non humaine, ce sont les virus dits zoonotiques (Figure 2A). Ils peuvent être transmis directement à l’Homme, comme les virus foamy, ou spumavirus, d’origine simienne (SFV), ou faire intervenir plusieurs espèces hôtes qui sont alors qualifiés d’espèces intermédiaires. C’est le cas de nombreux virus tel que le virus Ebola dont le réservoir naturel semble être la chauve-souris (Leroy et al., 2005), et pour lequel la transmission à l’Homme s’effectue via l’infection de primates non humains (Morse et al., 2012). La mise en évidence du réservoir naturel d’un nouveau virus émergent est une étape importante dans la compréhension du virus. Des études sur l’origine du MERS-CoV (Middle East respiratory syndrome – Coronavirus), ont permis d’obtenir, grâce à des méthodes de séquençage à haut-débit, la séquence du génome viral entier dans un laps de temps très restreint, de dater l’apparition du virus et de le comparer à d’autres souches virales zoonotiques. La recherche du réservoir naturel du virus a alors pu être orientée vers des espèces de chauve-souris puis vers l’existence potentielle d’une espèce intermédiaire, comme le dromadaire (Cotten et al., 2013).
Lorsque des virus zoonotiques (A) entrent en contact avec l’Homme (C) ils rencontrent la barrière d’espèce qui est composée de facteurs cellulaires qui interfèrent avec le cycle viral : les facteurs de restriction. Les virus se répliquent dans le nouvel hôte de manière plus ou moins efficace (D). Des mutations chez les virus se répliquant inefficacement peuvent conduire à une adaptation de ces virus au nouvel hôte (E). Ces virus sont alors dits émergents (F) chez l’Homme. Parmi ces virus certains se transmettront efficacement dans la population humaine (G) et entreront dans le réservoir humain (A'), alors que d’autres, dont l’adaptation est sub-optimale, ne se transmettront pas entre Hommes. Que ce soit dans le réservoir zoonotique ou humain la réplication virale conduit à l’apparition de mutations et donc de nouveaux variants génétiques (B) qui seront sélectionnés par leur capacité à se répliquer dans l’hôte. La diversité génétique des virus est représentée par les différentes couleurs. Les pictogrammes numérotés de 1 à 5 font référence aux différents stades de l’émergence d’un virus humain décrits dans la figure 1.
Les virus émergents peuvent aussi être non-zoonotiques. C’est le cas du poliovirus, qui aurait émergé à partir de coxsackievirus de type A infectant l’Homme, par accumulation de mutations (Figure 2B) et un changement dans le tropisme cellulaire (capacité à infecter un type cellulaire) (Jiang et al., 2007). En effet, alors que les coxsackievirus de type A infectent la peau et les muqueuses, les poliovirus peuvent infecter le système nerveux central notamment les motoneurones et la moelle épinière.
L’identification des réservoirs viraux permet de mieux appréhender les modes de transmission des virus à l’Homme et ainsi de pouvoir mettre en place des mesures prophylactiques (préventives) pour éviter tout risques épidémiques ou stopper une épidémie préexistante. Dans le cas du SRAS, dont l’épidémie a touché majoritairement les pays d’Asie du Sud-Est, l’identification de la civette palmiste à masque (Paguma larvata) comme hôte intermédiaire a permis de stopper l’épidémie par la fermeture des marchés animaliers (Flanagan et al., 2012).
Le passage d’un virus à un nouvel hôte reste cependant un événement rare (Figure 2G), il existe des barrières d’espèces qui limitent l’infection d’un nouvel hôte par un virus.
Barrières d’espèces et adaptations au nouvel hôte
Lors de son introduction dans un nouvel hôte, le virus est confronté à des barrières d’espèces qui sont constituées par des différences entre l’hôte initial et le nouvel hôte. Les molécules de l’hôte qui interfèrent avec le cycle viral sont appelées facteurs de restriction. Par exemple, la tétherine, est une molécule présente dans la membrane des cellules de mammifères qui participe à la barrière d’espèce entre le chimpanzé et l’Homme pour le VIS (virus de l’immunodéficience simienne, dont dérive le VIH) car elle diffère au niveau de son domaine transmembranaire dans ces deux espèces. Au cours des différents sauts d’espèces qui ont marqué l’émergence des 3 groupes de VIH-1 (M, N et O), ces virus se sont adaptés de façon différente à la tétherine pour chacun des 3 passages du chimpanzé à l’Homme. La protéine Vpu du VIH-1 M a ainsi acquis une activité anti-tétherine humaine optimale, qui pourrait expliquer son succès évolutif et son caractère pandémique, tandis que les autres groupes de VIH-1, N et O qui présentent respectivement une adaptation sub-optimale ou absente à l’inhibition de la tétherine humaine, ne sont pas pandémiques (Sauter et al., 2009).
Cependant la transmission inter-espèce de certains virus ne semble pas nécessiter d’adaptation (Figure 2F). Ainsi, les virus de l’hépatite E du porc et de l’Homme sont capables d’infections croisées et sont génétiquement très proches (Goens & Perdue, 2004).
Après qu’un virus a franchi la barrière d’espèce, il peut acquérir des propriétés qui lui permettent de se maintenir dans son nouvel hôte, tout en échappant aux pressions sélectives naturelles ou artificielles (anticorps neutralisants, molécules antivirales). Il s’agit de modifications génétiques qui surviennent au cours des étapes majeures du cycle réplicatif du virus et qui sont fixées dans la population car favorables aux virus qui les possèdent (Domingo, 2010) (Figure 2E). Les virus influenza pandémiques, notamment H1N1 de 1918 (grippe espagnole) et de 2009 présentent des substitutions d’acides aminés au niveau de la séquence de leurs glycoprotéines de surface (hémagglutinine HA et neuraminidase NA), par rapport aux virus aviaires dont ils sont issus. Ces mutations sont à l’origine d’un équilibre fonctionnel entre les activités de liaison et de libération des deux protéines du virus, qui pourrait participer à son adaptation à l’hôte humain (Xu et al., 2012). D’autres virus, comme le VIH ou le virus de l’hépatite B, sont connus pour avoir présenté une augmentation de leur valeur sélective (fitness) via l’acquisition d’une résistance antivirale en trois étapes : la sélection des variants de la quasi-espèce les moins sensibles aux drogues testées ; l’acquisition de mutations additionnelles augmentant la résistance souvent tributaires d’une diminution de la valeur sélective, en l’absence de médicaments ; et enfin l’accumulation de mutations compensatoires, rétablissant une capacité réplicative élevée chez certains variants résistants (Nijhuis et al., 2009).
Espèce et quasi-espèce virale
La notion d’espèce virale est assez récente puisqu’elle n’a qu’une vingtaine d’années. Elle a été introduite par l’ICTV International Committee on Taxonomy of Viruses, pour deux raisons : tenter de rapprocher la classification des virus des autres classifications (les notions de famille et genre existaient déjà, mais pas la notion d’espèce) et aussi essayer de tenir compte des avancées scientifiques et de la place de plus en plus importante de la génomique (beaucoup de genres sont définis sur des critères sérologiques).
Une espèce virale est définie comme une classe polythétique composée de virus qui constituent un lignage réplicatif et qui occupent une niche particulière. Une classe polythétique est constituée de membres ayant des propriétés communes mais qui ne les partagent pas toutes entre eux. La définition d’une espèce virale peut être différente d’une famille à l’autre. En revanche pour les taxons d’ordres supérieurs, comme les familles, les critères d’appartenance définis par l’ICTV sont stricts et partagés par tous les membres.
La notion de quasi-espèce virale n’a, quant à elle, pas de référence taxonomique. Elle exprime le fait que, dans un échantillon donné, un virus est en fait un groupe de variants génétiques, c’est-à-dire une population de virions qui sont très proches, mais dont les séquences présentent tout de même des différences de quelques nucléotides.
Il est intéressant de noter que les virus sont des entités physiques réelles alors que les familles ou espèce virales sont des concepts abstraits produits par la logique. Les liens entre virus et espèces représentent donc la frontière entre biologie et logique !
Texte de l'encadré par Cyril Barbezange, Scientific Institute of Public Health, Bruxelles.
Variabilité et émergence
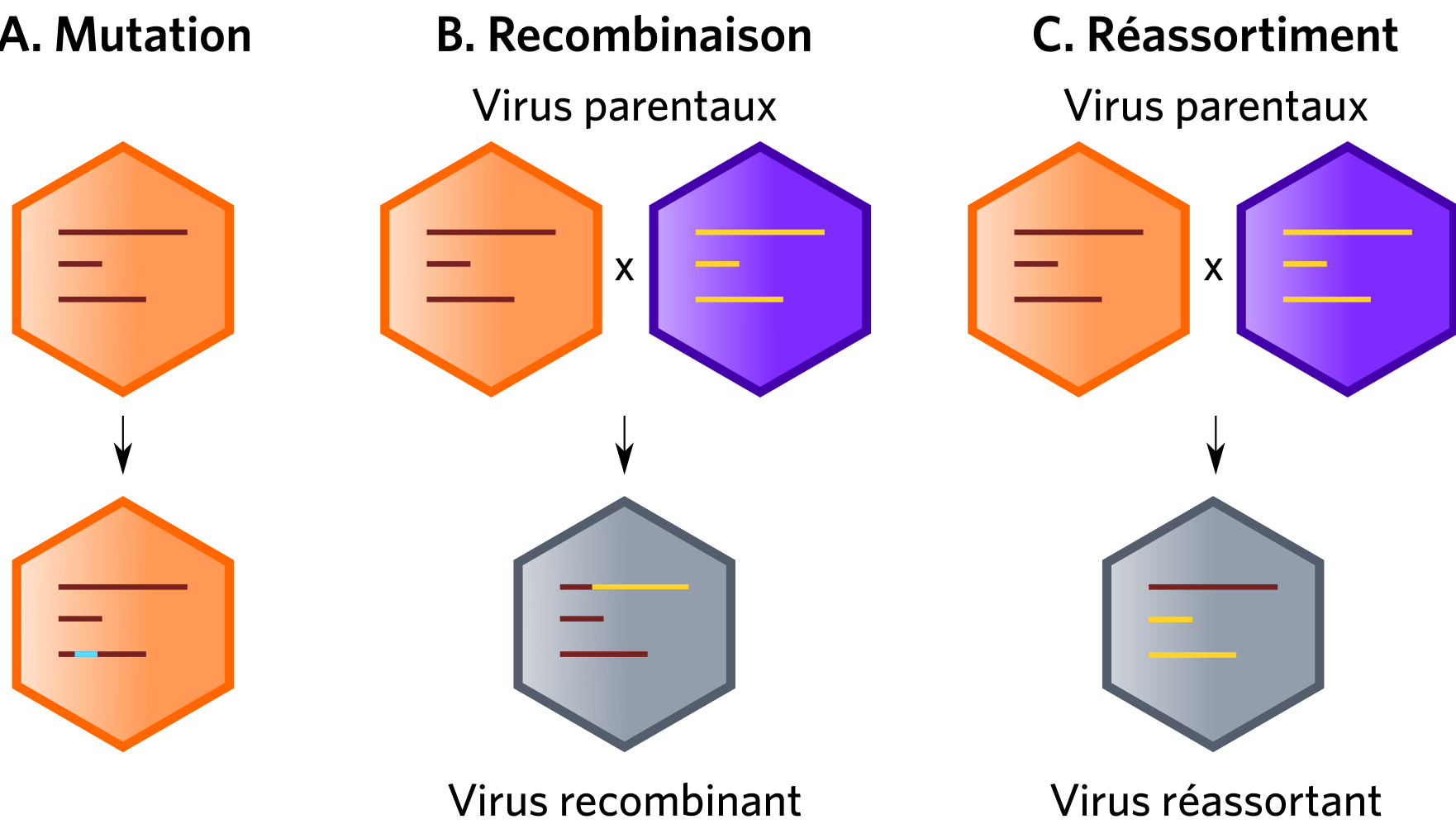
Les quasi-espèces virales forment un réservoir dynamique de variants génétiques (Figure 2B) jouant un rôle à toutes les étapes de l’émergence. Cette variabilité génétique découle de l’action cumulée des mutations, recombinaisons ou réassortiments des génomes viraux (Figure 3).
Ces mutations se produisant en permanence, elles maintiennent des sous populations sélectionnables lors de l’introduction d’un virus dans un nouvel hôte (Domingo, 2010). Elles permettent par exemple de faire face aux pressions de sélections, qu’elles soient naturelles ou artificielles comme l’adaptation du VIH-1 aux facteurs de restrictions (Sauter et al., 2010) ou aux antiviraux (Charpentier et al., 2004).
Les recombinaisons et réassortiments (Figure 3) sont quant à eux des moteurs puissants dans les émergences. À titre d’exemple, les poliovirus sauvages et vaccinaux sont capables de recombiner avec d’autres Entérovirus, formant ainsi de nouveaux poliovirus potentiellement virulents (Jegouic et al., 2009 ; Jiang et al., 2007). Ces recombinaisons sont limitées par la compatibilité entre les régions codant les protéines structurales et les régions codant les protéines non structurales des génomes parentaux. Seuls les recombinants ayant des gènes codant pour la capside originaire du poliovirus sont viables. Les recombinants polio-entérovirus possédant une capside d’entérovirus non-polio ne sont pas capables de se répliquer (Liu et al., 2010). Une recombinaison entre des gènes de poliovirus codant pour des protéines non-structurales avec des gènes de structure d’un coxsackie virus de type C ne permet pas l’encapsidation du génome et abouti à une particule non viable. La protéine 2Catpase codées par les régions non structurales régule l’encapsidation et possède une affinité spécifique pour la région structurale d’un même virus. Cela est à l’origine de l’incompatibilité de recombinaison entre certains entérovirus. Les poliovirus ont à l’origine émergés à partir des coxsackies virus. Cette spéciation s’est accompagnée d’un changement de récepteurs cellulaire et donc de tropisme en passant du récepteur ICAM-1 au récepteur CD-155. Il semble cependant que les régions structurales des poliovirus aient conservé une affinité pour la protéine 2Catpase des coxsackies virus actuels. (Liu et al., 2010) Ainsi, cela suggère que l’évolution des entérovirus de type C est engagée dans une voie de non-retour vers l’émergence de nouveaux poliovirus. Chaque année apparaissent ainsi des cas de poliomyélite chez l’Homme dû à ces recombinants. Il existe également de nombreux exemples décrits de réassortiment entre virus segmentés, particulièrement pour les virus influenza A ayant conduit à des pandémies chez l’Homme (Holmes et al., 2005) ou lors de co-infections de différents Rotavirus (Tran et al., 2013).
Variabilité génétique et émergence virale sont donc liées de façon inéluctable, bien qu’il existe d’autres facteurs favorisant les émergences virales humaines.
Facteurs favorisant l’émergence virale
L’émergence ou la ré-émergence d’un virus est un phénomène multifactoriel, favorisé par la perturbation d’un ou plusieurs facteurs au sein d’un écosystème.
Elle peut être issue d’un bouleversement des niches écologiques des animaux hôtes, entraînant un contact favorisé avec l’être humain (Figure 2C). La déforestation par exemple, augmente le risque de contact avec des animaux sauvages potentiellement infectés (Wolfe et al., 2005). Dans certains cas les contacts privilégiés sont aussi le fruit de comportements sociaux. Dans des régions du Cameroun, les hommes chassent et consomment des singes, dont certains sont infectés par le foamy virus. Ces pratiques culturelles augmentent le risque de transmission des singes à l’Homme (Subramanian, 2012), en augmentant notamment les risques de blessures profondes, portes d’entrées du foamy virus chez l’Homme (Betsem et al., 2011). Dans le cas de virus transmis par des insectes vecteurs, des changements dans leurs aires de répartition peuvent également favoriser l’émergence de virus dans la population humaine. Ces dernières décennies, la migration des moustiques Aedes aegypti et Aedes albopictus (le moustique tigre) vers les zones urbaines ont conduit à l’émergence du virus Zika et du virus Chikungunya chez les humains. En conséquence, l’augmentation de la fréquence des infections humaines a permis la sélection de variants génétiques plus efficaces de ces virus à l’origine des récentes épidémies (Weaver & Forrester, 2015). L’émergence virale est également favorisée par de nouveaux comportements à l’échelle mondiale. Du fait de la mondialisation, les flux de personnes se sont accrus, facilités et accélérés, permettant alors une meilleure dissémination des virus. Le MERS-CoV par exemple, endémique du Moyen-Orient, a été importé en Europe par des patients transportés en avion (Mailles et al., 2013).
Enfin, l’émergence virale peut être due à un défaut d’hygiène et de couverture vaccinale et médicale. On a constaté depuis le début du conflit en Syrie la ré-émergence de la poliomyélite infantile causée par une souche de poliovirus sauvage endémique du Pakistan (Arie, 2013).
Conclusion
L’émergence virale est un phénomène naturel, dynamique et perpétuel soutenu par la diversité génétique des virus. L’introduction, l’établissement et la dissémination de nouveaux virus est la conséquence d’une adaptation virale efficace.
D’importants évènements pandémiques ont mis en lumière le rôle clef des animaux dans les stades précoces de l’émergence virale chez l’Homme. Ce qui a conduit au concept One Health, qui suppose que si l’on contrôle le virus dans le réservoir animal, s’il existe et est connu, par effet ricochet l’hôte humain se trouve alors être protégé (Rubin et al., 2013). Ainsi la vaccination des renards en Europe de l’Ouest a permis d’éradiquer les cas de rage humaine dans les pays ayant mis en place un programme de vaccination efficace du réservoir sauvage (Freuling et al., 2013).
Cependant tous les virus émergents ne sont pas pandémiques. Certains ne passent pas la barrière d’espèce, restreints par des facteurs spécifiques du nouvel hôte, tandis que d’autres avec une valeur sélective faible induisent uniquement des infections humaines sporadiques comme certaines souches de virus influenza (Xu et al., 2012). Mieux comprendre les mécanismes d’émergences, les réservoirs et la dynamique des populations de virus, permettra une meilleure réponse aux futures émergences virales humaines.
Ce travail a été initié dans le cadre d’un projet de master 2, en coordination avec le Dr Marion Desdouits de l’Institut Curie.
Bibliographie
- Arie S. (2013). Polio outbreak leads to calls for a “vaccination ceasefire” in Syria. BMJ (Clinical research ed.), 347, 66.
- Betsem E., Rua R., Tortevoye P., Froment A. & Gessain, A. (2011). Frequent and recent human acquisition of simian foamy viruses through apes’ bites in central Africa. PLoS pathogens, 7(10), e1002306.
- Charpentier C., Dwyer D. E., Mammano F., Lecossier D. & Hance A. J. (2004). Role of Minority Populations of Human Immunodeficiency Virus Type 1 in the Evolution of Viral Resistance to Protease Inhibitors. Journal of Virology, 78(8), 4234–4247.
- Cotten M., Lam T. T., Watson S. J., Palser A. L., Petrova V., Grant, P. & Nastouli E. (2013). Full-Genome Deep Sequencing and Phylogenetic Analysis of Novel Human Betacoronavirus. Emerging infectious diseases, 19(5), 736–742.
- Domingo E. (2010). Mechanisms of viral emergence. Veterinary research, 41(6), 38.
- Flanagan M. L., Parrish C. R., Cobey S., Glass G. E., Bush R. M. & Leighton T. J. (2012). Anticipating the species jump : surveillance for emerging viral threats. Zoonoses and public health, 59 (3), 155–63.
- Freuling C. M., Hampson K., Selhorst T., Schröder R., Meslin F. X., Mettenleiter T. C. & Müller T. (2013). The elimination of fox rabies from Europe : determinants of success and lessons for the future. Philosophical transactions of the Royal Society of London. Series B, Biological sciences , 368(1623), 20120142.
- Goens S. D. & Perdue M. L. (2004). Hepatitis E viruses in humans and animals. Animal health research reviews / Conference of Research Workers in Animal Diseases , 5(2), 145–56.
- Holmes E. C., Ghedin E., Miller N., Taylor J., Bao Y., George K. S. & Taubenberger, J. K. (2005). Whole-Genome Analysis of Human Influenza A Virus Reveals Multiple Persistent Lineages and Reassortment among Recent H3N2 Viruses. PLOS Biology, 3(9), e300.
- Jegouic S., Joffret M., Blanchard C., Riquet F. B., Colbere-garapin F., Rakoto-andrianarivelo M. & Delpeyroux F. (2009). Recombination between Polioviruses and Co-Circulating Coxsackie A Viruses : Role in the Emergence of Pathogenic Vaccine-Derived Polioviruses. PLoS Pathogens, 5(5), e1000412.
- Jiang P., Faase J. A. J., Toyoda H., Paul A., Wimmer E. & Gorbalenya A. E. (2007). Evidence for emergence of diverse polioviruses from C-cluster coxsackie A viruses and implications for global poliovirus eradication. Proceedings of the National Academy of Sciences of the United States of America , 104(22), 9457–62.
- Jones K. E., Patel N. G., Levy M. A., Storeygard A., Balk D., Gittleman J. L. & Daszak P. (2008). Global trends in emerging infectious diseases. Nature, 451(7181), 990–3.
- Leroy E. M., Kumulungui B., Pourrut X., Rouquet P., Hassanin A., Yaba P. 1 Swanepoel, R. (2005). Fruit bats as reservoirs of Ebola virus. Nature, 438(7068), 575–6.
- Liu Y., Wang C., Mueller S., Paul A. V, Wimmer E. & Jiang, P. (2010). Direct Interaction between Two Viral Proteins, the Nonstructural Protein 2C ATPase and the Capsid Protein VP3, Is Required for Enterovirus Morphogenesis. PLoS Pathogens, 6(8), e1001066.
- Mailles A., Blanckaert K., Chaud P., Van Der Werf S., Lina B., Caro V. & Che D. (2013). First cases of Middle East Respiratory Syndrome Coronavirus (MERS-CoV) infections in France, investigations and implications for the prevention of human-to-human transmission, France, May 2013. European communicable disease bulletin, 18(24).
- Morse S. S., Mazet J. A. K., Woolhouse M., Parrish C. R., Carroll D., Karesh W. B., & Daszak P. (2012). Prediction and prevention of the next pandemic zoonosis. Lancet, 380, 1956–65.
- Nijhuis M., Van Maarseveen N. M. & Boucher C. A. B. (2009). Antiviral resistance and impact on viral replication capacity : evolution of viruses under antiviral pressure occurs in three phases. Handbook of experimental pharmacology, (189), 299–320.
- Pike B. L., Saylors K. E., Fair J. N., Lebreton M., Tamoufe U., Djoko C. F. & Wolfe N. D. (2010). The origin and prevention of pandemics. Clinical infectious diseases : an official publication of the Infectious Diseases Society of America , 50(12), 1636–40.
- Rubin C., Myers T., Stokes W., Dunham B., Harris S., Lautner B. & Annelli J. (2013). Review of Institute of Medicine and National Research Council Recommendations for One Health Initiative. Emerging Infectious Diseases, 19(12), 1913–1917.
- Sauter D., Schindler M., Specht A., Landford W. N., Kim K., Votteler J., & Hatziioannou T. (2010). The evolution of pandemic and non-pandemic HIV-1 strains has been driven by Tetherin antagonism. Cell Host Microbe, 6(5), 409–421.
- Sauter D., Schindler M., Specht A., Landford W. N., Mu J., Kim K., & Kirchhoff F. (2009). Article Tetherin-Driven Adaptation of Vpu and Nef Function and the Evolution of Pandemic and Nonpandemic HIV-1 Strains. Cell Host & Microbe, 6, 409–421.
- Subramanian M. (2012). Zoonotic disease risk and the bushmeat trade : assessing awareness among hunters and traders in Sierra Leone. EcoHealth, 9(4), 471–82.
- Tran T. N. H., Nakagomi T. & Nakagomi O. (2013). Evidence for Genetic Reassortment between Human Rotaviruses by Full Genome Sequencing of G3P [4] and G2P [4] Strains Co-circulating in India. Tropical Medicine and Health, 41(1), 13–20.
- Weaver S. C., Forrester N. L. (2015). Chikungunya : Evolutionary history and recent epidemic spread. Antiviral Research, 120, 32-39.
- Wolfe N. D., Daszak P., Kilpatrick A. M. & Burke D. S. (2005). Bushmeat hunting, deforestation, and prediction of zoonoses emergence. Emerging infectious diseases, 11(12), 1822–7.
- Xu R., Zhu X., Mcbride R., Nycholat C. M., Yu W., Paulson J. C. & Wilson I. A. (2012). Functional Balance of the Hemagglutinin and Neuraminidase Activities Accompanies the Emergence of the 2009 H1N1 Influenza. Journal of Virology, 86(17), 9221–9232.