Certains chevaux, comme les Camarguais, naissent avec une robe sombre qui s'éclaircit avec l'âge. La détermination du locus impliqué dans ce changement de coloration a permis de proposer un mécanisme explicatif au grisonnement de ces chevaux.
Il est bien connu que Crin-Blanc, cheval de race camarguaise du film éponyme (A. Lamorisse, 1953), était en fait… gris très clair ! Alors que les poulains camarguais naissent noirs ou bruns sombres, leur robe éclaircit avec l’âge jusqu’à devenir blanche (Figure 1).
D’autres races de chevaux arborent le même phénotype (les lipizzans, par exemple) et, d’une manière générale, cet éclaircissement de la robe est observé chez tous les chevaux gris. L’origine de la couleur grise serait due à une unique mutation, apparue il y a un peu plus de mille ans [6]. Elle a été vite sélectionnée dans les races hispaniques et notamment pour les chevaux de rois. Il semble peu probable que l’environnement dans lequel vivent ces chevaux soit la cause de leur couleur de robe, vu qu’il s’en trouve partout dans le monde. Cependant la couleur grise est plus fréquente chez les chevaux orientaux (Pur sang Arabe et dérivés) et hispaniques : peut-être est-elle donc une adaptation aux climats chauds [1] ? Cette question demeure en suspens. Quoi qu’il en soit, il existerait un allèle responsable de la couleur grise, présent chez tous les chevaux gris, et notamment les Camarguais chez qui il a été sélectionné par les Hommes. Les chevaux non gris sont en effet interdits dans le stud-book de la race du cheval de Camargue [1].
Existence d’un locus « gris »
Le croisement d’un cheval gris homozygote pour le gène responsable de cette couleur de robe (noté G) avec un cheval non-gris (homozygote pour l’autre allèle du gène G, noté g) donne des descendants hétérozygotes G//g. Ces descendants sont tous gris, ce qui signifie que l’allèle G est dominant et que g est récessif. La dominance de l’allèle G a été établie au début du XXe siècle, par les premiers généticiens s’étant intéressés à ce caractère [3].
L’existence d’un locus responsable du phénotype « robe grise » chez les chevaux a donc été établie très tôt. Mais la localisation précise de l’allèle à l’origine de cette coloration restait inconnue.
Étant donné que les chevaux possèdent trente-deux paires de chromosomes, la première étape est d’identifier le chromosome porteur du locus G. Il s’agit ensuite de réduire la taille de la région où se trouve ce locus, étant donné qu’un chromosome porte en moyenne entre cinq cents et mille gènes, afin d’identifier le gène correspondant au locus G.
Localisation du locus G
La méthode historiquement utilisée pour situer un locus consiste à chercher les déséquilibres de liaison. Deux loci sont liés génétiquement s’ils sont transmis ensemble à la descendance, autrement dit s’ils ne sont pas séparés par le brassage intrachromosomique. La distance génétique entre deux allèles est définie comme le pourcentage de recombinaison entre ces allèles et exprimée en centiMorgan (1 cM = 1 % de recombinaison).
Les méthodes de localisation d’un locus
D’autres méthodes plus modernes sont mises en œuvre aujourd’hui, reposant sur les nouvelles techniques de séquençage à haut débit (NGS, Next-Generation Sequencing). Les marqueurs les plus fréquemment génotypés sont les SNP (polymorphisme mononucléotidique) et non plus les microsatellites. Des études à grande échelle (GWAS, Genome-Wide Association Study) sont désormais privilégiées, portant sur un très grand nombre d’individus (plusieurs milliers), dont les variations génétiques de l’ensemble du génome sont évaluées (plusieurs centaines de milliers de SNP). Lire par exemple à ce sujet Du génotype au phénotype : les motifs des coccinelles arlequins.
Mesurer le déséquilibre de liaison avec des marqueurs placés tout le long du génome permet de déterminer l’emplacement d’un locus. Des marqueurs couramment utilisés sont les séquences microsatellites. Ce sont des séquences d’ADN non codantes constituées d’un motif d’un à quatre nucléotides, répété cinq à cent fois, et très polymorphiques. La séquence du motif et le nombre de répétitions à une localisation donnée varient selon les individus. Le locus G est lié génétiquement à des marqueurs présents chez tous les chevaux gris, mais pas chez les autres chevaux.
Plusieurs études, menées dans des familles de chevaux gris appartenant à différentes races, ont montré que le locus G est situé sur le chromosome 25 (ECA25 pour Equus caballus 25).
Par exemple, l’étude menée par Swinburne et ses collaborateurs en 2002 est basée sur la descendance d’un étalon gris, Paris House [4]. Ayant un père gris et une mère blanche, et étant lui-même gris, Paris House est forcément de génotype G//g. Le croisement de Paris House avec des femelles non grises (donc g//g), a donné une descendance de 42 individus constitués, comme l'on pouvait s'y attendre, d'environ 50 % (19/42) d’individus gris et de 50 % (23/42) de non gris.
Parmi tous les marqueurs présents sur le génome, seuls ceux qui sont à l’état hétérozygote chez Paris House sont informatifs. En effet, si un marqueur est présent à l’état homozygote, son association au locus gris ne peut pas être détectée, puisque les deux allèles G et g sont dans ce cas associés au même variant du marqueur.
Pour savoir si un marqueur particulier est associé au locus G, on calcule un score LOD : plus celui-ci est élevé, plus la liaison du marqueur au trait est forte. Le score LOD (logarithm of odds) est une valeur statistique qui compare, pour deux modèles théoriques donnés, la probabilité que chaque modèle aboutisse aux données réelles. Dans le cas du locus G, le score LOD est calculé pour chaque marqueur i selon la formule :
$$LOD_{i} = \log \frac{L(G \text{ associé au marqueur } i)}{L(G \text{ non associé au marqueur } i)}$$
La fonction L est appelée vraisemblance du modèle, il s’agit de la probabilité d’observation des données (la distance génétique entre G et le marqueur i) en supposant que G est associé au marqueur i (premier modèle, au numérateur) ou non (deuxième modèle, au dénominateur).
Si G est effectivement associé au marqueur, le rapport des vraisemblances est très grand, et donc le score LOD l’est également. Si au contraire, G n’est pas associé au marqueur, le score LOD est très petit.
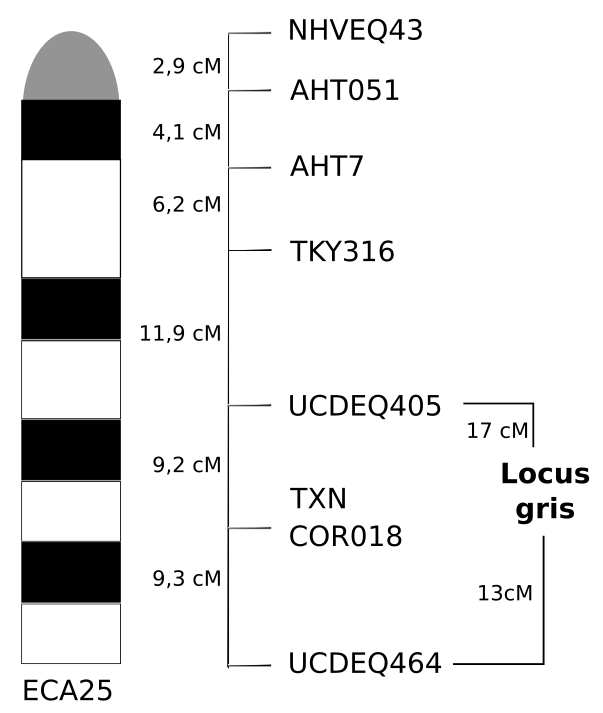
Le score LOD a été calculé pour différents marqueurs situés sur le chromosome ECA25 (Figure 2).
Le score est calculé pour différentes distances entre le marqueur et le locus G (première ligne). Les loci non informatifs ne sont pas considérés [4].
L’examen du tableau montre que les scores LOD les plus importants sont obtenus pour une distance de 17 cM entre le locus G et le marqueur TKY316 et pour une distance de 13 cM entre ce locus et le marqueur UCDEQ464 (Figure 3).
Une autre étude menée sur des chevaux Quarter-Horse la même année précise ce résultat [5], et place le locus gris au niveau du marqueur COR080. Une troisième étude portant sur deux familles de chevaux Franches-Montagnes aboutit au même résultat [1].
Recherche du gène causal du phénotype gris
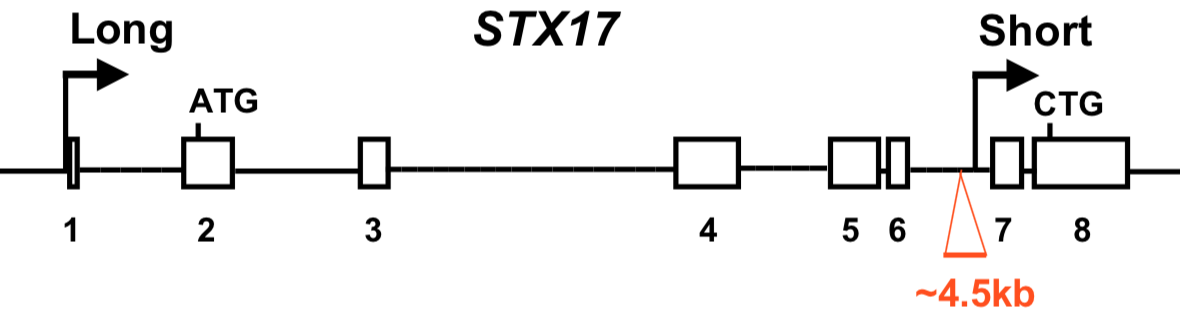
Six ans après la localisation du locus G sur le chromosome 25, la mutation à l’origine du phénotype gris a été identifiée [6]. Quatre gènes sont présents dans cette région. Cependant, aucun polymorphisme de séquence des exons de ces gènes n’est retrouvé uniquement chez les chevaux gris. La seule différence est identifiée dans un intron du gène STX17, qui possède une insertion de 4,5 kb, soit 4 500 nucléotide (Figure 4). Il s’agit d’une duplication en tandem présente au sein de l’intron.
Une deuxième étude par séquençage précis de la région du locus gris (325 kb) a confirmé que seule cette duplication en tandem est associée au locus gris [7].
Les rectangles numérotés représentent les exons. Les introns correspondent aux traits entre les exons. L’insertion présente chez les chevaux gris est en rouge.
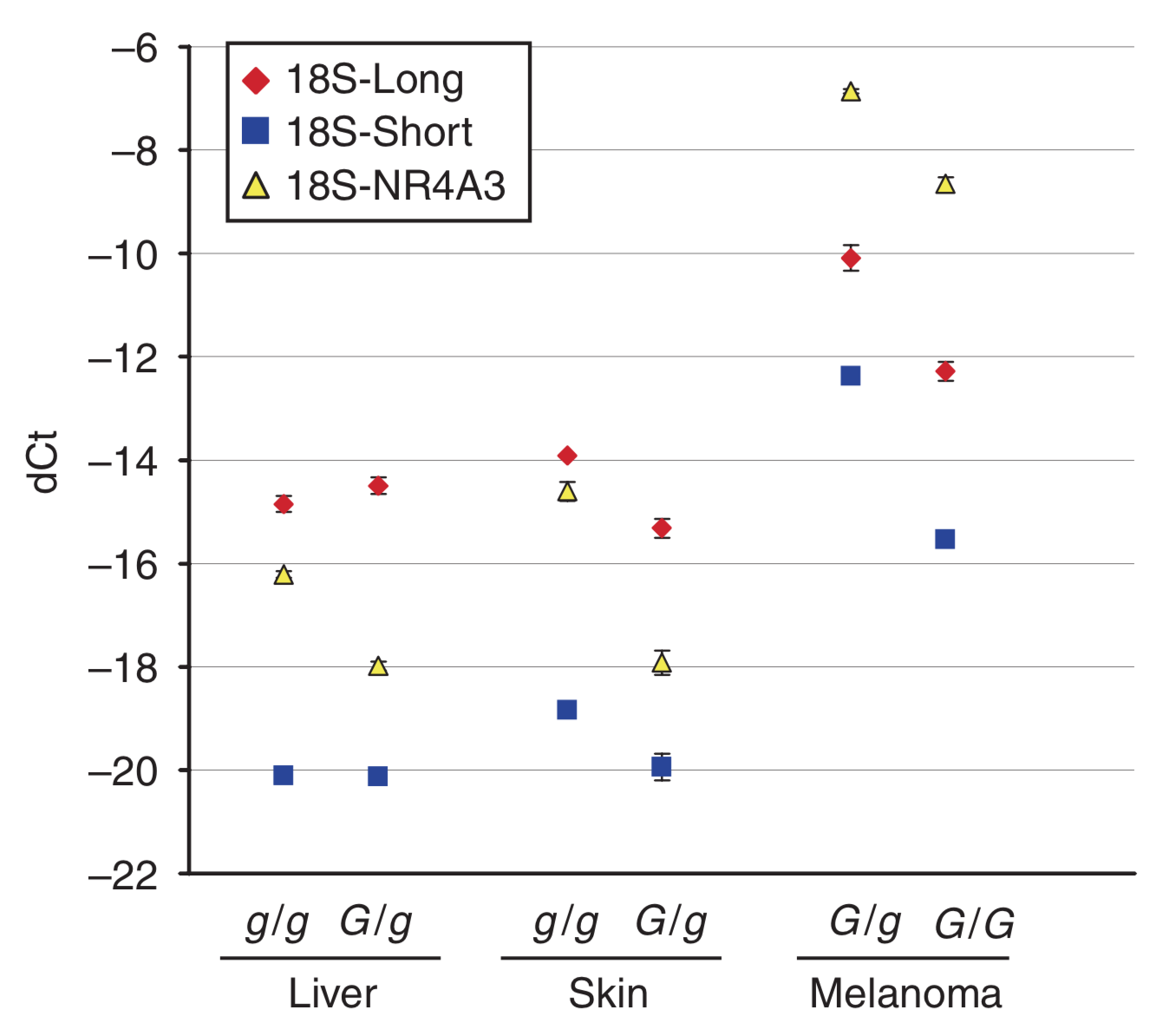
Pour le gène STX17, on mesure la quantité de transcrits longs (Long) et courts (Short) ; voir Figure 4. Le niveau d’expression de référence est celui de l’ARN 18S. Ct représente le nombre de cycles permettant d’atteindre une quantité seuil d’ADN produit, et dCt la différence entre cette valeur pour le transcrit d’intérêt et pour la référence. Plus dCt est faible, moins le transcrit est exprimé.
Cette mutation peut-elle avoir des effets sur l’expression des gènes localisés à proximité ? Pour le savoir, une équipe de chercheurs a étudié le niveau d’expression de deux de ces gènes, STX17 et NR4A3, dans différents tissus (Figure 5). Les résultats montrent que si le niveau d’expression est relativement similaire dans le foie et la peau, STX17 et NR4A3 sont surexprimés dans les cellules de mélanome (tumeur provenant d’une prolifération des mélanocytes). Ces deux gènes étant des activateurs du cycle cellulaire, ils pourraient être à l’origine du développement de ces mélanomes, plus fréquemment observés chez les chevaux gris.
Mécanisme explicatif hypothétique
Comment, à la lumière de ces différentes données, expliquer le changement de coloration progressif des chevaux à robe grise ? En stimulant la prolifération des mélanocytes et la synthèse de mélanine, STX17 et NR4A3 causeraient un assombrissement de la robe chez les jeunes chevaux. Mais cette hyper-prolifération épuiserait très vite le stock de cellules souches à l’origine des mélanocytes. En vieillissant, les poils seraient donc dépourvus de pigments, ce qui expliquerait l’éclaircissement progressif de la robe. De plus, l’accroissement de synthèse de mélanine par les mélanocytes et la prolifération accrue de ceux-ci pourraient causer le développement de mélanomes, ou cancers de la peau, plus fréquents en moyenne chez les chevaux gris que chez les autres chevaux (70 à 80 % des chevaux gris développent un mélanome en vieillissant). Ce mécanisme reste hypothétique et n’a pas été démontré in vivo, mais il est convaincant au vu de l’étude génétique.
Conclusion
Les chevaux gris sont un modèle non conventionnel d’étude du développement de mélanomes. Ils sont toutefois intéressants car les gènes majoritairement mis en cause ne sont pas les mêmes que chez d’autres modèles (la souris par exemple). La confrontation de différents modèles permet d’appréhender les différents aspects et les différentes sources de développement des mélanomes [9]. Les chevaux gris peuvent même être utilisés pour tester de nouveaux médicaments [10].
La cause de l’éclaircissement des chevaux gris est génétique. Elle est associée à un risque élevé de développer un mélanome, donc désavantageuse. Elle existe pourtant encore aujourd’hui, et domine dans certaines populations. Monter un cheval blanc était prestigieux, et l’apanage des rois et des empereurs. Les chefs, sur les tableaux, sont représentés sur des chevaux blancs (Figure 6) alors que les personnages de rang inférieur chevauchent des bais ou des alezans. Cet exemple illustre à quel point la domestication modifie le fitness des individus. Aucun caractère n’est avantageux dans l’absolu. Sa valeur sélective dépend de l’environnement et peut changer au cours du temps.
Bibliographie
- Henner, J. et al. Genetic mapping of the (G)-locus, responsible for the coat color phenotype ‘progressive greying with age’ in horses (Equus caballus). Mamm. Genome 13, 535–537 (2002).
- Mendel, G. Experiments in plant hybridization (1865). 41
- A.T. Bowling - The Genetics of the Horse - 2000 | Equus (Genus) | Zebra. Scribd Available at: https://www.scribd.com/document/218852255/A-T-Bowling-The-Genetics-of-the-Horse-2000. (Accessed: 5th May 2018)
- Swinburne, J. E., Hopkins, A. & Binns, M. M. Assignment of the horse grey coat colour gene to ECA25 using whole genome scanning. Anim. Genet. 33, 338–342 (2002).
- Locke, M. M., Penedo, M. C. T., Bricker, S. J., Millon, L. V. & Murray, J. D. Linkage of the grey coat colour locus to microsatellites on horse chromosome 25. Anim. Genet. 33, 329–337 (2002).
- Rosengren Pielberg, G. et al. A cis-acting regulatory mutation causes premature hair graying and susceptibility to melanoma in the horse. Nat. Genet. 40, 1004–1009 (2008).
- Sundström, E. et al. Copy number expansion of the STX17 duplication in melanoma tissue from Grey horses. BMC Genomics 13, 365 (2012).
- Curik, I. et al. Complex Inheritance of Melanoma and Pigmentation of Coat and Skin in Grey Horses. PLoS Genet. 9, e1003248 (2013).
- van der Weyden, L. et al. Cross-species models of human melanoma. J. Patholo. 238, 152-165 (2016)
- Liebscher, G. et al. In vitro anticancer activity of Betulinic acid and derivatives thereof on equine melanoma cell lines from grey horses and in vivo safety assessment of the compound NVX-207 in two horses. Chem. Biol. Interact. 246, 20-29 (2016)